Molecular Epidemiological Investigation of HPV 56 in Precancerous Cervical Cancer Patients in Yunnan Province
-
摘要:
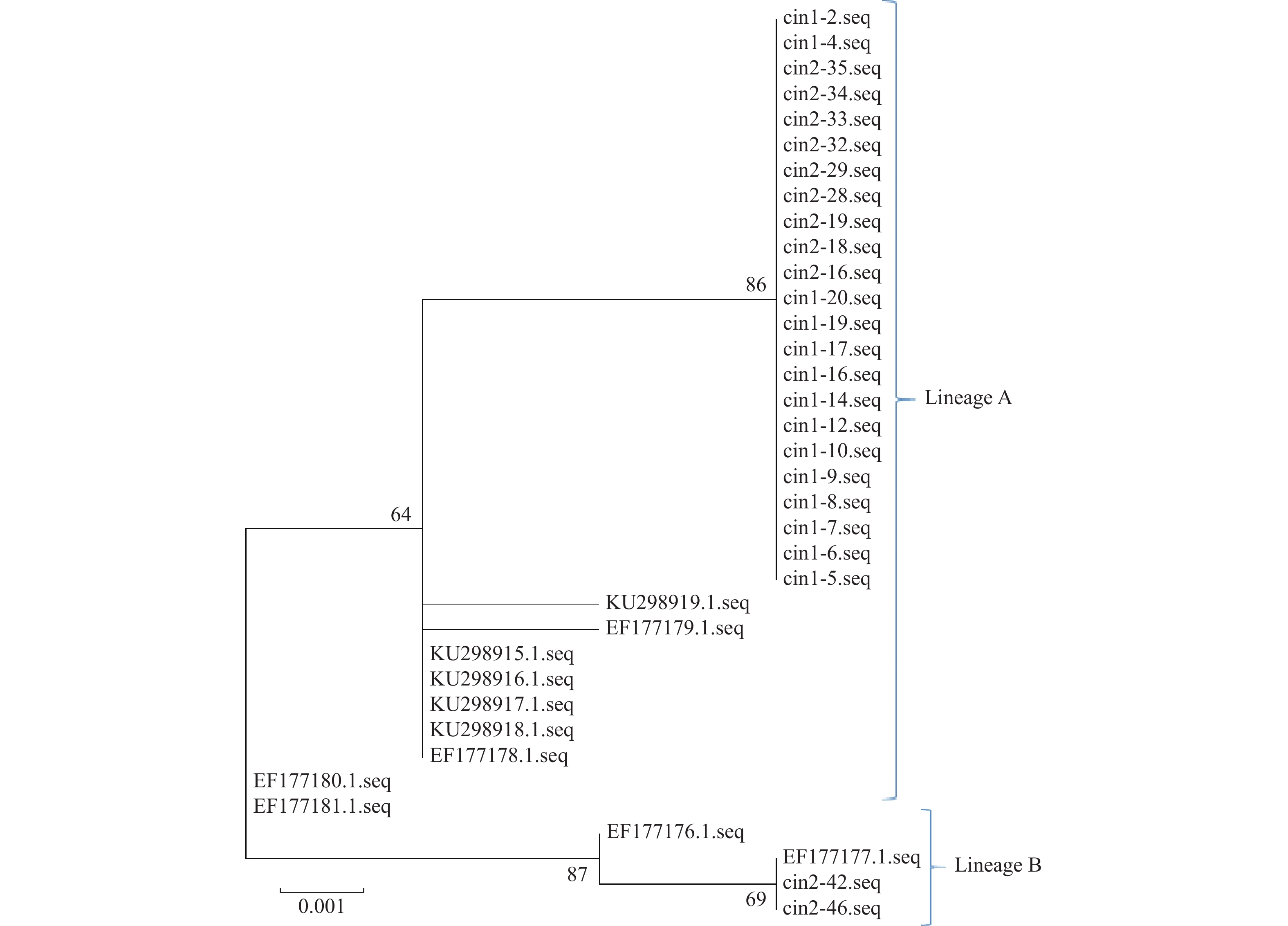
目的 研究单一感染HPV56型的癌前病变患者中HPV56 E6、E7和L1基因变异特征,获取云南地区宫颈癌癌前病变患者中HPV56型分子流行病学数据。 方法 随机选取300例宫颈癌癌前病变患者,通过HPV分型试剂盒对患者感染HPV的型别进行检测,并对HPV56型感染患者中HPV E6 E7和L1基因进行测序,最后完成对研究对象所感染的HPV56的谱系分布及HPV56 E6、E7和L1基因的遗传变异进行分析。 结果 研究对象所感染的HPV56病毒主要为A谱系,90%以上为A谱系,约10%为B谱系,HPV56 E6、E7、L1基因中共存在13个变异位点,其中E6基因中6个、E7基因中4个、L1基因中3个。HPV56 E6基因的6个变异位点中有2个为同义突变(T158C(S19S)、G437A(P112P)),4个为非同义突变(A141C(S14R)、A241G(N47S)、A263C(K54N)、G279A(D60N))。HPV56 E7基因的4个变异位点中均为非同义突变(C601A(D10E)、G605A(V12I)、G770C(E67Q)、G802C(Q77H))。HPV56 L1基因的3个变异位点中有2个为同义突变(G6454T(P321P)、A6868C(I459I)),1个为非同义突变(A5867G(N126D))。 结论 初步获得了云南地区宫颈癌癌前病变患者中HPV56 E6、E7和L1基因变异情况,获得了HPV56 E6、E7和L1基因的分子流行学特征,为进一步分析云南地区宫颈癌癌前病变患者中HPV56 E6、E7和L1基因的分子流行病学特征提供了必要的数据。 Abstract:Objective To study the genetic variation characteristics of HPV56 E6, E7 and L1 in patients with precancerous lesions infected with HPV56, and to obtain the molecular epidemiological data of HPV56 in patients with precancerous lesions of cervical cancer in Yunnan. Methods A total of 300 cervical intraepithelial neoplasia patients were enrolled and HPV genotyping kit was used to detect the types of HPV infection in the patients. The HPV E6, E7, and L1 genes in patients infected with HPV 56 were sequenced. Finally, the lineage distribution of HPV 56 and the genetic variations of HPV 56 E6, E7, and L1 genes in the study subjects were analyzed. Results In this study, the HPV56 virus infected by the study subjects mainly belonged to the A lineage, with more than 90% belonged to the A lineage, and about 10% belonged to the B lineage. There were 13 mutation loci in the E6, E7 and L1 genes of HPV56, including 6 in the E6 gene, 4 in the E7 gene and 3 in the L1 gene. Among the 6 mutation sites of HPV56 E6 gene, 2 were synonymous mutations (T158C (S19S), G437A (P112P)), and 4 were non-synonymous mutations (A141C (S14R), A241G (N47S), A263C (K54N), G279A (D60N)). The four mutation sites of HPV56 E7 gene were all non-synonymous mutations (C601A (D10E), G605A (V12I), G770C (E67Q), G802C (Q77H)). Among the three mutation sites of HPV56 L1 gene, two were synonymous mutations (G6454T (P321P), A6868C (I459I)), and one was non-synonymous mutation (A5867G (N126D)). Conclusion This study obtained preliminary data on the genetic variations of HPV 56 E6, E7, and L1 genes in CIN patients in Yunnan area, and obtained the molecular epidemiological characteristics of HPV 56 E6, E7, and L1 genes. It provides necessary data for further analysis of the molecular epidemiological characteristics of HPV 56 E6, E7, and L1 genes in CIN patients in Yunnan area. -
Key words:
- Cervical intraepithelial neoplasia /
- HPV56 /
- Variation /
- Lineage
-
表 1 HPV56 E6、E7、L1基因的扩增引物和测序引物
Table 1. The amplification primers and sequencing primers of E6 E7 and L1 genes of HPV56
基因 方向 序列5′-3′ 退火温度℃ E6 Forward ATTGGGAGTGACCGAAAAGG 63 Reverse ACAACACGCAGGTCCTCTTT E7 Forward GCTACAGATGTCAAAGTCCG 61 Reverse GCCTCTACTTCAAACCATCC L1-1 Forward GCCCCTTTAGGTAATGTGTGG 63 Reverse CCACATAGAATCACCATAGGCAT L1-1 Forward ATGATAGACACAGGATTTGGCG 63 Reverse ATACAACACACAAACACAGTTACAC 表 2 HPV56 E6、E7、L1基因变异情况
Table 2. E6,E7 and L1 gene variation of HPV56
核苷酸位置 E6 E7 L1 141 158 241 263 279 437 601 605 770 802 5867 6454 6868 X74483.1-E6 A T A A G G C G G G A G A cin1-2 − C − − A A A − C C G T C cin1-4 − C − − A A A − C − G T C cin1-5 − C − − A A A − C − 0 0 0 cin1-6 − C − − A A A − C − 0 0 0 cin1-7 − C − − A A A − C − G T C cin1-8 − C − − A A A − C − G T C cin1-9 − C − − A A A − C − G T C cin1-10 − C − − A A A − C − G T C cin1-12 − C − − A A A − C − G T C cin1-14 − C − − A A A − C − G T C cin1-16 − C − − A A A − C − G T C cin1-17 − C − − A A 0 0 0 0 G T C cin1-19 − C − − A A 0 0 0 0 0 0 0 cin1-20 − C − − A A 0 0 0 0 G T C cin1-21 0 0 0 0 0 0 0 0 0 0 G T C cin1-22 0 0 0 0 0 0 0 0 0 0 G T C cin2-2 0 0 0 0 0 0 A − C − 0 0 0 cin2-5 0 0 0 0 0 0 A − C − 0 0 0 cin2-12 0 0 0 0 0 0 A − C − 0 0 0 cin2-16 − C − − A A A − C − G T C cin2-18 − C − − A A A − C − 0 0 0 cin2-19 − C − − A A A − C − G T C cin2-22 0 0 0 0 0 0 A − C − 0 0 0 cin2-28 − C − − A A 0 0 0 0 G T C cin2-29 − C − − A A 0 0 0 0 0 0 0 cin2-32 − C − − A A 0 0 0 0 G T C cin2-33 − C − − A A A − C − 0 0 0 cin2-34 − C − − A A 0 0 0 0 0 0 0 cin2-35 − C − − A A A − C − G T C cin2-37 0 0 0 0 0 0 A − C − G T C cin2-42 C − G C − − − A − C 0 0 0 cin2-46 C − G C − − − A − C 0 0 0 氨基酸变化 S14R − N47S K54N D60N − D10E V12I E67Q Q77H N126D − − 标记为“0”的部分表示未扩增/测出基因序列; 标记为“-”的部分表示未检测到变异。 -
[1] Arbyn M,Weiderpass E,Bruni L,et al. Estimates of incidence and mortality of cervical cancer in 2018: A worldwide analysis[J]. Lancet Glob Health,2020,8(2):e191-e203. doi: 10.1016/S2214-109X(19)30482-6 [2] 李双,李明珠,丛青,等. 人乳头瘤病毒疫苗临床应用中国专家共识[J]. 中国妇产科临床杂志,2021,22(2):225-234. doi: 10.13390/j.issn.1672-1861.2021.02.045 [3] Burd E M. Human papillomavirus and cervical cancer[J]. Clin Microbiol Rev.,2003,16(1):1-17. doi: 10.1128/CMR.16.1.1-17.2003 [4] Lorincz A T, Quinn A P, Goldsborough M D, et al. Human papillomavirus type 56: A new virus detected in cervical cancers [J]. J Gen Virol, 1989, 70 ( Pt 11): 3099-3104. [5] Yang L,Yang H,Wu K,et al. Prevalence of HPV and variation of HPV 16/HPV 18 E6/E7 genes in cervical cancer in women in South West China[J]. J Med Virol,2014,86(11):1926-1936. doi: 10.1002/jmv.24043 [6] Zhao S,Zhao X,Hu S,et al. Distribution of high-risk human papillomavirus genotype prevalence and attribution to cervical precancerous lesions in rural North China[J]. Chin J Cancer Res,2019,31(4):663-672. doi: 10.21147/j.issn.1000-9604.2019.04.10 [7] Harari A,Chen Z,Burk R D. Human papillomavirus genomics: Past,present and future[J]. Curr Probl Dermatol,2014,45:1-18. [8] Zhou Z,Yang H,Yang L,et al. Human papillomavirus type 16 E6 and E7 gene variations associated with cervical cancer in a Han Chinese population[J]. Infect Genet Evol,2019,73:13-20. doi: 10.1016/j.meegid.2019.04.008 [9] Wu Y, Chen Y, Li L, et al. Analysis of mutations in the E6/E7 oncogenes and L1 gene of human papillomavirus 16 cervical cancer isolates from China [J]. J Gen Virol, 2006, 87(Pt 5): 1181-1188. [10] Lee C W,Bae J H,Lee S J,et al. Distribution of human papillomavirus type 16 E6 and E7 gene variants in the progression of cervical dysplasia in Korean women[J]. J Obstet Gynaecol Res,2011,37(10):1320-1326. doi: 10.1111/j.1447-0756.2010.01520.x [11] Sun Z,Ren G,Cui X,et al. Genetic diversity of HPV-16 E6,E7,and L1 genes in women with cervical lesions in Liaoning Province,China[J]. Int J Gynecol Cancer,2011,21(3):551-558. doi: 10.1097/IGC.0b013e3182112023 [12] Buck C B,Day P M,Trus B L. The papillomavirus major capsid protein L1[J]. Virology,2013,445(1-2):169-174. doi: 10.1016/j.virol.2013.05.038 [13] Jing Y,Wang T,Chen Z,et al. Phylogeny and polymorphism in the long control regions E6,E7,and L1 of HPV Type 56 in women from southwest China[J]. Mol Med Rep,2018,17(5):7131-7141. [14] Burk R D,Harari A,Chen Z. Human papillomavirus genome variants[J]. Virology,2013,445(1-2):232-243. doi: 10.1016/j.virol.2013.07.018 [15] 中华医学会. 临床诊疗指南. 妇产科学分册[M]. 北京: 人民卫生出版社, 2007: 69-73. [16] Munoz N,Bosch FX,de Sanjose S,et al. Epidemiologic classification of human papillomavirus types associated with cervical cancer[J]. N Engl J Med,2003,348(6):518-527. doi: 10.1056/NEJMoa021641 [17] Franceschi S,Rajkumar R,Snijders P J,et al. Papillomavirus infection in rural women in southern India[J]. Br J Cancer,2005,92(3):601-606. doi: 10.1038/sj.bjc.6602348 [18] Nielsen A,Kjaer S K,Munk C,et al. Type-specific HPV infection and multiple HPV types: Prevalence and risk factor profile in nearly 12,000 younger and older Danish women[J]. Sex Transm Dis,2008,35(3):276-282. doi: 10.1097/OLQ.0b013e31815ac5c7 [19] Liu J,Ma S,Qin C,et al. Prevalence and genotype distribution of human papillomavirus in Zhengzhou,China,in 2016[J]. Arch Virol,2020,165(3):731-736. doi: 10.1007/s00705-019-04515-3 [20] Wu H,Wu E,Ma L,et al. Lineage distribution and E2 sequence variation of high-risk human papillomavirus types isolated from patients with cervical cancer in Sichuan Province,China[J]. Arch Virol,2015,160(11):2845-2855. doi: 10.1007/s00705-015-2559-z [21] Dai S,Yao Y,Yan Z,et al. The association of human papillomavirus type 16 E2 variations with cervical cancer in a Han Chinese population[J]. Infect Genet Evol,2018,64:241-248. doi: 10.1016/j.meegid.2018.07.002 [22] Morgan E L,Scarth J A,Patterson M R,et al. E6-mediated activation of JNK drives EGFR signalling to promote proliferation and viral oncoprotein expression in cervical cancer[J]. Cell Death Differ,2020,28(5):1669-1687. [23] Scarth J A,Patterson M R,Morgan E L,et al. The human papillomavirus oncoproteins: A review of the host pathways targeted on the road to transformation[J]. J Gen Virol,2021,102(3):001540. [24] Howie H L,Katzenellenbogen R A,Galloway D A. Papillomavirus E6 proteins[J]. Virology,2009,384(2):324-334. doi: 10.1016/j.virol.2008.11.017 [25] Hiller T,Stubenrauch F,Iftner T. Isolation and functional analysis of five HPVE6 variants with respect to p53 degradation[J]. J Med Virol,2008,80(3):478-483. doi: 10.1002/jmv.21093 [26] Roman A,Munger K. The papillomavirus E7 proteins[J]. Virology,2013,445(1-2):138-168. doi: 10.1016/j.virol.2013.04.013 [27] Kirnbauer R,Taub J,Greenstone H,et al. Efficient self-assembly of human papillomavirus type 16 L1 and L1-L2 into virus-like particles[J]. J Virol,1993,67(12):6929-6936. doi: 10.1128/jvi.67.12.6929-6936.1993 [28] Tan W,Felber B K,Zolotukhin A S,et al. Efficient expression of the human papillomavirus type 16 L1 protein in epithelial cells by using Rev and the Rev-responsive element of human immunodeficiency virus or the cis-acting transactivation element of simian retrovirus type 1[J]. J Virol,1995,69(9):5607-5620. doi: 10.1128/jvi.69.9.5607-5620.1995 -






 下载:
下载: