Molecular Epidemiological Investigation of the Fourth Human Case of Eurasian Avian-like H1N1 Swine Influenza Virus Infection in Yunnan Province
-
摘要:
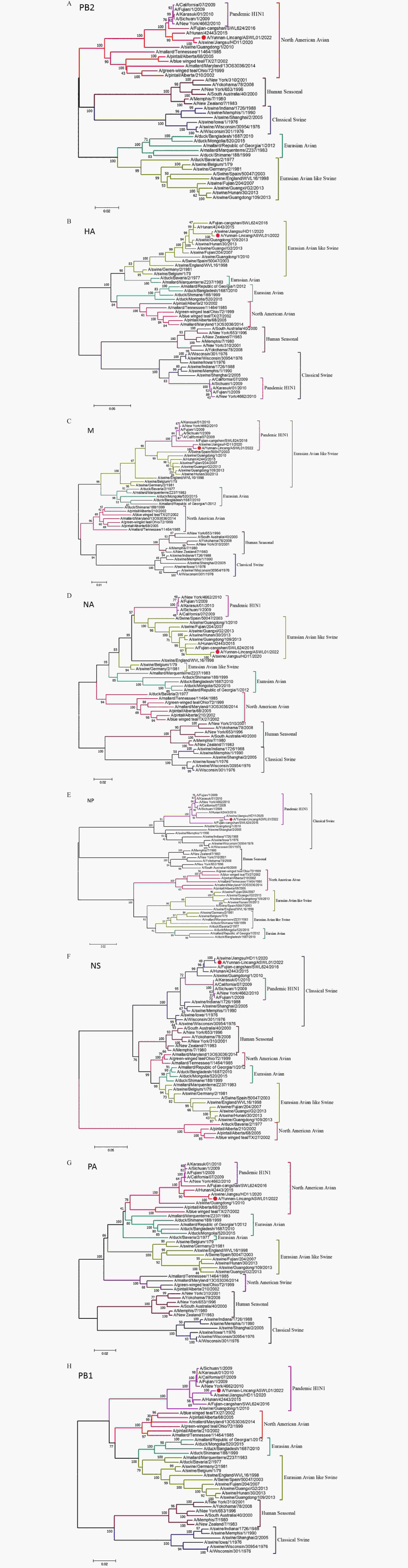
目的 对2022年云南省境内发现的1例(全省第4例)欧亚类禽猪流感病例进行分子流行病学调查,掌握其基因特征,揭示该新亚型欧亚类禽猪流感病毒对人类健康的潜在影响。 方法 使用实时荧光定量PCR检测技术对病例的咽拭子标本、密切接触者和居住环境标本进行核酸检测。使用MDCK细胞进行阳性标本病毒分离,使用豚鼠血红细胞凝集实验和实时荧光定量RT-PCR方法对细胞培养物进行鉴定。通过Illumina Miseq二代基因测序平台进行全基因组测序,使用MEGA7.0软件构建系统发育树并分析其基因分子特征。 结果 成功分离云南省首例G5基因型欧亚类禽猪流感病毒,并获得该病毒全基因组序列。该病毒具有在哺乳动物的适应性或毒力、传播性增强的相关分子特征,与江苏分离的一株猪源毒株序列具有99.2%~99.7%的核苷酸一致性。该病毒在人群中传播的风险进一步增加。 结论 加强猪流感监测工作对于预防可感染人类的新型流感病毒亚型具有重要意义。 -
关键词:
- 欧亚类禽H1NI猪流感 /
- 二代测序 /
- 系统发育分析 /
- 氨基酸特征
Abstract:Objective To conduct a unique and pioneering molecular epidemiological investigation of a case of Eurasian avian-like H1N1 swine influenza identified in Yunnan Province in 2022 (the fourth such case in the province) and to understand its genetic characteristics so as to reveal its potential impact on human health. Methods Real-time fluorescent quantitative PCR detection technology was used for the nucleic acid testing of the case's pharyngeal swab samples, close contacts, and environmental samples from the living area. Positive samples were subjected to virus isolation using MDCK cells. Cell cultures were authenticated using erythrocyte agglutination assay with guinea pig blood and real-time fluorescence quantitative RT-PCR. Whole genome sequencing was performed using the Illumina MiseqNext-generation sequencing platform, and a phylogenetic tree was constructed using MEGA 7.0 software to analyze the genetic molecular characteristics. Results The first G5 genotype Eurasian avian-like H1N1 swine influenza virus in Yunnan Province was successfully isolated, and the whole genome sequence of the virus was obtained. This virus possessed the molecular characteristics associated with increased adaptability, virulence, or transmissibility in mammals and had a nucleotide consistency of 99.2%~99.7% with a porcine strain isolated in Jiangsu province. These findings underscored the potential threat this virus poses to human health. Conclusion The study underscores the importance of further monitoring swine influenza in preventing new influenza virus subtypes that can infect humans. -
表 1 不同型别的欧亚类禽猪流感基因型分型依据
Table 1. Genotyping basis of different types of Eurasian avian-like swine influenza genotypes
分离株不同来源的基因片段 HA NA PB2 PB1 PA NP M NS 基因型 A/Jiangsu/1/2011a EA EA EA EA EA EA EA EA 1 A/swine/Hong_Kong/72/2007a EA EA EA EA EA EA EA CS 2 A/Hunan/ 42443 /2015aEA EA PDM PDM PDM PDM EA CS 3 A/swine/Guangdong/ 1361 /2010aEA EA PDM PDM PDM PDM PDM PDM 4 A/Fujian-cangshan/SWL624/2016a EA EA PDM PDM PDM PDM PDM CS 5 A/swine/Guangdong/1/2010a EA EA CS CS CS CS EA CS 6 A/swine/Hong_Kong/ 2421 /2012aEA EA EA EA EA EA PDM EA 7 A/swine/Guangdong/NS2897/2012a EA EA PDM EA PDM PDM PDM EA 8 A/swine/Guangxi/S2/2013a EA EA EA EA PDM PDM EA CS 9 A/swine/Hong_Kong/268/2012a EA EA CS CS CS CS CS CS 10 A/swine/Hong_Kong/201/2010a EA PDM CS CS CS CS CS CS 11 A/Yunnan-Lincang/ASWL01/2022b EA EA PDM PDM PDM PDM PDM CS 5 a:欧亚禽猪流感基因型分型参考株;b:云南临沧人感染欧亚禽猪流感分离株;EA:基因片段来源于欧亚类禽猪流感病毒;CS:基因片段来源于经典猪流感病毒;PDM:基因片段来源于A(H1N1)pdm09流感病毒。 表 2 A/Yunnan-Lincang/ASWL01/2022毒株的氨基酸分子特征分析
Table 2. The amino acid molecular characteristics of A/Yunnan-Lincang/ASWL01/2022 strain
基因 氨基酸
突变位点意义 毒株 CA/07/09 HN/42443/15 GD/1/10 FJ/624/16 JS/HD11/20 YN/01/22 HA E190D
G225D/E病毒受体结合特异性从α-2,
3-唾液酸转变为α-2,6-唾液酸D
DD
EV
ED
ED
ED
ENA H275Ya
N295Sa神经氨酸酶抑制剂耐药位点 H
NH
NH
NH
NH
NH
NPB2 L89V
T271A G590S
Q591R
D701N被证明对增加聚合酶活性、
增强病毒复制和传播性至关重要V
A
S
R
DV
A
G
R
DV
A
S
R
DV
A
S
Q
DV
A
S
R
DV
A
S
R
DT588I 除增加哺乳动物细胞中的聚合酶活性外,
还被证明介导干扰素-β表达的抑制T I T T I I 627 宿主特异性的决定因素,
禽流感是E,人类流感是KE E E E E E NS1 P42S
D92E增加哺乳动物病毒毒力或适应性 S
DS
DS
DS
DS
DS
DM2 S31N 金刚烷胺类抗病毒药物耐药 N N N N N N M1 T215A 增加哺乳动物病毒毒力或适应性 A A A A A A PA L336M
K356R
S409N增加聚合酶活性、增强病毒复制
和传播性至关重要M
R
NM
R
NL
K
NM
R
NM
R
NM
R
NPB1 X99H
I368R物种之间传播 H
IH
IH
IH
IH
IH
INP Q357K 增加哺乳动物病毒毒力或适应性 K K K K K K a:H1编码。 表 3 中国人感染EAH1N1 SIV病例
Table 3. The cases infected EAH1N1 SIV in China
年份 省份 毒株名 基因型 参考文献 2011 江苏 A/Jiangsu/1/2011 1 Yang,et al[10]. 2012 河北 A/Hebei-Yuhua/SWL1250/2012 1 Wang,et al[11]. 2015 湖南 A/Hunan/ 42443 /20153 Zhu,et al[13]. 2015 云南 A/Yunnan-Longyang/SWL1982/2015 3 Zhu,et al[14]. 2015 云南 A/Yunnan-Wuhua/SWL1869/2015 3 2016 福建 A/Fujian-Cangshan/SWL624/2016 5 Xie,et al[12]. 2018 天津 A/Tianjin-baodi/ 1606 /2018(H1N1)5 Li,et al[15]. 2020 云南 A/Yunnan⁃Mengzi/ 1462 /20204 Li,et al[17]. 2022 云南 A/Yunnan-Lincang/ASWL01/2022 5 孙艳红,等. -
[1] Schnitzler S U,Schnitzler P. An update on swine-origin influenza virus A/H1N1: A review[J]. Virus Genes,2009,39(3):279-292. doi: 10.1007/s11262-009-0404-8 [2] Li H,Leng H,Tang S,et al. Prevalence,genetics and evolutionary properties of Eurasian avian-like H1N1 swine influenza viruses in Liaoning[J]. Viruses,2022,14(3):643 doi: 10.3390/v14030643 [3] Scholtissek C,B ü rger H,Kistner O,et al. The nucleoprotein as a possible major factor in determining host specificity ofinfluenza H3N2 viruses[J]. Virology,1985,147(2):287-294. doi: 10.1016/0042-6822(85)90131-X [4] Dawood F S,Jain S,Finelli L,et al. Emergence of a novel swine-origin influenza A (H1N1) virus in humans[J]. N Engl J Med,2009,360(25):2605-2615. doi: 10.1056/NEJMoa0903810 [5] Pensaert M,Ottis K,Vandeputte J,et al. Evidence for the natural transmission of influenza A virus from wild ducts toswine and its potential importance for man[J]. Bull World Health Organ,1981,59(1):75-78. [6] Zhu H,Webby R,Lam T T,et al. History of swine influenza viruses in Asia[J]. Curr Top Microbiol Immunol,2013,370:57-68. [7] Feng Z,Zhu W,Yang L,et al. Epidemiology and genotypic diversity of Eurasian avian-like H1N1 swine influenza viruses in China[J]. Virol Sin,2021,36(1):43-51. doi: 10.1007/s12250-020-00257-8 [8] Yang H,Chen Y,Qiao C,et al. Prevalence,genetics,and transmissibility in ferrets of Eurasian avian-like H1N1swine influenza viruses[J]. Proc Natl Acad Sci U S A,2016,113(2):392-397. doi: 10.1073/pnas.1522643113 [9] Gregory V,Bennett M,Thomas Y,et al. Human infection by a swine influenza A (H1N1) virus in Switzerland[J]. Arch Virol,2003,148(4):793-802. doi: 10.1007/s00705-002-0953-9 [10] Yang H,Qiao C,Tang X,et al. Human infection from avian-like influenza A (H1N1) viruses in pigs,China[J]. Emerg Infect Dis,2012,18(7):1144-1146. doi: 10.3201/eid1807.120009 [11] Wang D Y,Qi S X,Li X Y,et al. Human infection with Eurasian avian-like influenza A(H1N1) virus,China[J]. Emerg Infect Dis,2013,19(10):1709-1711. doi: 10.3201/eid1910.130420 [12] Xie J F,Zhang Y H,Zhao L,et al. Emergence of Eurasian avian-like swine influenza A (H1N1) virus from an adultcase in Fujian province,China[J]. Virol Sin,2018,33(3):282-286. doi: 10.1007/s12250-018-0034-1 [13] Zhu W,Zhang H,Xiang X,et al. Reassortant Eurasian avian-like influenza A(H1N1) virus from a severely illchild,hunan province,China,2015[J]. Emerg Infect Dis,2016,22(11):1930-1936. doi: 10.3201/eid2211.160181 [14] Zhu W,Feng Z,Chen Y,et al. Mammalian-adaptive mutation NP-Q357K in Eurasian H1N1 swine influenza virusesdetermines the virulence phenotype in mice[J]. Emerg Microbes Infect,2019,8(1):989-999. doi: 10.1080/22221751.2019.1635873 [15] Li X,Guo L,Liu C,et al. Human infection with a novel reassortant Eurasian-avian lineage swine H1N1 virusin northern China[J]. Emerg Microbes Infect,2019,8(1):1535-1545. doi: 10.1080/22221751.2019.1679611 [16] Qi X,Cui L,Jiao Y,et al. Antigenic and genetic characterization of a European avian-like H1N1 swine influenza virus from a boy in China in 2011[J]. Arch Virol,2013,158(1):39-53. doi: 10.1007/s00705-012-1423-7 [17] 李梓,赵晓南,黄维娟,等. 云南省首例人感染G4基因型欧亚类禽H1N1猪流感病毒病原学特征分析[J]. 病毒学报,2022,38(2):290-297. [18] 中国国家流感中心. 全国流感监测技术指南(2017版)[EB/OL]. (2017-09-30). https://ivdc.chinacdc.cn/cnic/fascc/201802/t20180202_158592.htm. [19] Vijaykrishna D,Smith G J,Pybus O G,et al. Long-term evolution and transmission dynamics of swine influenza A virus[J]. Nature,2011,473(7348):519-522. doi: 10.1038/nature10004 [20] Zhu H,Li X,Chen H,et al. Genetic characterization and pathogenicity of a Eurasian avian-like H1N1 swineinfluenza reassortant virus[J]. Virol J,2022,19(1):205. doi: 10.1186/s12985-022-01936-6 [21] Vincent A,Awada L,Brown I,et al. Review of influenza A virus in swine worldwide: A call for increased surveillanceand research[J]. Zoonoses Public Health,2014,61(1):4-17. doi: 10.1111/zph.12049 [22] Yang H,Chen Y,Shi J,et al. Reassortant H1N1 influenza virus vaccines protect pigs against pandemic H1N1influenza virus and H1N2 swine influenza virus challenge[J]. Vet Microbiol,2011,152(3-4):229-234. doi: 10.1016/j.vetmic.2011.04.032 [23] Qiao C,Liu L,Yang H,et al. Novel triple reassortant H1N2 influenza viruses bearing six internal genes of thepandemic 2009/H1N1 influenza virus were detected in pigs in China[J]. J Clin Virol,2014,61(4):529-534. doi: 10.1016/j.jcv.2014.10.014 [24] Gu M,Chen K,Ge Z,et al. Zoonotic threat of G4 genotype Eurasian avian-like swine influenza A(H1N1)viruses,China,2020[J]. Emerg Infect Dis,2022,28(8):1664-1668. doi: 10.3201/eid2808.212530 [25] Sun H,Xiao Y,Liu J,et al. Prevalent Eurasian avian-like H1N1 swine influenza virus with 2009 pandemic viralgenes facilitating human infection[J]. Proc Natl Acad Sci U S A,2020,117(29):17204-17210. doi: 10.1073/pnas.1921186117 [26] Peiris J S,de Jong M D,Guan Y. Avian influenza virus (H5N1): A threat to human health[J]. Clin Microbiol Rev,2007,20(2):243-267. doi: 10.1128/CMR.00037-06 [27] Webster R G,Bean W J,Gorman O T,et al. Evolution and ecology of influenza A viruses[J]. Microbiol Rev,1992,56(1):152-179. doi: 10.1128/mr.56.1.152-179.1992 [28] Matrosovich M,Tuzikov A,Bovin N,et al. Early alterations of the receptor-binding properties of H1,H2,and H3 avianinfluenza virus hemagglutinins after their introduction into mammals[J]. J Virol,2000,74(18):8502-8512. doi: 10.1128/JVI.74.18.8502-8512.2000 -





 下载:
下载: