Identification of Papaveraceae Based on Chloroplast DNA matK+rbcL+ trnH-psbA
-
摘要:
目的 利用DNA条形码技术探索罂粟的鉴定方法,寻找可靠的DNA条形码序列对罂粟属植物进行种内和种间的鉴别。 方法 采用5种不同种类的虞美人和花菱草作为罂粟鉴别的种内植物、种间植物。利用叶绿体基因组matK、rbcL、trnH-psbA和核糖体基因组ITS2、ITS4、ITS5引物对提取的DNA进行扩增,并双向测序拼接,利用MEGA.X.对序列进行分析比对,计算种内、种间遗传距离,同时使用该软件构建NJ系统发育树。 结果 将测序结果录入Genbank中进行比对分析,结果显示单一DNA条形码序列无法有效区分虞美人与罂粟。通过Genbank下载数据使用matK+rbcL+trnH-psbA组合条形码构建系统发育树成功,对罂粟和虞美人样本测序结果进行验证。经验证,matK+rbcL+trnH-psbA组合条形码构建NJ发育树成功,能有效区分虞美人和罂粟。 结论 matK+rbcL+trnH-psbA作为组合DNA条形码使用,可以有效鉴别罂粟属内植物。 -
关键词:
- DNA条形码 /
- matK+rbcL+trnH-psbA /
- 分子鉴定 /
- 罂粟
Abstract:Objective To explore the identification method of Papaver with the use of DNA barcoding technology and find reliable DNA barcoding sequence for intraspecific and interspecific identification of Papaver. Methods Five different species of Papaver rhoeas and Eschscholtzia californica Cham were used as intraspecific and interspecific plants for the identification of Papaver. The extracted DNA was amplified using primers matK, rbcL, trnH-psbA from chloroplast genomes and ITS2, ITS4, ITS5 from ribosomal genomes, and then bidirectional sequencing and splicing were performed using MEGA X. Sequences were analyzed and aligned, intraspecific and interspecific genetic distances were calculated, and N-J phylogenetic trees were constructed using the software. Results The sequencing results, which were entered into Genbank for comparative analysis, showed that a single DNA barcode sequence was not effective in distinguishing between Papaver somniferum and Papaver rhoeas. At the same time, a phylogenetic tree was successfully constructed using matK+rbcL+trnH-psbA combined barcode through Genbank download data, and the sequencing results of Papaver samples in this study were verified by this method. The combination of matK+rbcL+trnH-psbA barcode successfully constructed the N-J tree, which could effectively distinguish between Papaver somniferum and Papaver rhoeas in this study. Conclusion matK+rbcL+ trnH-psbA can be used as a combined DNA barcode for the identification of Papaver. -
Key words:
- DNA barcoding /
- matK+rbcL+trnH-psbA /
- Molecular identification /
- Papaveraceae
-
罂粟科全世界约有38属700余种,我国有18属362种,主要分布于我国的西南部和西北部[1-2]。紫堇属、绿绒蒿属、花菱草属与罂粟属同属于罂粟科,是近亲关系的植物。罂粟属中的不同种植物,又以虞美人(Papaver rhoeas.L)与罂粟最为相似。虞美人因其花朵鲜艳,有很高的观赏价值,被作为园林景观植物材料广泛应用[1, 3]。
罂粟(Papaver somniferum.L)是“非法种植罂粟原植物罪”犯罪构成中的核心要件,明确种植对象是罂粟对定罪量刑十分重要。目前,罂粟的检验鉴定主要依据形态学,区别于传统主要依靠形态学的罂粟原植株鉴定方法,DNA条形码技术不受个体形态、发育阶段的影响,通用性强、准确性高。DNA条形码技术(DNA barcoding)是利用生物基因组中普遍存在的一段较短的、标准的DNA序列作为分子标记,通过参考数据库同源比对,以及物种进化树来确定物种间的亲缘关系[4]。
因为罂粟被管制的特殊性,罂粟条形码DNA研究比较少,大多数研究直接使用Genbank中的数据来筛选DNA条形码[5]。由于Genbank中数据来源的不确定,缺乏标准的鉴定规范,以及大量进口的特殊虞美人品种,都为罂粟检验鉴定的具体司法实践带来了诸多实际困难。罂粟的DNA鉴定仍有巨大的研究空间,目前市面上也没有成熟可靠的罂粟DNA试剂盒可供选择。本研究以5种不同亚种的虞美人作为种内比对研究对象,将花菱草属的加州虞美人(花菱草)作为种间比对对象,从叶绿体基因序列psbA-trnH、matK和rbcL以及核糖体基因序列ITS着手,拟探索一种可行的鉴定方法,为今后罂粟鉴定的规范作探索。
1. 材料与方法
1.1 实验材料
从安徽宿州植物培育基地、中国科学院武汉植物园、广州进口花卉园艺培植基地、成都花卉园艺培育基地处获得加州虞美人(Eschscholtzia californica Cham)、东方虞美人(Papaver Oriental.L)、沙地虞美人(Papaver ammophilum)、阿尔卑斯虞美人(Papaver alpinum)、塞浦路斯虞美人(Papaver cyprium)、冰岛虞美人(Papaver nudicaule)。从昆明市公安局禁毒支队处获得云南本土罂粟和新疆地区罂粟(Papaver somniferum.L)植株和种子,见表1。
表 1 供试样本编号与分组Table 1. Serial numbers of laboratory samples and section简称 名称 拉丁学名 数量 种属 Genbank中是否有数据 分组 PSL 云南罂粟 Papaver somniferum.L 25株 罂粟属 有 入库比对组 PSL-1 新疆罂粟 Papaver somniferum.L 20株 罂粟属 有 入库比对组 POL 东方虞美人 Papaver Oriental.L 15株 罂粟属 有 入库比对组 PAM 沙地虞美人 Papaver ammophilum 15株 罂粟属 无 待鉴定组 PAL 阿尔卑斯虞美人 Papaver alpinum 15株 罂粟属 有 入库比对组 PCY 塞浦路斯虞美人 Papaver cyprium 15株 罂粟属 无 待鉴定组 PNU 冰岛虞美人 Papaver nudicaule 15株 罂粟属 有 入库比对组 ECC 加州虞美人 Eschscholtzia californica Cham 10株 花菱草属 有 入库比对组 1.2 实验方法与步骤
1.2.1 DNA的提取
从上述8种植物每种植株取其新鲜叶片,按植物种类混合,每种植物各取20 mg干燥样本,加入液氮充分研磨,采用TSINGKE植物DNA提取试剂盒(普通版)提取植物DNA。
1.2.2 引物的来源
在NCBI(https://www.ncbi.nlm.nih.gov/)中查找到相关序列,并用NCBI primer-blast(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)和Premier.5软件设计出引物,见表2。
表 2 扩增引物序列的设计Table 2. The sequences of primer for amplification引物名称 序列 Tm值(℃) 参考Genbank ID rbcL-F ATGTCACCACAAACAGAAACTAAAGC 58.16 DQ912886 rbcL-R TCGCATGTACCTGCAGTTGC 59.09 matK-F CGATCTATTCATTCAATATTTC 48.90 JF626525 matK-R TCTAGCACACGAAAGTCGAAGT 58.65 ITS4(新设计) GCCTCCGCTTATTGATATCTGC 56.33 KF022360 ITS5(新设计) GGAAGTAAAAGTCGTAACAACAGG 54.28 ITS4-1(查文献) TCCTCCGCTTATTGATATGC 54.53 MT707523 ITS5-1(查文献) GGAAGTAAAAGTCGTAACAAGG 53.29 ITS2-F ATGCGATACTTGGTGTGAAT 52.99 MH711392 ITS2-R GACGCTTCTCCAGACTACAAT 56.26 PSBA-TRNH-F GTTATGCATGAACGTAATGCTC 54.84 AY328311.1 PSBA-TRNH-R CGCGCATGGTGGATTCACAATCC 62.24 1.2.3 PCR扩增
反应体系为50 μL体系,Easy Taq Super Mix:45 μL,正向引物:2 μL(浓度10 μmol/L),反向引物:2 μL(浓度10 μmol/L),DNA模板:1 μL。PCR扩增条件:预变性:98 ℃,2 min;35个循环(变性:98 ℃,10 s;引物退火:56 ℃,10 s;延伸:72 ℃,10 s);终延伸:72 ℃,5 min;4 ℃延长保存。
1.2.4 测序分析
用1%琼脂糖凝胶进行PCR扩增产物电泳验证,对扩增产物委托擎科生物公司进行双向测序并对结果进行分析。通过Sequencing Analysis 5.2分析峰图数据,最后判定数据是否可用,如果可用,结果保存为abi格式和seq格式。
1.2.5 测序结果处理
测序良好的结果用ContigExpress软件进行拼接,去除引物区域和低质量区域。使用MEGA X计算K2P(Kimura 2-parameter)遗传距离,包括计算序列总体平均遗传距离(Average overall mean),种间平均遗传距离(Average between group distances)和种内平均遗传距离(Average within group distances)。使用MEGA X构建N-J发育树。
2. 结果
2.1 扩增结果
使用ITS2、ITS4、ITS5、rbcL、psbA-trnH、matK6种引物序列对8种植物样本DNA进行扩增,冰岛虞美人(PNU)在ITS2、ITS4、ITS5基因序列上均未获得产物。塞浦路斯虞美人(PCY)在rbcL和psbA-trnH 2个基因序列上均未获得产物,见图1。
2.2 测序结果及信息位点评估
2.2.1 对ITS序列的评估
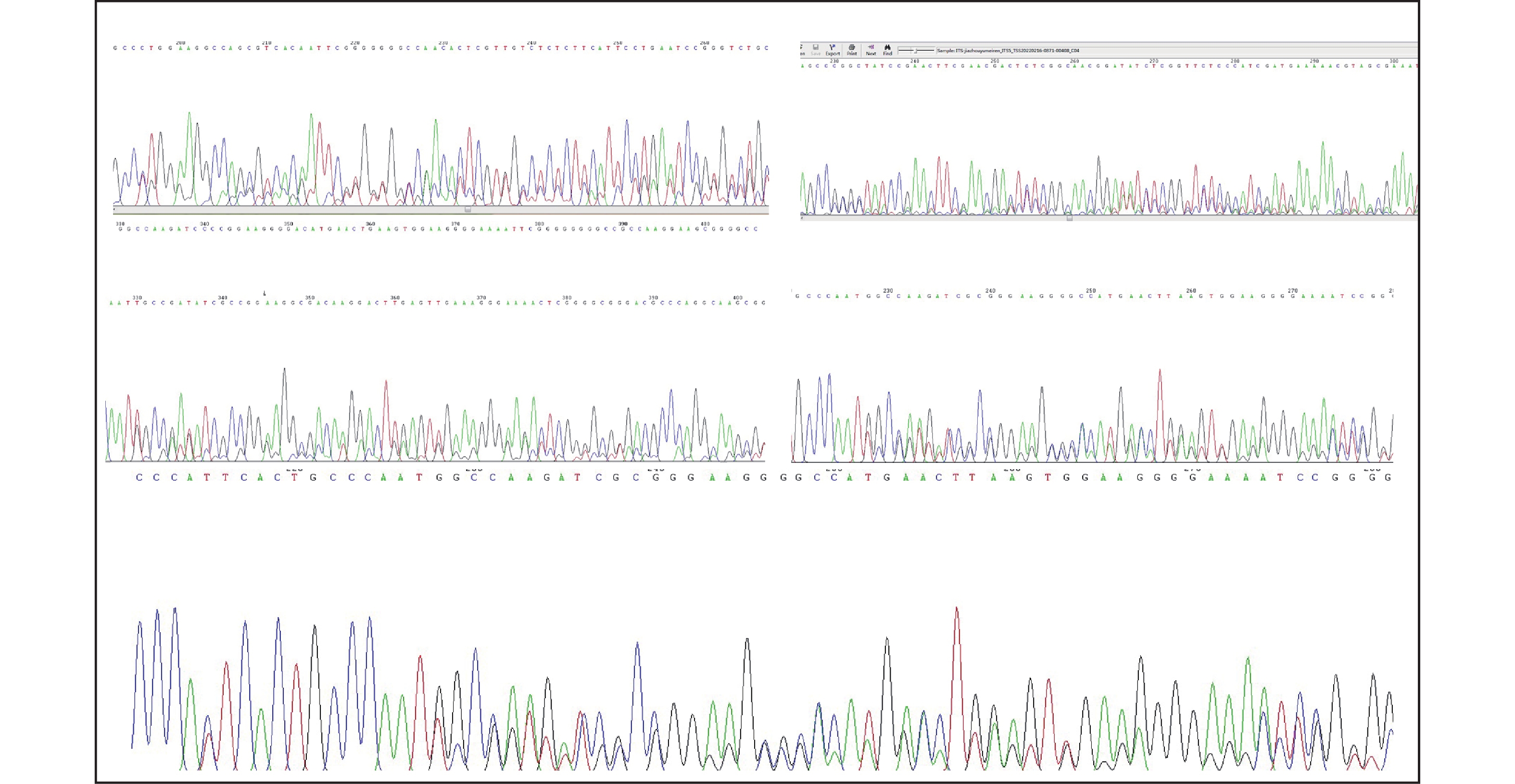
ITS是rDNA中介于18S和5.8S之间(ITS1),以及5.8S和26S之间(ITS2)的非编码转录间隔区,由于ITS1、ITS2序列的进化速率快,常被用于植物种属内和种间亲缘关系的鉴定。本研究针对ITS4和ITS5重新设计的引物进行扩增测序。ITS4和ITS5的碱基序列长度大约是ITS2的2倍,碱基序列基本覆盖ITS2。测序结果分析发现ITS2、ITS4、ITS5在东方虞美人(POL)、沙地虞美人(PAM)、塞浦路斯虞美人(PCY)样本中出现了双峰现象,见图3。考虑到本研究存在多个植株样本混合取样的情况,故采取了单株植物单独取样进行验证,同时对引物和扩增产物采取了PAGE胶纯化,结果显示ITS序列仍然存在双峰的现象,排除了污染的可能性。考虑到本实验测序采取双向测序,从峰形上看,以单峰为主,单峰和双峰混合出现,考虑为DNA单链上存在碱基的缺失造成测序结果移码出现双峰、套峰,与ITS序列本身变异有关。ITS序列虽然容易产生变异,适合作为物种鉴定的后选基因序列,但同时变异也意味着存在大量的碱基突变和碱基缺失,碱基的突变和缺失容易给罂粟鉴定带来困难。
2.2.2 测序结果入库比对
因东方虞美人(POL)、沙地虞美人(PAM)、塞浦路斯虞美人(PCY)的ITS4、ITS5测序结果中存在双峰和套峰的情况,为保证入库比对准确性,表3中仅展示了罂粟(PSL)、阿尔卑斯虞美人(PAL)的ITS4、ITS5测序后入库比对的结果。表3中还选取了测序成功率最高的叶绿体matK测序结果入库进行比对。将ITS、matK序列测序数据去除测序结果两端信号弱或重叠峰区域,用ContigExpress进行拼接,拼接结果在NCBI里使用BLAST Analysis功能进行比对校验,查看其与数据库里基因组测序结果之间的相似匹配情况,见表3。
表 3 罂粟属植物的BLAST比对结果Table 3. BLAST comparison results of Papaver somniferum and its adulterants样本名称 BLAST 最佳匹配物种 最高评分 基因匹配度(%) 数据Genbank编号 PSL-ITS4
PSL-ITS5Papaver rhoeas 虞美人 1292 100 DQ912886 Papaver somniferum 罂粟 1352 100 DQ912880 PAL-ITS4
PSL-ITS5Papaver rhoeas 虞美人 1214 100 AY328311 Papaver nudicaule 野罂粟 1273 100 KF022360 PSL-matK Papaver rhoeas 虞美人 1447 100 MW411801 Papaver somniferum 罂粟 1474 100 MT707522 PAL-matK Papaver nudicaule 野罂粟 1476 100 MW411801 Stylophorum lasiocarpum 金罂粟 1473 100 MW232434 PAM-matK Papaver rhoeas 虞美人 1467 100 NC037831 Papaver somniferum 罂粟 1439 100 MK820043 PYC-matK Papaver rhoeas 罂粟花 1482 100 MF943221 Papaver triniifolium 三叶罂粟 1459 100 MT707533 ECC-matK Eschscholzia californica 加州花菱草 1489 100 Mk281585 2.2.3 遗传距离分析和构建发育树
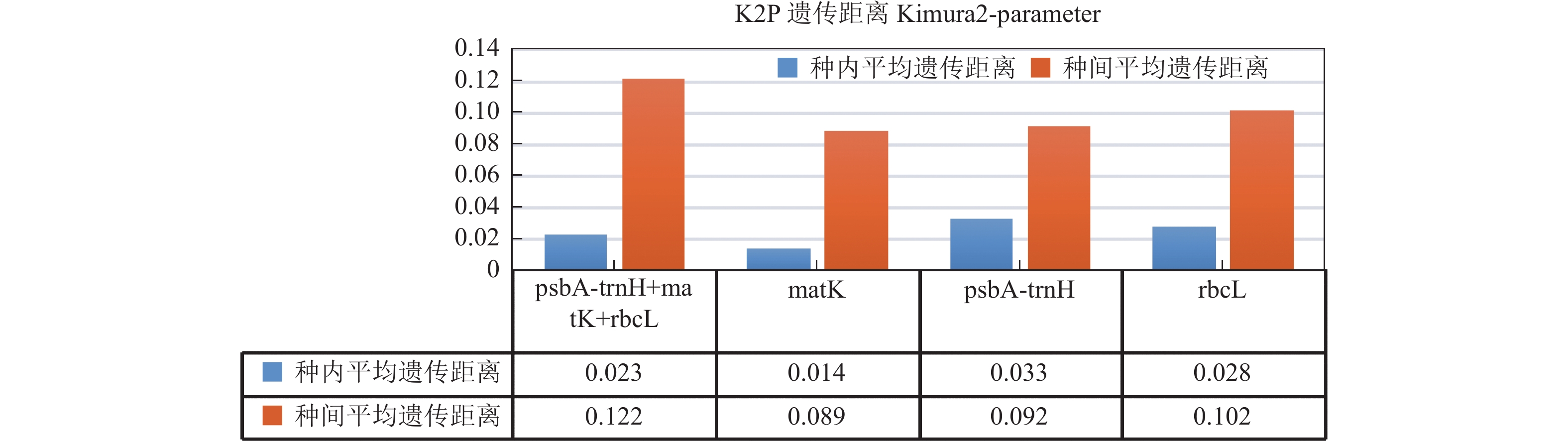
理想的DNA条形码,种内距离应明显小于种间距离。考虑到ITS2、ITS4、ITS5两个基因序列存在双峰情况(图3),冰岛虞美人(PNU)在ITS序列上出现扩增缺失,为保证遗传距离对计算分析准确性,本研究仅利用叶绿体基因进行发育树构建,而放弃利用ITS的相关操作。
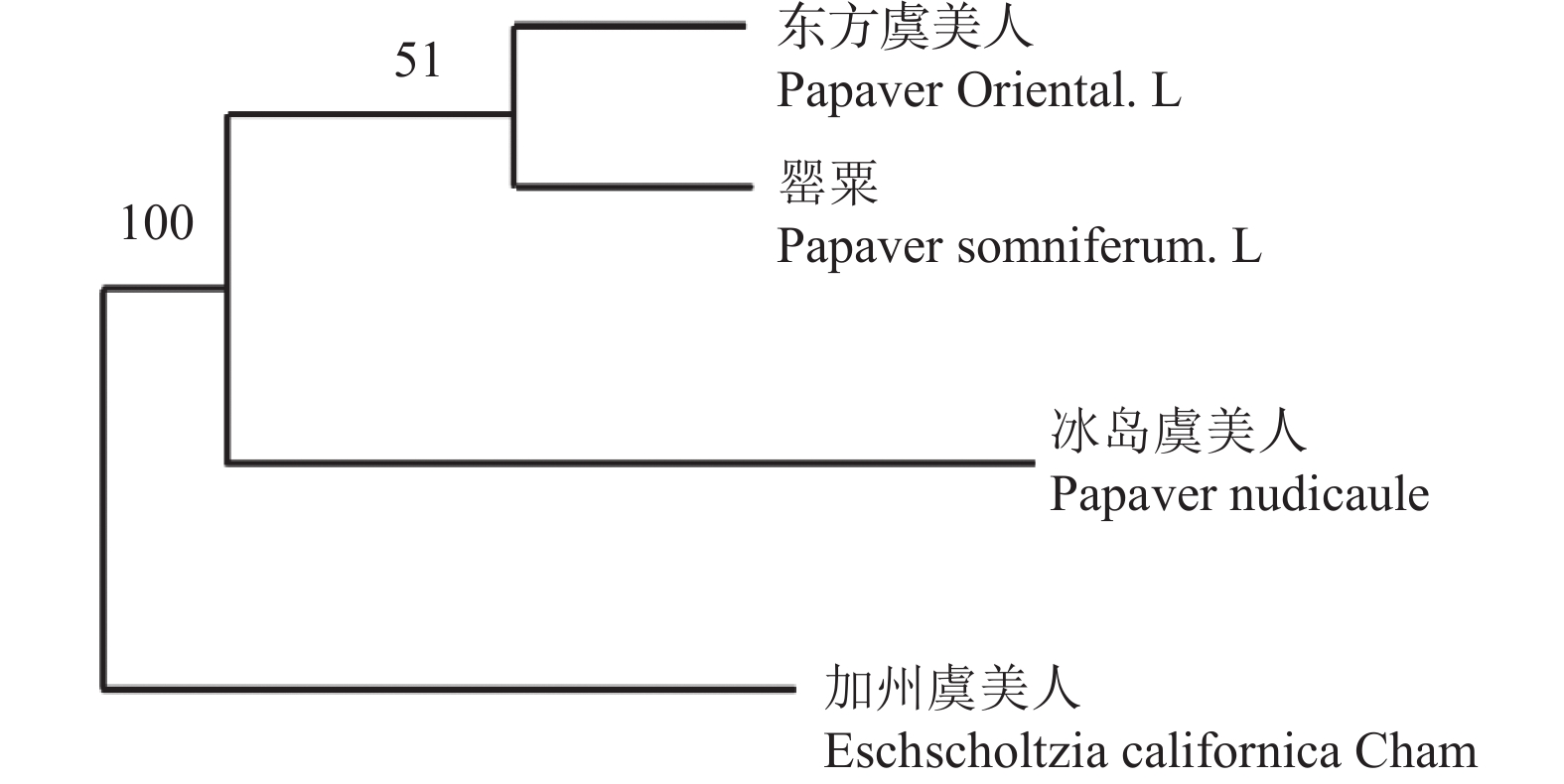
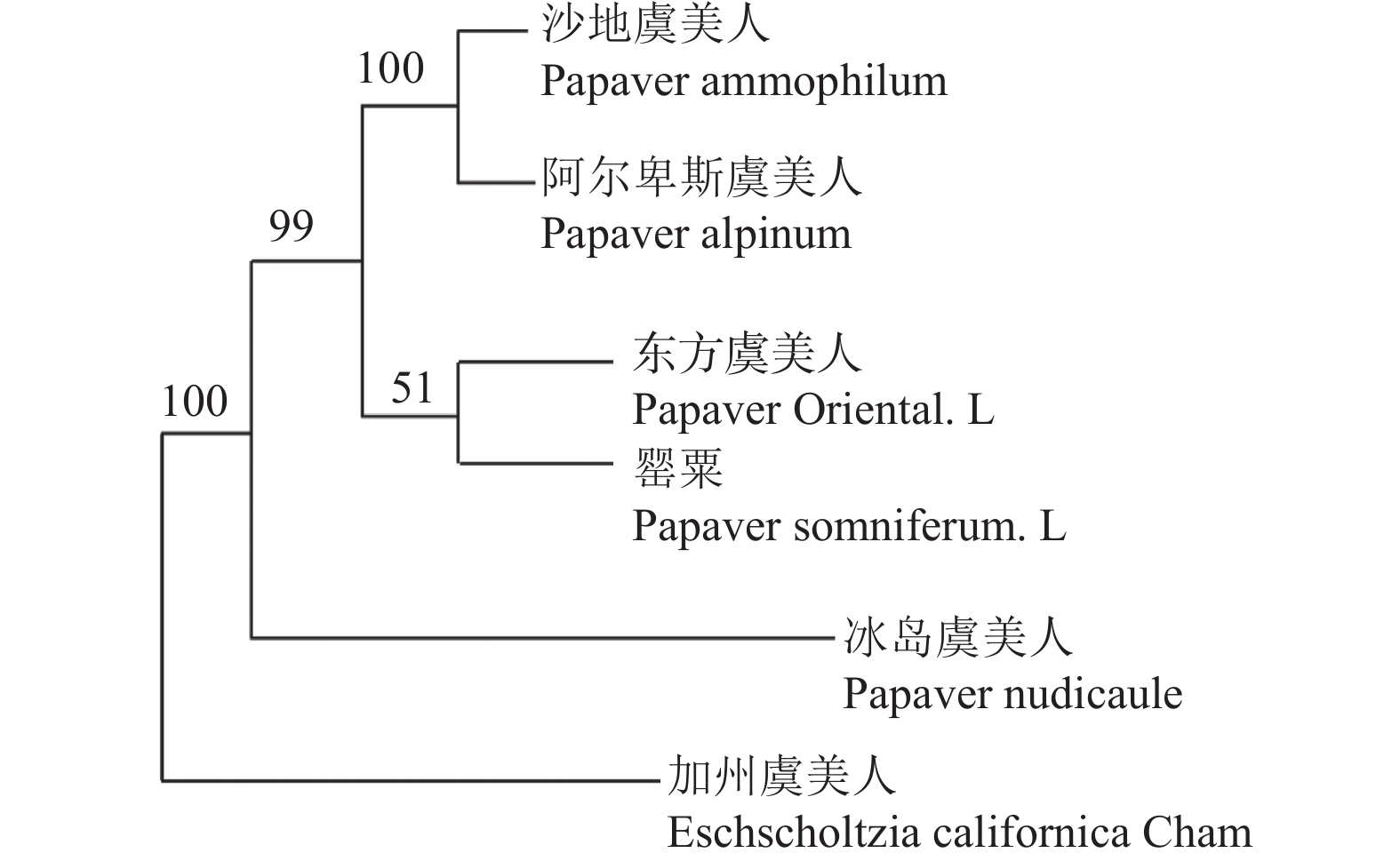
首先,本研究从Genbank中下载了罂粟(Papaver somniferum.L,编号DQ912880)、冰岛虞美人(Papaver nudicaule,编号KF022360)、东方虞美人(Papaver Oriental.L,编号MH711392)、加州虞美人(Eschscholzia californica,编号Mk281585)的叶绿体psbA-trnH、matK和rbcL基因序列数据,通过3个基因序列psbA-trnH + matK + rbcL联合分析,使用MEGA.X软件计算种内、种间遗传距离,并构建N-J系统发育树(图3)。根据psbA-trnH + matK + rbcL联合分析显示种内平均遗传距离明显小于种间平均遗传距离(图4),以及N-J系统发育树对物种的区分度,表明psbA-trnH + matK + rbcL联合分析方法有效。
其次,因塞浦路斯虞美人(PCY)在psbA-trnH、rbcL两个基因序列上出现扩增无产物的现象,故不对塞浦路斯虞美人(PCY)进行N-J建树分析。用MEGA X软件对罂粟(PSL)、阿尔卑斯虞美人(PAL)、东方虞美人(POL)、沙地虞美人(PAM)、冰岛虞美人(PNU)、加州虞美人(ECC)使用psbA-trnH + matK + rbcL联合分析法构建N-J系统发育树邻接法分析序列矩阵所有的碱基作同等加权,空位采用缺失处理,邻接树上各分支的相对支持率采用bootstrap进行1000次重复取样检验。结果(图5)显示,psbA-trnH + matK + rbcL联合分析能够很好地将罂粟与其他罂粟属植物区分开来,特别是能够将Genbank中没有数据的沙地虞美人(PAM)与其他虞美人区分开。
3. 讨论
罂粟作为“非法种植毒品原植物罪”罪名成立的核心,能准确鉴定是否为罂粟植株对定罪量刑十分重要。如今虞美人作为观赏花卉大量出现在市面上,虞美人种类繁多,且属于罂粟科和罂粟属植物,在植物种子和幼苗阶段很难鉴别是否为罂粟。DNA条形码技术为罂粟鉴定提供了新的方向。但由于罂粟被列为毒品被管制的特殊性,对其相关研究不多[6-24]。
3.1 直接使用Genbank中的数据作为鉴定标准存在鉴定风险
目前,国内对罂粟DNA条形码的研究多使用从Genbank中直接下载数据分析。如张爽等[11]是通过从Genbank中下载我国7个罂粟属植物,进行数据分析后认为trnL-trnF对国内罂粟属植物具有很好的区分效果。通过从数据库中下载数据,构建进化树和计算遗传距,对基因序列进行评估,来分析并筛选候选基因序列,该方法路径理论性强。但在实际工作中,直接使用Genbank数据库中的数据作为鉴定标准,存在很大的鉴定风险。
对未知物种进行鉴定,必须要有物种鉴定的行业标准与参照,并用标准的操作获得稳定的结果。本次研究中通过实验数据入库比对后发现,如表3所示,云南罂粟和新疆罂粟的ITS序列经入库比对,最佳匹配物种均有虞美人(Papaver rhoeas)。本研究中的其他虞美人样本,ITS序列入库比对,出现了与罂粟(Papaver somniferum)100%相匹配的情况;表3叶绿体3个基因序列中,沙地虞美人(PAM)的matK基因序列与罂粟(PSL)100%相匹配;在psbA-trnH、rbcL序列中,也都出现了虞美人与罂粟相匹配的现象。这对将Genbank中的数据直接作为金标准来进行物种鉴别造成了阻碍。同时,无论使用UPGMA(类平均法)、ME(Minimum evolution,最小进化法)、NJ(Neighbor-joining,邻接法)或贝叶斯(Bayesian)推断等方法构建系统发育树的过程中,都有很多人为主观操作的环节,如对基因的剪切与整理,使用不同软件的算法等,都会对结果产生影响。
因此,从Genbank中下载数据进行理论分析,并用于DNA条形码的筛选的理论研究是可行的,但将其作为认定罂粟的金标准目前还存在巨大的鉴定风险。
3.2 单独使用ITS、psbA-trnH、rbcL、matK序列无法区分罂粟属内植物
单独使用核基因组序列和叶绿体基因组序列对罂粟属内植物进行区分存在很大的鉴定风险。
罂粟条形码技术研究目前主要集中在ITS序列,虽然ITS核基因序列在其他中药领域有广泛的应用[13-14],但就罂粟属内的植物来说,单独依靠一种基因序列进行测序后入库比对存在很大的鉴定风险。刘玮琦等[23]使用ITS4、ITS5对疆罂粟属(Roemeria)、罂粟属(Papaver)、绿绒蒿属(Meconopsis)、海罂粟属(Glaucium) 和蓟罂粟属(Argemone)构建N-J发育树进行鉴定,显示结果理想。但本实验中使用ITS4、ITS5对罂粟属内的罂粟和虞美人进行鉴定却存在很多困难。一方面是东方虞美人(POL)、沙地虞美人(PAM)、塞浦路斯虞美人(PCY)在ITS4、ITS5中存在碱基缺失的现象,见图2;另一方面是冰岛虞美人(PNU)使用ITS4、ITS5引物扩增后没有扩增产物。
单独用叶绿体psbA-trnH、rbcL、matK候选基因序列进行遗传距离分析和构建N-J发育树也存在风险,市场上常见的塞浦路斯虞美人(PCY)在psbA-trnH、rbcL基因序列没有扩增产物。
3.3 psbA-trnH+matK+rbcL联合分析能够有效将罂粟属内植物
本实验采用了叶绿体基因组条形码联合分析方法,针对罂粟属植物,即罂粟和市场上常见的各种虞美人开展了区分鉴定,取得了良好的效果。相关研究表明,组合条形码的分辨率普遍高于单一条形码,具有更高的普遍适用性[24]。本研究从Genbank中下载了罂粟(Papaver somniferum.L)、冰岛虞美人(Papaver nudicaule)、东方虞美人(Papaver Oriental.L)、加州虞美人(Eschscholzia californica)的叶绿体基因序列数据,通过对3个基因序列psbA-trnH+matK+rbcL联合分析,构建种内、种间N-J系统发育树,确定方法的有效性。然后将本研究中7种植物(塞浦路斯虞美人以外)的叶绿体基因序列进行psbA-trnH+matK+rbcL联合分析,并构建N-J系统发育树。经过对2种发育树的比较,发现psbA-trnH+matK+rbcL联合分析能够有效将罂粟属内的植物进行区分。
3.4 关于罂粟的鉴定标准的展望
《中华人民共和国刑法》中,关于“非法种植毒品原植物罪”中的罂粟,按照中国植物志中的定义,应为罂粟(英文学名为Papaver somniferum.L)[1]。但在Genbank中,能与Papaver somniferum.L在通用DNA条形码基因序列上相匹配的虞美人品种却又有很多,这给司法实践关于鉴定标准的问题带来了很多的实际困难。罂粟的鉴定,其本身对定罪量刑十分关键,具有很强的司法实践意义。随着进口观赏物种的丰富,很多国外的观赏虞美人都含有吗啡、可待因、罂粟碱等成分,这些植物是否需要管制,是否应当被定义为罂粟,都需要检验鉴定的结论作为支撑。
本研究中发现,面对种类繁多复杂的虞美人品种,通过罂粟的核基因DNA条形码ITS序列对罂粟属内的植株进行区分存在困难。单独使用常用的叶绿体DNA条形码,区分效果也不佳。而使用叶绿体psbA-trnH+matK+rbcL联合分析能够针对本研究中的虞美人品种进行有效区分,这可以为今后制定罂粟DNA鉴定行业标准提供一定的理论支持。
目前,我们国家没有自己的DNA条形码数据库,很多研究都是将自己的测序结果录入国外数据库进行比较,得出相应的结论。建立我们国家的罂粟DNA数据库和罂粟DNA鉴定行业标准势在必行。只有从罂粟的概念、DNA数据等方面建立行业标准和规范,方能为推进我们国家对“非法种植毒品原植物罪”的司法实践提供有效遵循。
-
表 1 供试样本编号与分组
Table 1. Serial numbers of laboratory samples and section
简称 名称 拉丁学名 数量 种属 Genbank中是否有数据 分组 PSL 云南罂粟 Papaver somniferum.L 25株 罂粟属 有 入库比对组 PSL-1 新疆罂粟 Papaver somniferum.L 20株 罂粟属 有 入库比对组 POL 东方虞美人 Papaver Oriental.L 15株 罂粟属 有 入库比对组 PAM 沙地虞美人 Papaver ammophilum 15株 罂粟属 无 待鉴定组 PAL 阿尔卑斯虞美人 Papaver alpinum 15株 罂粟属 有 入库比对组 PCY 塞浦路斯虞美人 Papaver cyprium 15株 罂粟属 无 待鉴定组 PNU 冰岛虞美人 Papaver nudicaule 15株 罂粟属 有 入库比对组 ECC 加州虞美人 Eschscholtzia californica Cham 10株 花菱草属 有 入库比对组 表 2 扩增引物序列的设计
Table 2. The sequences of primer for amplification
引物名称 序列 Tm值(℃) 参考Genbank ID rbcL-F ATGTCACCACAAACAGAAACTAAAGC 58.16 DQ912886 rbcL-R TCGCATGTACCTGCAGTTGC 59.09 matK-F CGATCTATTCATTCAATATTTC 48.90 JF626525 matK-R TCTAGCACACGAAAGTCGAAGT 58.65 ITS4(新设计) GCCTCCGCTTATTGATATCTGC 56.33 KF022360 ITS5(新设计) GGAAGTAAAAGTCGTAACAACAGG 54.28 ITS4-1(查文献) TCCTCCGCTTATTGATATGC 54.53 MT707523 ITS5-1(查文献) GGAAGTAAAAGTCGTAACAAGG 53.29 ITS2-F ATGCGATACTTGGTGTGAAT 52.99 MH711392 ITS2-R GACGCTTCTCCAGACTACAAT 56.26 PSBA-TRNH-F GTTATGCATGAACGTAATGCTC 54.84 AY328311.1 PSBA-TRNH-R CGCGCATGGTGGATTCACAATCC 62.24 表 3 罂粟属植物的BLAST比对结果
Table 3. BLAST comparison results of Papaver somniferum and its adulterants
样本名称 BLAST 最佳匹配物种 最高评分 基因匹配度(%) 数据Genbank编号 PSL-ITS4
PSL-ITS5Papaver rhoeas 虞美人 1292 100 DQ912886 Papaver somniferum 罂粟 1352 100 DQ912880 PAL-ITS4
PSL-ITS5Papaver rhoeas 虞美人 1214 100 AY328311 Papaver nudicaule 野罂粟 1273 100 KF022360 PSL-matK Papaver rhoeas 虞美人 1447 100 MW411801 Papaver somniferum 罂粟 1474 100 MT707522 PAL-matK Papaver nudicaule 野罂粟 1476 100 MW411801 Stylophorum lasiocarpum 金罂粟 1473 100 MW232434 PAM-matK Papaver rhoeas 虞美人 1467 100 NC037831 Papaver somniferum 罂粟 1439 100 MK820043 PYC-matK Papaver rhoeas 罂粟花 1482 100 MF943221 Papaver triniifolium 三叶罂粟 1459 100 MT707533 ECC-matK Eschscholzia californica 加州花菱草 1489 100 Mk281585 -
[1] 中国科学院中国植物志编辑委员会. 中国植物志 [M]. 第32卷. 北京: 科学出版社, 1999: 51-60. [2] 段秋关. 毒品的定义及构成要素[J]. 西北大学学报(哲学社会科学版),1999,12(1):46-50. [3] 王白石,高珊,宋炳柯,等. 罂粟分子遗传标记研究现状及开发前景[J]. 中国法医学杂志,2018,33(3):273-272. doi: 10.13618/j.issn.1001-5728.2018.03.012 [4] 宋炳轲,杨雪莹,倪萍娅,等. DNA条形码技术在毒品原植物大麻鉴定中的应用[J]. 广西植物,2014,34(4):24. [5] Wang B,Gao S,Song B,et al. Research progress and prospect on developing of molecular markers for opium poppy (Papaver somniferum L)[J]. Chinese J Forensic Med,2018,33(3):273-275. [6] 姚辉,张辉,陈士林. 《中国药典》收载动物药材DNA条形码鉴定研究策略与方法[J]. 中国现代中药,2019,21(9):1137-1146. [7] Zhang Z,Kong Y,Li Y,et al. Phylogeny of some Papaveraceae plants in Xinjiang based on DNA barcoding technology[J]. Arid Zone Res,2014,31(2):322-328. [8] 娄千,辛天怡,宋经元. DNA条形码技术在中药全产业链的应用进展[J]. 药学学报,2020,55(08):2306-2312. [9] Zhou J,Cui Y,Chen X,et al. Complete chloroplast genomes of Papaver rhoeas and Papaver orientale: Molecular structures, comparative analysis, and phylogenetic analysis[J]. Molecules,2018,23(2):437-452. doi: 10.3390/molecules23020437 [10] 杨鹏,沈文华,石建明,等. 唇形科常见药用植物DNA条形码的鉴定研究[J]. 中草药,2017,48(7):1397-1402. [11] 张爽,刘宇婧,吴沿胜,等. 罂粟属植物核心DNA条形码的筛选[J]. 中国中药杂志,2015,40(15):2964-2969. [12] Lahaye R,Van der Bank M,Bogarin D,et al. DNA barcoding the floras of biodiversity hotspots[J]. Proc Natl Acad Sci USA,2008,105(8):2923. doi: 10.1073/pnas.0709936105 [13] 杨慧洁,杨世海,张淑丽,等. 药用植物DNA条形码研究进展[J]. 中草药,2014,45(18):2581-2587. doi: 10.7501/j.issn.0253-2670.2014.18.001 [14] 付涛,王志龙,钱萍仙. 高等植物DNA条形码最新研究进展及其应用[J]. 核农学报,2016,30(5):887-896. [15] 黄志海,丘小惠,宫璐,等. 板蓝根与南板蓝根及其混淆品的ITS2条形码鉴定[J]. 中药材,2017,40(1):50-53. doi: 10.13863/j.issn1001-4454.2017.01.012 [16] 赵晴. 中药材种子DNA条形码鉴定研究进展[J]. 中草药,2019,50(14):3471-3476. doi: 10.7501/j.issn.0253-2670.2019.14.030 [17] 路帆,程宝文,李虹,等. AFLP技术鉴别罂粟, 虞美人和大麻种属差异的初步研究[J]. 中国法医学杂志,2008,23(3):157. doi: 10.3969/j.issn.1001-5728.2008.03.005 [18] Zhang S,Liu Y,Wu Y S,et al. Screening potential DNA barcode regions of genus Papaver[J]. Zhongguo Zhong Yao Za Zhi,2015,40(15):2964. [19] 张哲,孔艳,李岩. 基于DNA条形码技术对新疆罂粟科部分植物系统发生[J]. 干旱区研究,2014,31(2):322. doi: 10.13866/j.azr.2014.02.003 [20] 杨雪莹,宋炳轲,裴黎,等. 植物物证的DNA条形码鉴定分析[J]. 中国法医学杂志,2015,30(2):189. doi: 10.13618/j.issn.1001-5728.2015.02.023 [21] Lee E J,H wang I K,Kim N Y,et al. An assessment of the utility of universal and specific genetic markers for opium poppy identification[J]. Forensic Sci,2010,55(5):1202-1208. doi: 10.1111/j.1556-4029.2010.01423.x [22] Ren F M, WangY W, Xu Z C, et al. DNA barcoding of Corydalis, the most taxonomically complicated genus of Papaveraceae[J]. Ecol Evol,2019,9(4):1934-1945. doi: 10.1002/ece3.4886 [23] 刘玮琦,梁靓,李兰,等. 基于ITS序列的 DNA条形码技术鉴定罂粟科种子[J]. 中国口岸科学技术,2022,2(4):61-65. doi: 10.3969/j.issn.1002-4689.2022.04.010 [24] 王洁,于维森,步淑华,等. DNA 条形码在有毒高等陆生植物鉴定和溯源中的应用进展[J]. 现代预防医学,2021,48(3):435-437. 期刊类型引用(3)
1. 罗湘胤,游磊,叶峥秀,张泽志,石宪铭,居婷婷,李琳,张勇洪. 药用植物冷水花DNA条形码分析和基因组大小测定. 湖北医药学院学报. 2024(01): 13-17 .  百度学术
百度学术2. 陈梓媛,华中一,袁媛. 全球物种数量最多的叶绿体基因组数据库的建立及应用进展. 中国中药杂志. 2024(23): 6257-6263 .  百度学术
百度学术3. 刘青青,杨开宇,马玉花,施会,冶贵生,冯智鹏. 沙棘rbcL基因的扩增及序列分析. 北方园艺. 2023(21): 42-52 .  百度学术
百度学术其他类型引用(0)
-






 下载:
下载:

 下载:
下载:

