Whole Genome Analysis of Coxsackievirus A10 in Yunnan Province
-
摘要:
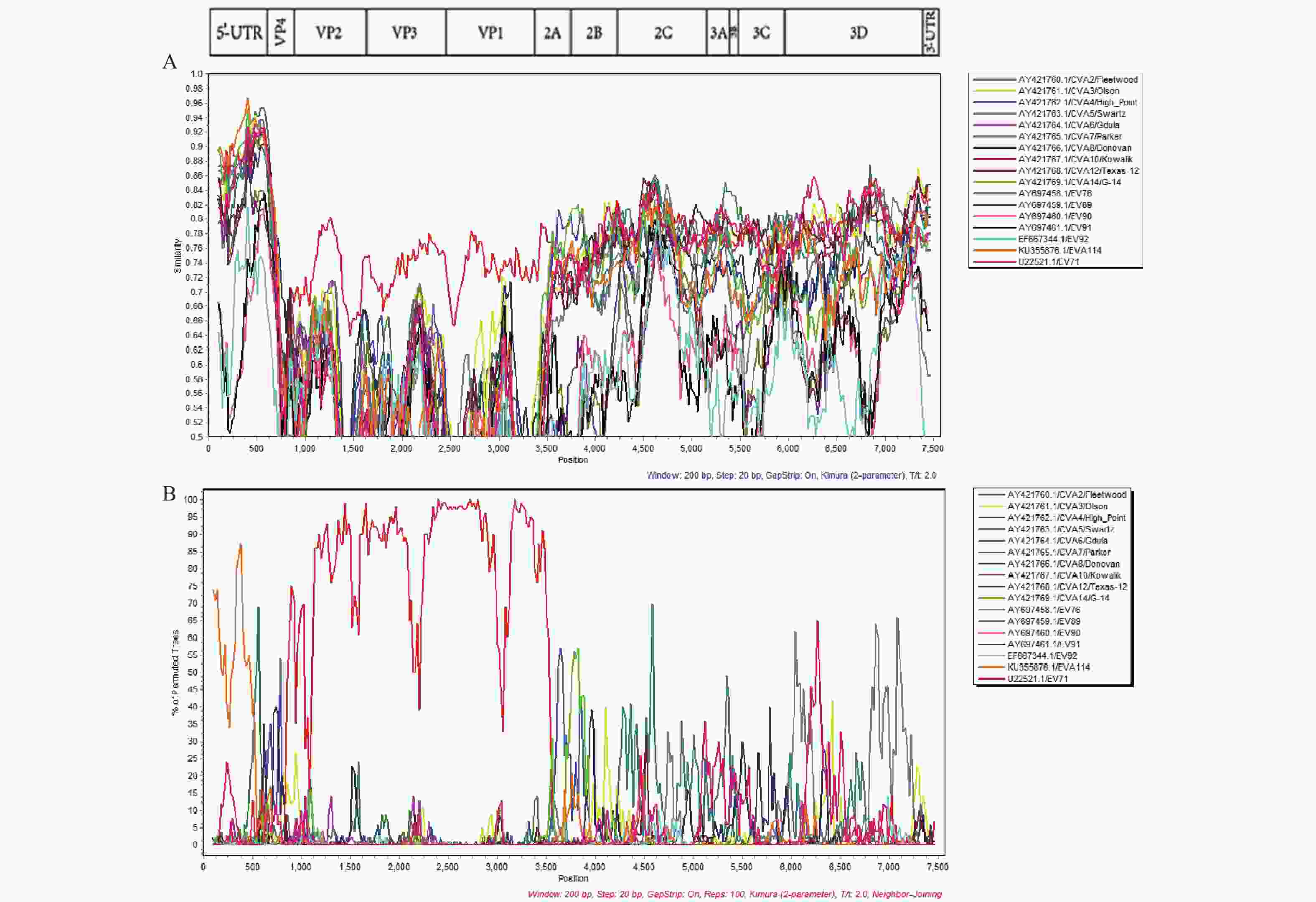
目的 分析从2022年手足口病患儿粪便样品中分离到的柯萨奇病毒A组10型(coxsackievirus A10,CVA10)分离株的全基因组特征。 方法 经人横纹肌肉瘤细胞(human rhabdomyosarcoma,RD)分离得到该分离株,将其命名为155/YN/CHN/2022。提取病毒RNA,采用反转录聚合酶链反应 (reverse transcription polymerase chain reaction,RT-PCR)扩增其VP1序列,鉴定血清型。然后分段全基组扩增并拼接155/YN/CHN/2022的全基因组序列,利用MEGA7.0、DNAStar7.1和Simplot3.5.1等软件对序列进行分析。 结果 CVA10分离株155/YN/CHN/2022经扩增及拼接后得到的全基因组序列长为7403 nt,其中5'UTR区为746 nt,3'UTR区为76 nt,编码区长6552 nt,该分离株与CVA10原型株的核苷酸序列和氨基酸序列一致性分别为78.43%和95.30%,而与其他国内外CVA10分离株的核苷酸序列和氨基酸序列一致性分别为91.18%~95.06%和97.63%~98.54%。系统发育分析发现,155/YN/CHN/2022位于C基因型。P1、P2、P3系统发育结果显示,在P2、P3非编码区,该分离株与其他血清型毒株可能发生过重组。Simplot重组分析也显示,CVA10分离株155/YN/CHN/2022可能在非编码区出现重组事件。 结论 CVA10分离株155/YN/CHN/2022属于C基因型,和近年来中国大陆流行株属于同一基因型。 -
关键词:
- 手足口病 /
- 柯萨奇病毒A组10型 /
- 全基因组 /
- 进化分析
Abstract:Objective To analyze the whole-genome characteristics of a coxsackievirus A10 (CVA10) isolate obtained from fecal samples of children with hand, foot, and mouth disease in 2022. Methods The isolate was obtained from human rhabdomyosarcoma (RD) cells and named 155/YN/CHN/2022. Viral RNA was extracted, and the VP1 sequence was amplified by reverse transcription polymerase chain reaction (RT-PCR) to identify the serotype. The whole genome sequence of 155/YN/CHN/2022 was amplified and assembled. Sequence analyses were performed using software such as MEGA7.0, DNAStar7.1, and Simplot3.5.1. Results The complete genomic sequence of CVA10 isolate 155/YN/CHN/2022 was 7403 nt long, with a 746 nt 5'UTR region, a 76 nt 3'UTR region, and a 6551 nt coding region. The nucleotide and amino acid sequence identities of this isolate compared to the prototype strain of CVA10 were 78.43% and 95.30%, respectively, while the identities compared to other domestic and foreign CVA10 isolates ranged from 91.18% to 95.06% for nucleotide sequences and 97.63% to 98.54% for amino acid sequences. Phylogenetic analysis revealed that 155/YN/CHN/2022 belonged to the C genotype. Phylogenetic analyses of P1, P2, and P3 regions suggested this isolate may have undergone recombination with other serotype strains in the P2 and P3 non-coding regions. Simplot recombination analysis also indicated possible recombination events in the non-coding regions of P2 and P3 for CVA10 isolate 155/YN/CHN/2022. Conclusion CVA10 isolate 155/YN/CHN/2022 belongs to the genotype C, which is consistent with recent epidemic strains in China's mainland. -
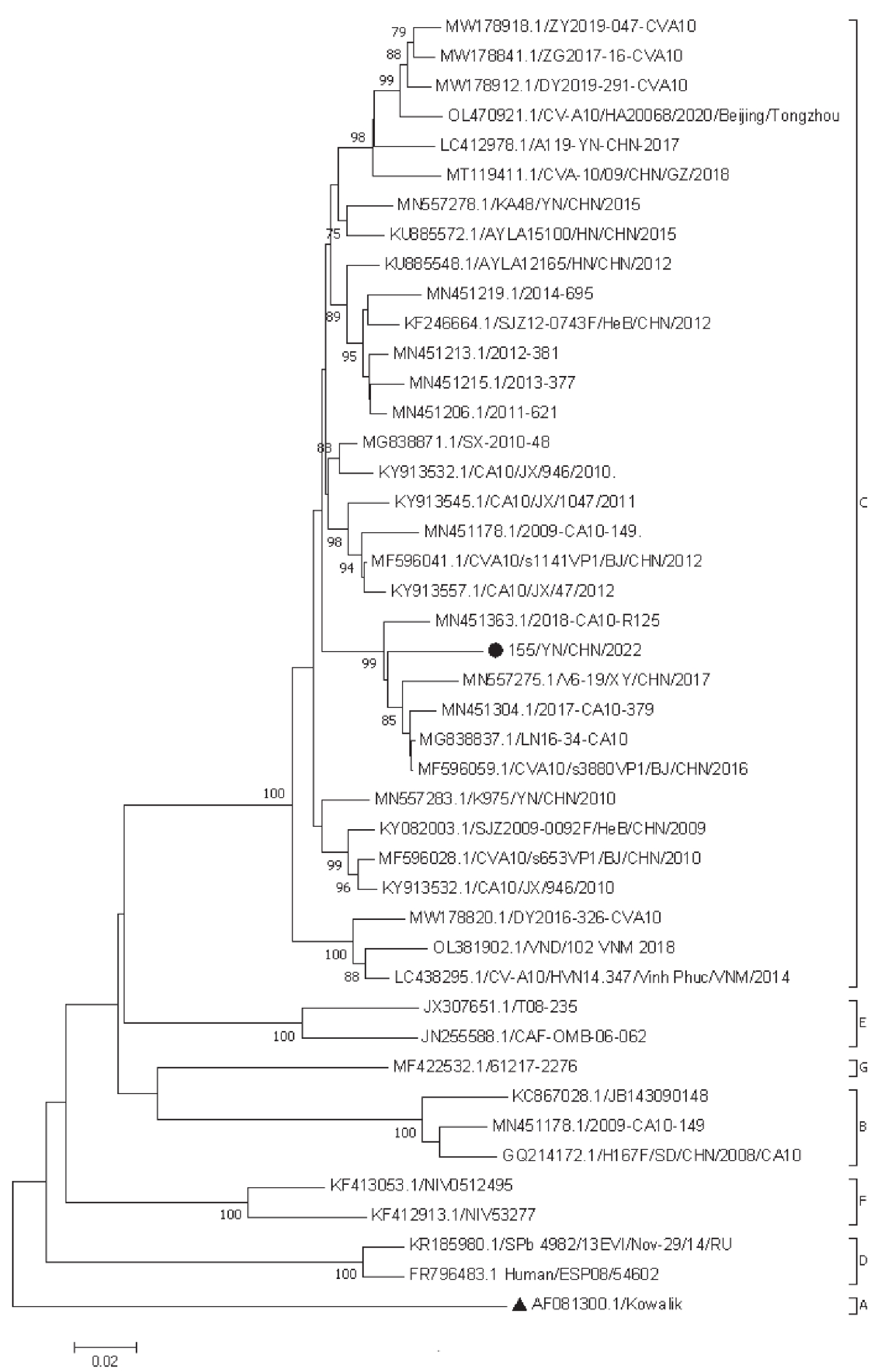
图 2 CVA10毒株与 EVA不同血清型毒株的 P1、P2 和 P3 区系统进化树 (从左到右分别为P1、P2和P3区系统进化树)
注:P1、P2 和 P3 区系统进化树图中的“●”为本研究中的CVA10毒株;“▲”为CVA10原型株;“■”为其他CVA10分离株。
Figure 2. Phylogenetic trees of P1,P2 and P3 regions of CVA10 strain and EVA serotype strains in this study (P1,P2 and P3 region phylogenetic trees from left to right,respectively)
表 1 全基因组序列扩增及测序引物
Table 1. Primers for whole genome sequence amplification and sequencing
引物 序列(5'→3') 核苷酸位置 方向 A-OAS GGRTANCCRTCRTARAACCAYTG 3109 –3089 反向引物 A-OS CCNTGGATHAGYAACCANCA 2268 –2287 正向引物 E201F TTAAAACAGCCTGTGGGTTG 1–20 正向引物 A10N1r GGGTTGCCCCATAGGCAG 812–795 反向引物 A10N1f GTAGCCACTGGAGGATCT 1052 -1069 正向引物 A10N2R CACAGCTTGTTGTGTCACTT 2511 -2493 反向引物 A10N3F CATGTATGTGCCCCCTGGCG 3261 –3280 正向引物 A10N4f CTTAGGCCGCTTAGATGC 3960 –3977 正向引物 A10N5r CCAAGGACACAATATGTC 6598 –6581 反向引物 A10N6f TTGGTTCCGAGGGAGCGG 7042 –7059 正向引物 A10N7f CTTGCCAGATCACCAATT 6046 –6063 正向引物 CA168R GGTTATAACAAATTTACCCCC 7404 –7385 反向引物 表 2 CVA10分离株与其原型株、其他CVA10株核苷酸和氨基酸一致性比对
Table 2. Nucleotide and amino acid sequence identity comparison of CVA10 isolate with prototype and other CVA10 strains in all sequenced genomic regions
基因组
区域原型株 其他A10分离株 核苷酸
一致性(%)氨基酸
一致性(%)核苷酸
一致性(%)氨基酸
一致性(%)5'UTR 83.9~96.2 95.98~98.39 VP4 77.78 94.20 94.69~98.55 94.20~100 VP2 77.66 96.08 93.59~98.30 96.08~100 VP3 76.69 94.3~98.3 93.65~99.15 91.2~100 VP1 76.87 92.95 95.97~100 98.66~100 2A 81.33 95.33 94.22~99.11 92.67~99.33 2B 78.84 96.97 93.94~98.99 95.96~100 2C 83.50 96.05 94.33~98.89 99.39~99.7 3A 82.95 95.35 94.57~90.61 96.51~100 3B 90.91 100.00 93.94~98.48 89.3~96.8 3C 78.08 95.08 93.98~97.87 90.71~97.27 3D 83.08 95.24 90.70~97.12 95.89~97.84 3'UTR 93.83 93.42~97.01 Genome 78.43 95.30 91.18~95.06 97.63~98.54 表 3 155/YN/CHN/2022分离株与其他EVA病毒分离株之间的基因组不同区域的一致性比较
Table 3. Consistency comparison of different genomic regions between 155/YN/CHN/2022 isolates and other EVA virus isolates
基因区段 血清型 毒株 核苷酸同源性(%) 基因登陆号 5'UTR CVA10 PD19-80 98.39 MW929277.1 VP4 A10 144_GD/CHN_2021 98.55 PP078948.1 VP2 A10 GD/CHN_2021 98.30 PP078947.1 VP3 A10 GD/CHN_2021 99.15 PP078948.1 VP1 A10 A296-YN-CHN-2022 100.00 LC791297.1 2A A10 GD/CHN_2021 99.11 PP078948.1 2B A10 GD/CHN_2021 98.99 PP078948.1 2C A10 GD/CHN_2021 98.89 PP078948.1 3A A10 GD/CHN_2021 99.61 PP078948.1 3B A10 GD/CHN_2021 98.48 PP078948.1 3C A10 GD/CHN_2021 97.87 PP078948.1 3D A16 CVA16/TH/MUMT-1/2016 97.12 OM417116.1 3'UTR A16 V18-026 97.01 MH780757.1 Genome A10 GD/CHN_2021 95.06 PP078948.1 P1 A10 GD/CHN_2021 98.65 PP078948.1 P2 A10 GD/CHN_2021 98.96 PP078948.1 P3 A16 C138/CHW/AUS/2016 91.39 MH111071.1 -
[1] 赖晴润,刘长坤,刘艳艳,等. 柯萨奇病毒A组10型的生物学特征和免疫原性分析[J]. 微生物学免疫学进展,2023,51(2):46-52. [2] Duan S,Yang F,Li Y,et al. Pathogenic analysis of coxsackievirus A10 in rhesus macaques[J]. Virologica Sinica,2022,37(4):610-618. doi: 10.1016/j.virs.2022.06.007 [3] Zhu R,Xu L,Zheng Q,et al. Discovery and structural characterization of a therapeutic antibody against coxsackievirus A10[J]. Science Advances,2018,4(9):eaat7459. doi: 10.1126/sciadv.aat7459 [4] Zhu L,Sun Y,Fan J,et al. Structures of coxsackievirus A10 unveil the molecular mechanisms of receptor binding and viral uncoating[J]. Nature communications,2018,9(1):4985. doi: 10.1038/s41467-018-07531-0 [5] Zhang J,Xu D,Liu H,et al. Characterization of coxsackievirus A10 strains isolated from children with hand,foot,and mouth disease[J]. Journal of Medical Virology.,2022,94(2):601-609. doi: 10.1002/jmv.27268 [6] 吴志强,熊焰,刘亭,等. 2016—2018年武汉地区手足口病病原学和流行病学调查[J]. 武汉大学学报(医学版),2019,40(3):429-434. [7] 孙鹏恺. 2019—2023年浙江省金华市手足口病监测与检测分析[D]. 武汉: 长江大学,2024. [8] 王钰,汤晶晶,柏桂珍,等. 上呼吸道感染儿童中肠道病毒流行病学特征分析[J]. 中国病毒病杂志,2024,14(2):152-158. [9] Tapparel C,Siegrist F,Petty T J,et al. Picornavirus and enterovirus diversity with associated human diseases[J]. Infect Genet Evol,2013,14(1):282-293. [10] Li X,Liu Z,Yan X,et al. VP2 residue N142 of coxsackievirus A10 is critical for the interaction with KREMEN1 receptor and neutralizing antibodies and the pathogenicity in mice[J]. PLoS Pathogens,2023,19(10):e1011662. doi: 10.1371/journal.ppat.1011662 [11] Lian H,Yi L,Qiu M,et al. Genomic epidemiology of CVA10 in Guangdong,China,2013–2021[J]. Virology Journal,2024,21(1):122-129. doi: 10.1186/s12985-024-02389-9 [12] 柳鸿艳. 太原市2016年—2020年手足口病流行病学及柯萨奇病毒A10型基因特征分析[D]. 太原: 山西医科大学,2023. [13] 徐丹,杨薇,姚学君,等. 盐城市2018—2019年手足口病和疱疹性咽峡炎病原学特征分析[J]. 江苏预防医学,2022,33(2):158-162. [14] 许丹菡,郭伟,张名,等. 2019年云南省手足口病相关的柯萨奇病毒A组10型分离株的全基因组分析[J]. 中国病毒病杂志,2022,12(4):290-295. [15] Muslin C,Mac Kain A,Bessaud M,et al. Recombination in enteroviruses,a multi-step modular evolutionary process[J]. Viruses,2019,11(9):859-867. doi: 10.3390/v11090859 [16] 樊欢,鲍倡俊,朱立国,等. 2015—2022年江苏省柯萨奇病毒A10型全基因组序列特征分析[J]. 中华微生物学和免疫学杂志,2023,43(12):945-954. [17] Zhang Y X,Huang Y M,Li Q J,et al. A highly conserved amino acid in VP1 regulates maturation of enterovirus 71[J]. PLoS Pathogens.,2017,13(9):e1006625. doi: 10.1371/journal.ppat.1006625 [18] 姚学君,管书慧,刘秀兰,等. 中国大陆地区2004—2016年柯萨奇病毒A组10型的分子流行病学特征分析[J]. 中华疾病控制杂志,2017,21(11):1111-1114. -






 下载:
下载: