Genomic Analysis of Cellular Senescence and Osteoporosis Target Genes and Small Molecule Screening
-
摘要:
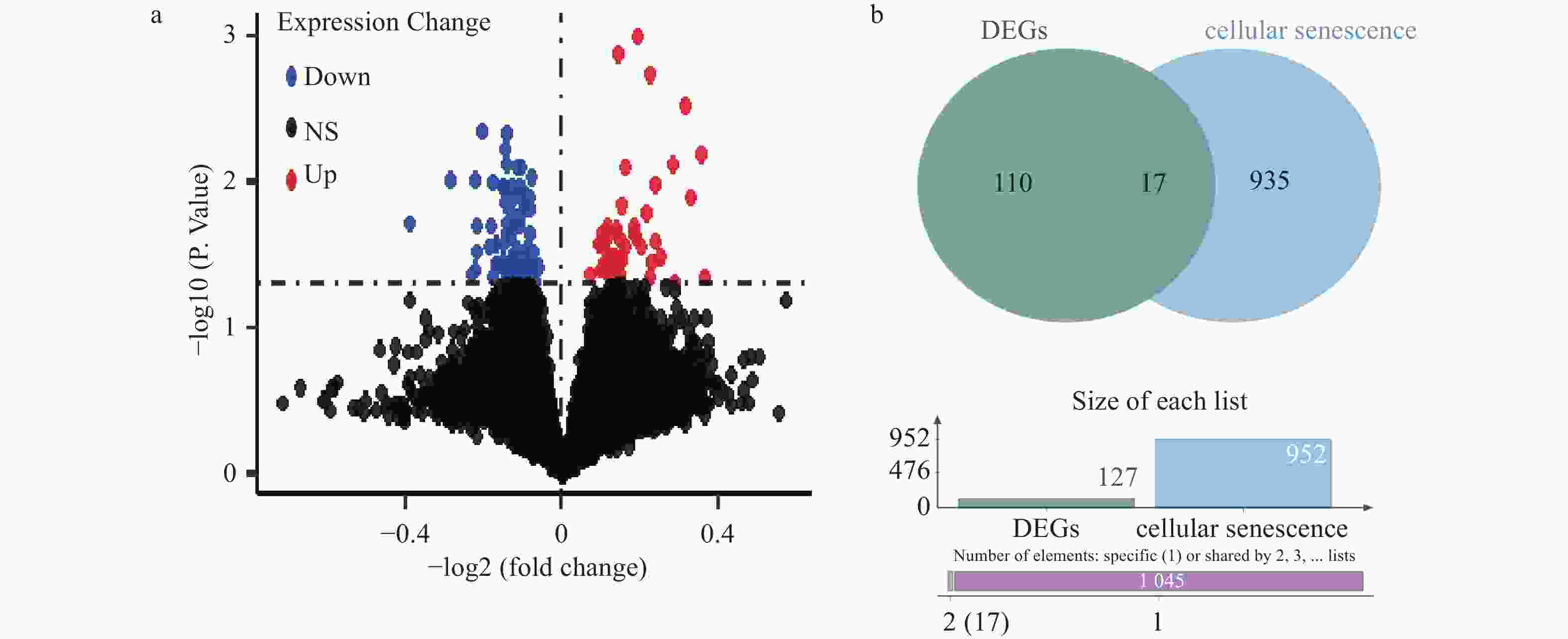
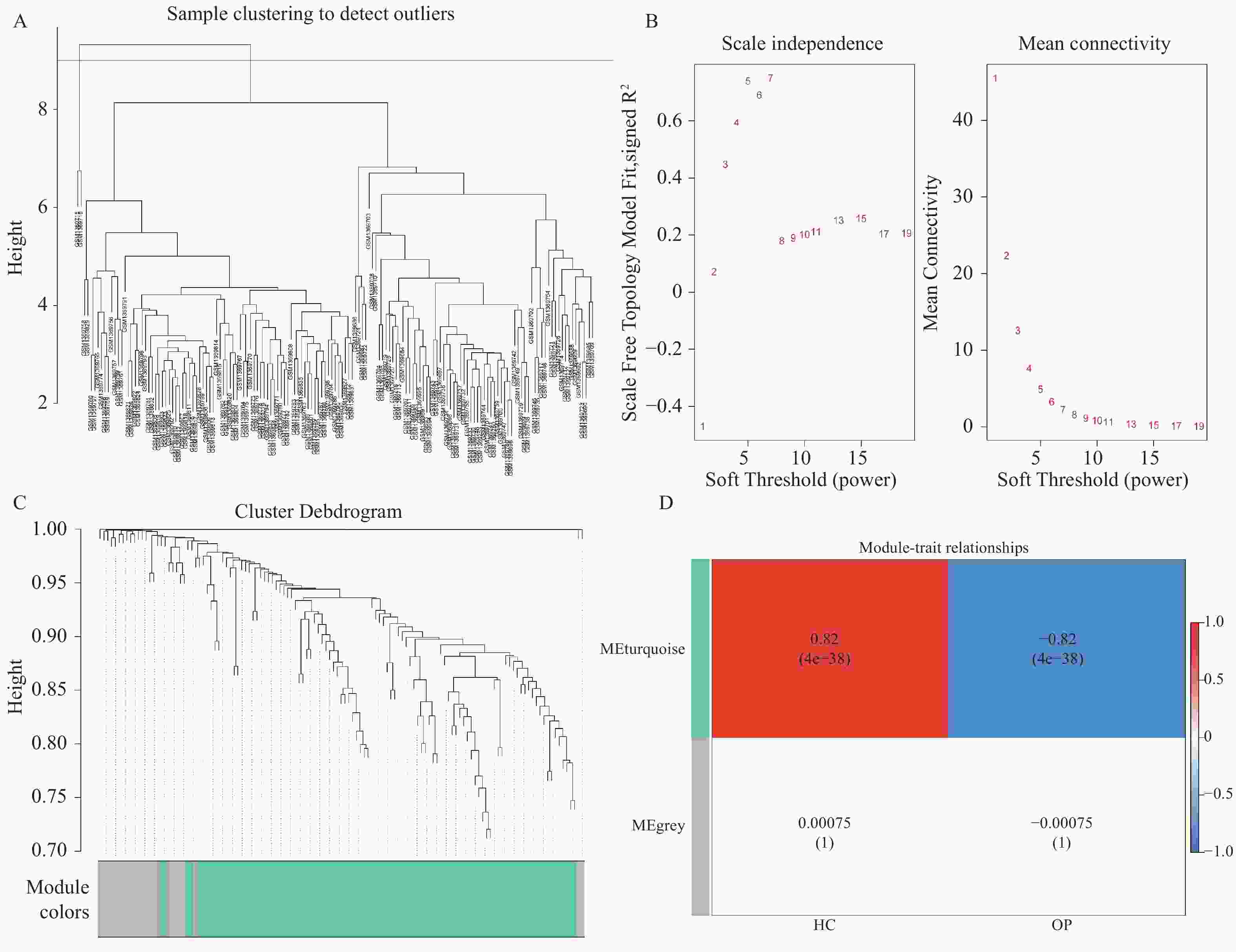
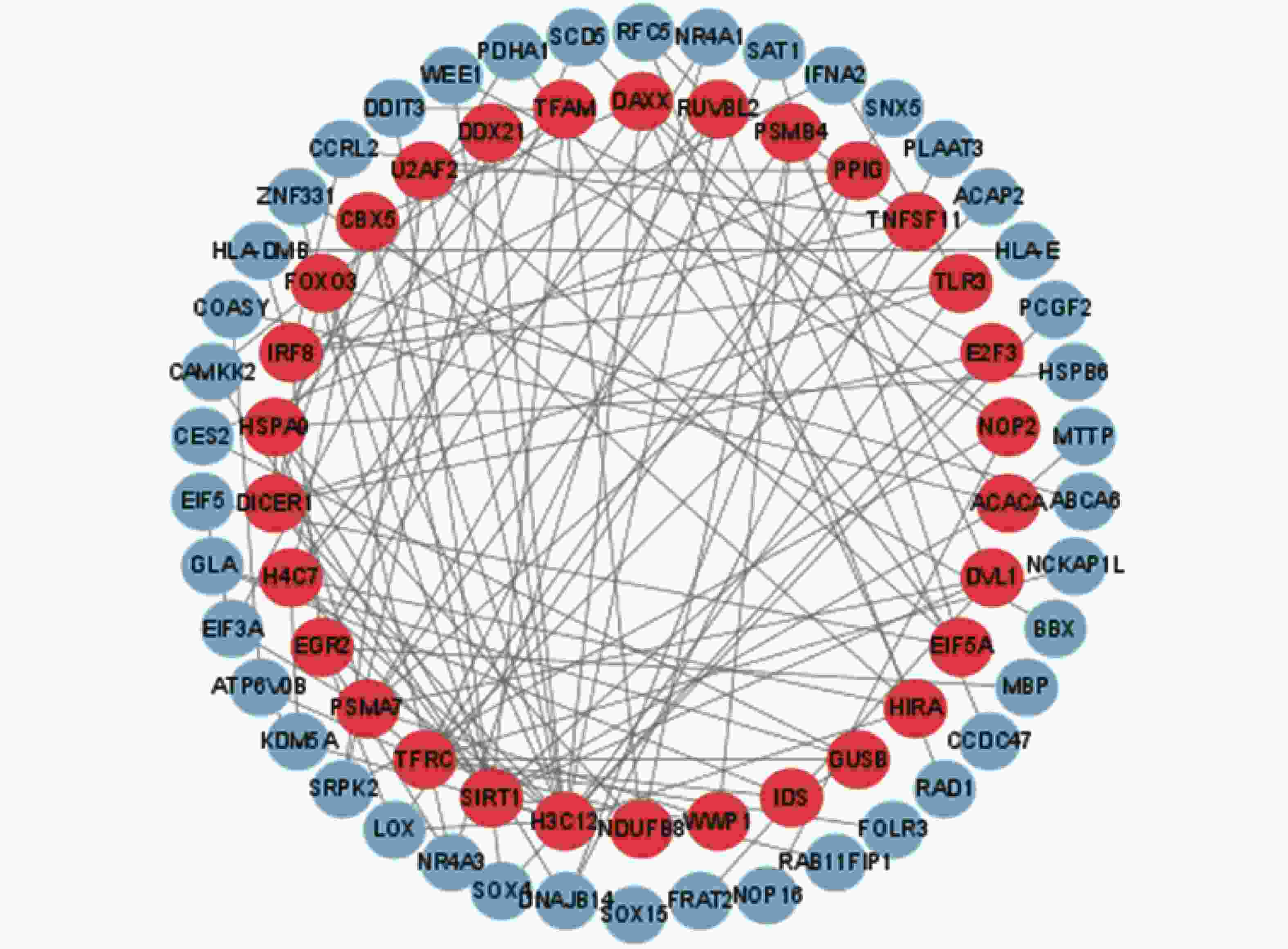
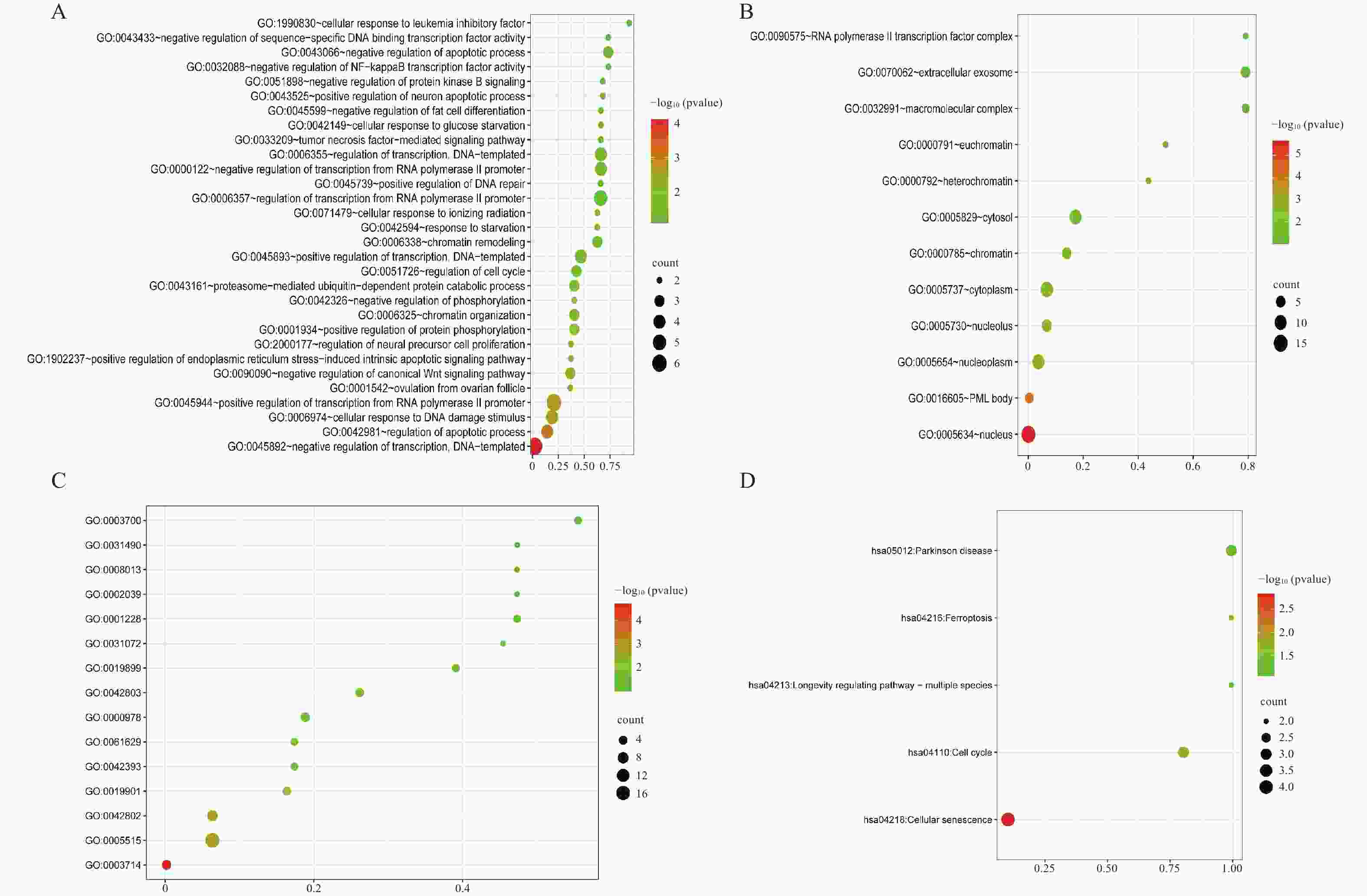
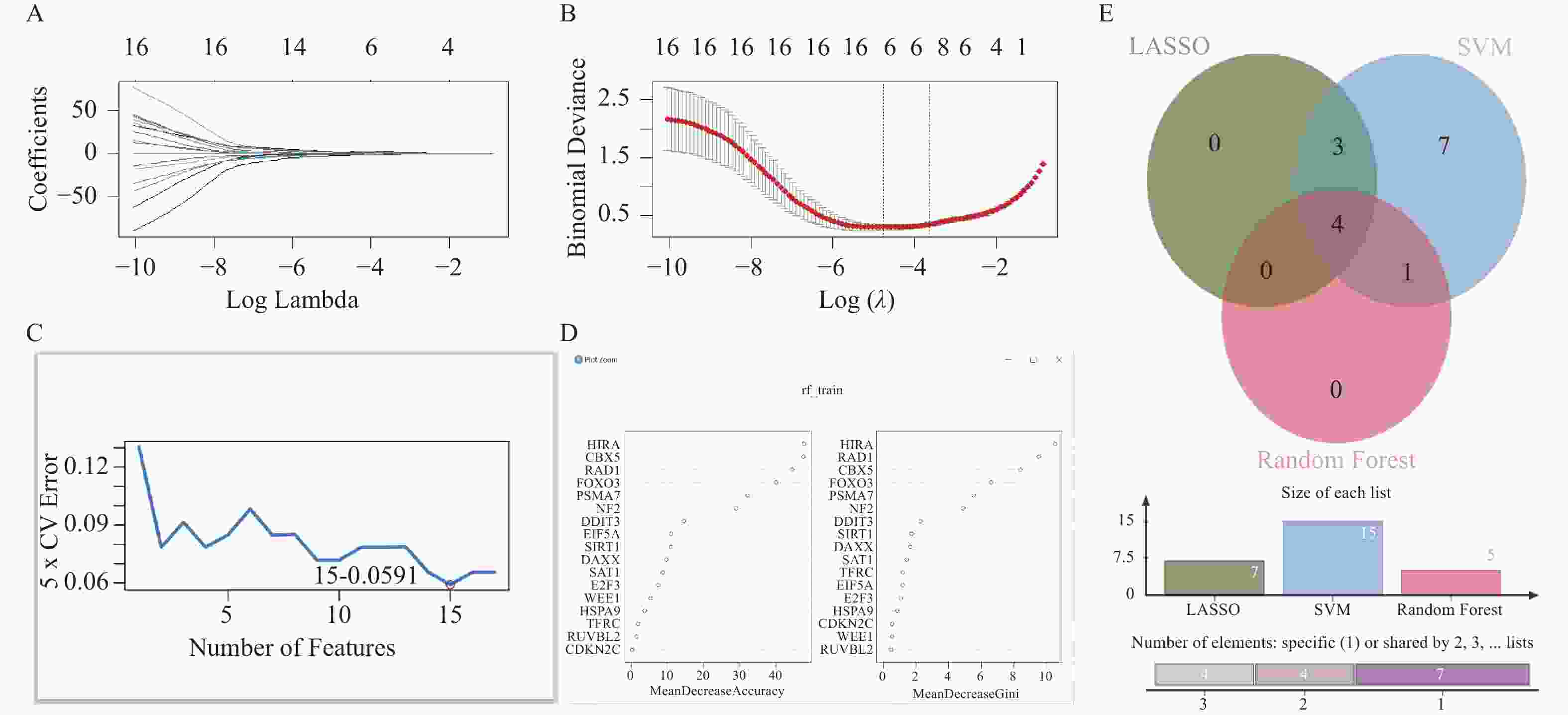
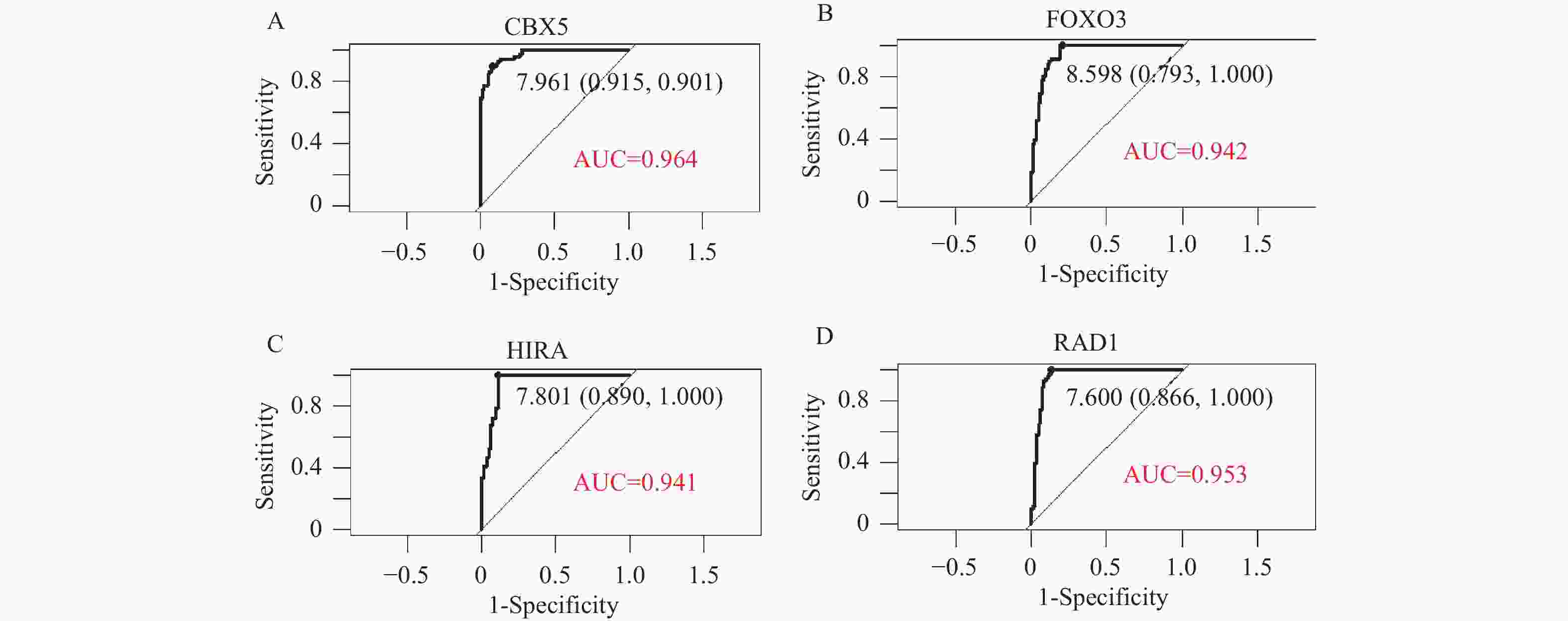
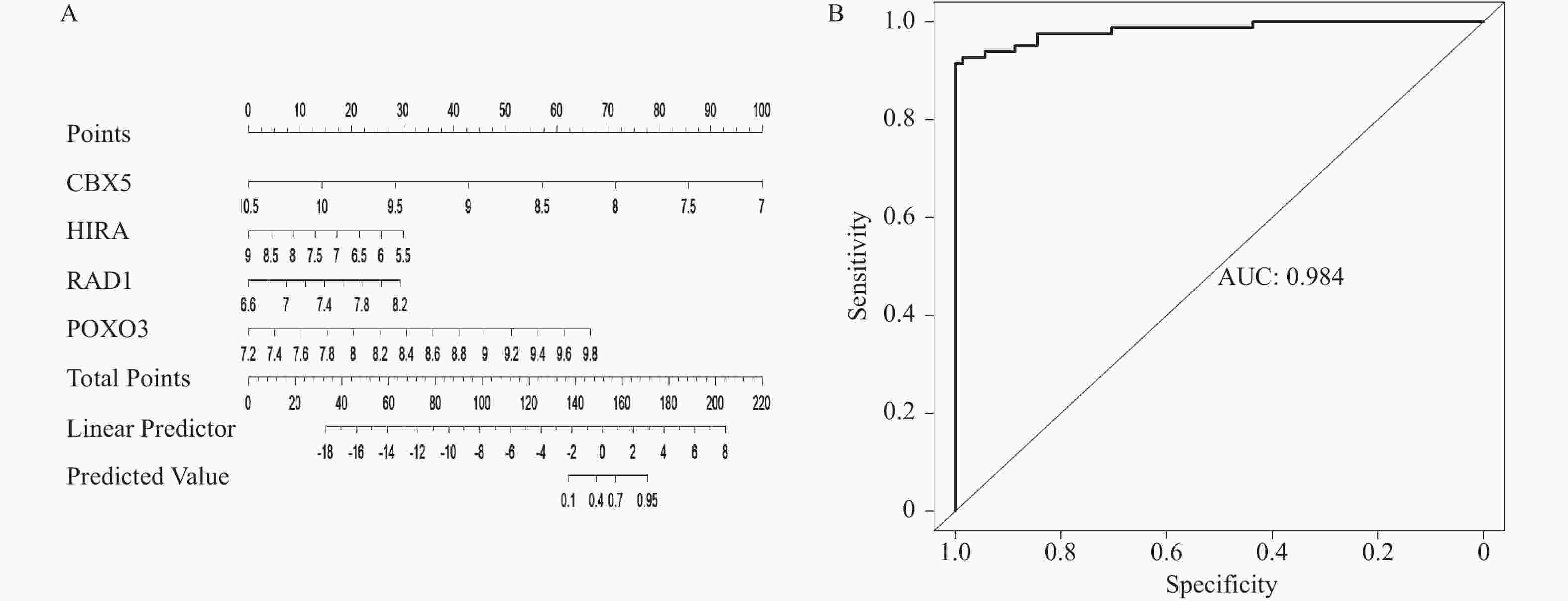
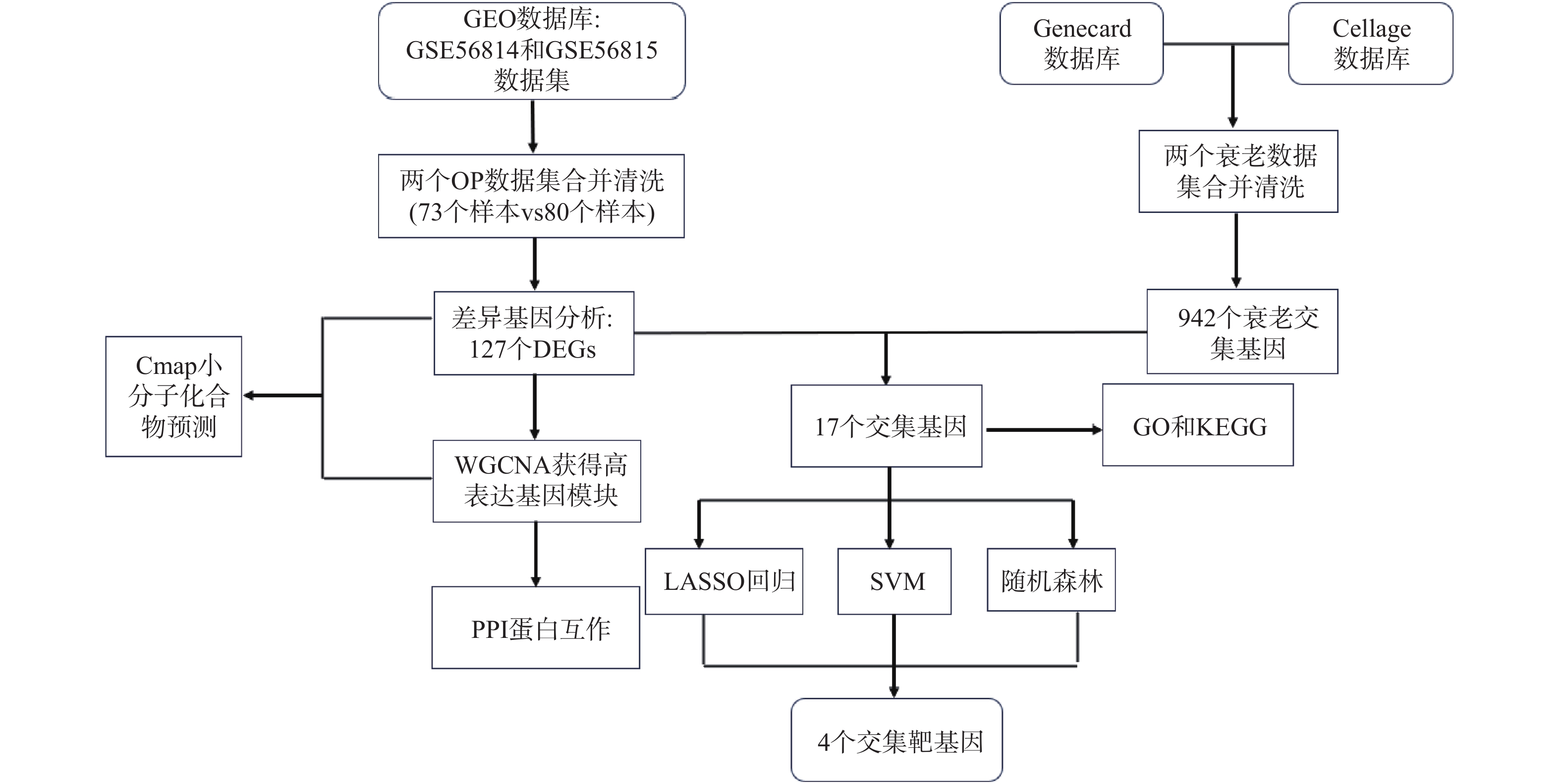
目的 利用公共数据库中的数据探索衰老与骨质疏松的共同靶基因,筛选有潜在治疗作用的小分子化合物。 方法 从公共基因表达数据库(gene expression omnibus,GEO)中下载骨质疏松数据集GSE56814和GSE56815并合并,从Genecard数据库和Cell age数据库中选取衰老基因并合并,使用R软件对数据进行清洗,筛选出差异基因,制作火山图,进行加权基因共表达筛选出关键模块和基因以及使用机器学习方法筛选靶基因,利用微生信平台绘制韦恩图,Cytoscape软件进行蛋白互作结果可视化,使用STRING 数据库进行差异编码蛋白的相互作用分析,利用Cmap数据库进行小分子化合物的预测。绘制受试者工作特征曲线,找出最有意义的靶基因,在此基础上建立逻辑回归模型并绘制列线图。 结果 使用limma包共筛选出127个差异基因。对差异基因进行加权基因共表达网络分析,筛选出103个高表达基因。采用拉索回归、支持向量机和随机森林模型3种机器学习方法,最终获得4个靶基因:FOXO3、HIRA、CBX5、RAD1。列线图和校准曲线显示出良好的预测效果。使用Cmap数据库筛选出有潜在治疗作用的5种小分子化合物:樱黄素(O-甲基化异黄酮)、XMD-885、杠柳次苷、依维莫司和XMD- 1150 。结论 FOXO3、HIRA基因目前已证实是衰老和骨质疏松共同的靶基因,樱黄素对衰老和骨质疏松有潜在治疗作用。 Abstract:Objective To explore common target genes related to aging and osteoporosis using data from public databases and to screen for small molecule compounds with potential therapeutic effects. Methods Osteoporosis datasets GSE56814 and GSE56815 were downloaded and merged from the public gene expression database (Gene Expression Omnibus (GEO)) . Senescent genes were selected and merged from the genecard database and the cell age database. Data cleaning and differential gene selection were performed using R software, and a volcano plot was created. Weighted gene co-expression network analysis was performed to screen out key modules and genes, and the machine learning methods were employed to screen target genes. A Venn diagram was drawn using the the Microbioinformatics platform, and cytoscape software was used to visualise protein interactions results. Interaction analysis of differentially encoded proteins was carried out using the STRING database, and prediction of small molecule compounds was carried out using Cmap database. Finally, Receiver operating characteristic (ROC) curves were plotted to find out the most meaningful target genes, followed by the establishment of a logistic regression model and the creation of a nomogram. Results A total of 127 differential genes were screened using the limma package. Weighted gene co-expression network analysis was performed on the differential genes and 103 highly expressed genes were screened. Three machine learning methods, lasso regression, support vector machine and random forest model, were used to finally obtain four target genes: FOXO3, HIRA, CBX5 and RAD1. The nomogram and calibration curves showed good predictive results. Five small molecule compounds with potential therapeutic effects were identified using the Cmap database: sakuraflavin (O-methylated isoflavone), XMD-885, kangaroo hyoscyamine, everolimus and XMD-1150. Conclusion FOXO3 and HIRA genes have now been confirmed to be the common target genes for aging and osteoporosis, and sakuraflavin has potential therapeutic effects on both conditions. -
Key words:
- Cellular senescence /
- Osteoporosis /
- Target genes /
- Small molecule compounds /
- Genomic analysis
-
表 1 CMap分析发现的潜在衰老和骨质疏松症药物小分子化合物
Table 1. Potential small molecular compounds for aging and osteoporosis found in CMap analysis
连通性评分 药物名称 说明 −98.39 prunetin Breast cancer resistance protein inhibitor −97.82 XMD-885 Leucine rich repeat kinase inhibitor −97.64 periplocymarin Apoptosis stimulant −96.95 everolimus MTOR inhibitor −96.37 XMD- 1150 Leucine rich repeat kinase inhibitor -
[1] Calimport S R G,Bentley B L,Stewart C E,et al. To help aging populations,classify organismal senescence[J]. Science,2019,366(6465):576-578. doi: 10.1126/science.aay7319 [2] 陈卫,郭亚隆. 中国的人口负增长与人口老龄化[J]. 北京社会科学,2023(8):101-112. [3] 杜鹏. 中国人口老龄化现状与社会保障体系发展[J]. 社会保障评论,2023,7(2):31-47. [4] Le Couteur D G,Thillainadesan J. What is an aging-related disease? An epidemiological perspective[J]. The Journals of Gerontology Series A: Biological Sciences and Medical Sciences,2022,77(11):2168-2174. doi: 10.1093/gerona/glac039 [5] Dönertaş H M,Fabian D K,Valenzuela M F,et al. Common genetic associations between age-related diseases[J]. Nat Aging,2021,1(4):400-412. doi: 10.1038/s43587-021-00051-5 [6] Fougère B,Boulanger E,Nourhashémi F,et al. Chronic inflammation: Accelerator of biological aging[J]. J Gerontol A Biol Sci Med Sci,2017,72(9):1218-1225. doi: 10.1093/gerona/glw240 [7] Filippov M A,Tatarnikova O G,Pozdnyakova N V,et al. Inflammation/bioenergetics-associated neurodegenerative pathologies and concomitant diseases: A role of mitochondria targeted catalase and xanthophylls[J]. Neural Regen Res,2021,16(2):223-233. doi: 10.4103/1673-5374.290878 [8] Wang Y,Shen J,Chen Y,et al. PINK1 protects against oxidative stress induced senescence of human nucleus pulposus cells via regulating mitophagy[J]. Biochem Biophys Res Commun,2018,504(2):406-414. doi: 10.1016/j.bbrc.2018.06.031 [9] Tian Y,Hu Y,Hou X,et al. Impacts and mechanisms of PM2.5 on bone[J]. Rev Environ Health,2023,39(4):765-775. [10] 王永炫,李梅,章振林,等. 《原发性骨质疏松症诊疗指南(2022)》要点解读[J]. 协和医学杂志,2023,14(6):1203-1207. [11] Almeida M,Laurent M R,Dubois V,et al. Estrogens and androgens in skeletal physiology and pathophysiology[J]. Physiol Rev,2017,97(1):135-187. doi: 10.1152/physrev.00033.2015 [12] Ahire JJ,Kumar V,Rohilla A. Understanding osteoporosis: Human bone density,genetic mechanisms,gut microbiota,and future prospects[J]. Probiotics Antimicrob Proteins,2024,16(3):875-883. doi: 10.1007/s12602-023-10185-0 [13] Zhou Z,Lu Y,Wang Y,et al. Let-7c regulates proliferation and osteodifferentiation of human adipose-derived mesenchymal stem cells under oxidative stress by targeting SCD-1[J]. Am J Physiol Cell Physiol,2019,316(1):C57-C69. doi: 10.1152/ajpcell.00211.2018 [14] Li H,Xiao Z,Quarles L D,et al. Osteoporosis: Mechanism,molecular target and current status on drug development[J]. Curr Med Chem,2021,28(8):1489-1507. doi: 10.2174/0929867327666200330142432 [15] Palacios S. Medical treatment of osteoporosis[J]. Climacteric,2022,25(1):43-49. doi: 10.1080/13697137.2021.1951697 [16] Yang T L,Shen H,Liu A,et al. A road map for understanding molecular and genetic determinants of osteoporosis[J]. Nat Rev Endocrinol,2020,16(2):91-103. doi: 10.1038/s41574-019-0282-7 [17] Farr J N,Rowsey J L,Eckhardt B A,et al. Independent roles of estrogen deficiency and cellular senescence in the pathogenesis of osteoporosis: Evidence in young adult mice and older humans[J]. J Bone Miner Res,2019,34(8):1407-1418. doi: 10.1002/jbmr.3729 [18] Shen G S,Zhou H B,Zhang H,et al. The GDF11-FTO-PPARγ axis controls the shift of osteoporotic MSC fate to adipocyte and inhibits bone formation during osteoporosis[J]. Biochim Biophys Acta Mol Basis Dis,2018,1864(12):3644-3654. doi: 10.1016/j.bbadis.2018.09.015 [19] Busse B,Djonic D,Milovanovic P,et al. Decrease in the osteocytelacunar density accompanied by hypermineralized lacunar occlusion reveals failure and delay of remodeling in aged human bone[J]. Aging Cell,2010,9(6):1065-1075. doi: 10.1111/j.1474-9726.2010.00633.x [20] Jilka R L,O'Brien C A. The role of osteocytes in age-related bone loss[J]. Curr Osteoporos Rep,2016,14(1):16-25. doi: 10.1007/s11914-016-0297-0 [21] Lv Y J,Yang Y,Sui B D,et al. Resveratrol counteracts bone loss via mitofilin-mediated osteogenic improvement of mesenchymal stem cells in senescence-accelerated mice[J]. Theranostics,2018,8(9):2387-2406. doi: 10.7150/thno.23620 [22] Tiede-Lewis L M,Dallas S L. Changes in the osteocyte lacunocanalicular network with aging[J]. Bone,2019,122:101-113. doi: 10.1016/j.bone.2019.01.025 [23] Aurora R,Veis D. Does aging activate T-cells to reduce bone mass and quality?[J]. Curr Osteoporos Rep,2022,20(5):326-333. doi: 10.1007/s11914-022-00745-8 [24] De Spiegeleer A,Beckwée D,Bautmans I,et al. Pharmacological interventions to improve muscle mass,muscle strength and physical performance in older people: An umbrella review of systematic reviews and meta-analyses[J]. Drugs Aging,2018,35(8):719-734. doi: 10.1007/s40266-018-0566-y [25] Choi H S. Why do we need proactive management for fracture prevention in long-term glucocorticoid users?[J]. Endocrinol Metab (Seoul),2020,35(3):549-551. doi: 10.3803/EnM.2020.308 [26] Cortet B,Lucas S,Legroux-Gerot I,et al. Bone disorders associated with diabetes mellitus and its treatments[J]. Joint Bone Spine,2019,86(3):315-320. doi: 10.1016/j.jbspin.2018.08.002 [27] Ashamalla M,Malik,W. S,et al. Investigating the factors that affect osteoporosis in an aging population[J]. The Health & Fitness Journal of Canada,2020,13(2):30-36. [28] Rubio-Gutierrez J C,Mendez-Hernández P,Guéguen Y,et al. Overview of traditional and environmental factors related to bone health[J]. Environ Sci Pollut Res Int,2022,29(21):31042-31058. doi: 10.1007/s11356-022-19024-1 [29] Javaheri B,Pitsillides A A. Aging and mechanoadaptive responsiveness of bone[J]. Curr Osteoporos Rep,2019,17(6):560-569. doi: 10.1007/s11914-019-00553-7 [30] Khor Y S,Wong P F. MicroRNAs-associated with FOXO3 in cellular senescence and other stress responses[J]. Biogerontology,2024,25(1):23-51. doi: 10.1007/s10522-023-10059-6 [31] Xia B,Li Y,Zhou J,et al. Identification of potential pathogenic genes associated with osteoporosis[J]. Bone Joint Res,2017,6(12):640-648. doi: 10.1302/2046-3758.612.BJR-2017-0102.R1 [32] Köksal Karayildirim Ç,Nalbantsoy A,Karabay Yavaşoğlu N Ü. Prunetin inhibits nitric oxide activity and induces apoptosis in urinary bladder cancer cells via CASP3 and TNF-α genes[J]. Mol Biol Rep,2021,48(11):7251-7259. doi: 10.1007/s11033-021-06719-w [33] Kim B,Jo C,Choi H Y,et al. Prunetin relaxed isolated rat aortic rings by blocking calcium channels[J]. Molecules,2018,23(9):2372. doi: 10.3390/molecules23092372 [34] Khan K,Pal S,Yadav M,et al. Prunetin signals via G-protein-coupled receptor,GPR30(GPER1): Stimulation of adenylyl cyclase and cAMP-mediated activation of MAPK signaling induces Runx2 expression in osteoblasts to promote bone regeneration[J]. J Nutr Biochem,2015,26(12):1491-501. doi: 10.1016/j.jnutbio.2015.07.021 [35] Wu J,Chen J,Yu X,et al. The potential pharmacological mechanism of prunetin against osteoporosis: transcriptome analysis,molecular docking,and experimental approaches[J]. Toxicol Mech Methods,2024,34(1):46-56. doi: 10.1080/15376516.2023.2253305 [36] Chandra A, Rajawat J. Skeletal aging and osteoporosis: Mechanisms and therapeutics[J]. Int J Mol Sci,2021,22(7):3553. doi: 10.3390/ijms22073553 [37] Pignolo R J,Law S F,Chandra A. Bone aging,cellular senescence,and osteoporosis[J]. JBMR Plus,2021,5(4):e10488. doi: 10.1002/jbm4.10488 [38] Manolagas S C. From estrogen-centric to aging and oxidative stress: A revised perspective of the pathogenesis of osteoporosis[J]. Endocr Rev,2010,31(3):266-300. doi: 10.1210/er.2009-0024 [39] Pietschmann P,Mechtcheriakova D,Meshcheryakova A,et al. Immunology of osteoporosis: A mini-review[J]. Gerontology,2016,62(2):128-137. doi: 10.1159/000431091 -






 下载:
下载: