Molecular Docking-Based Screening and Activity Studies of NDM-1 β-Lactamase Inhibitors against Klebsiella pneumoniae
-
摘要:
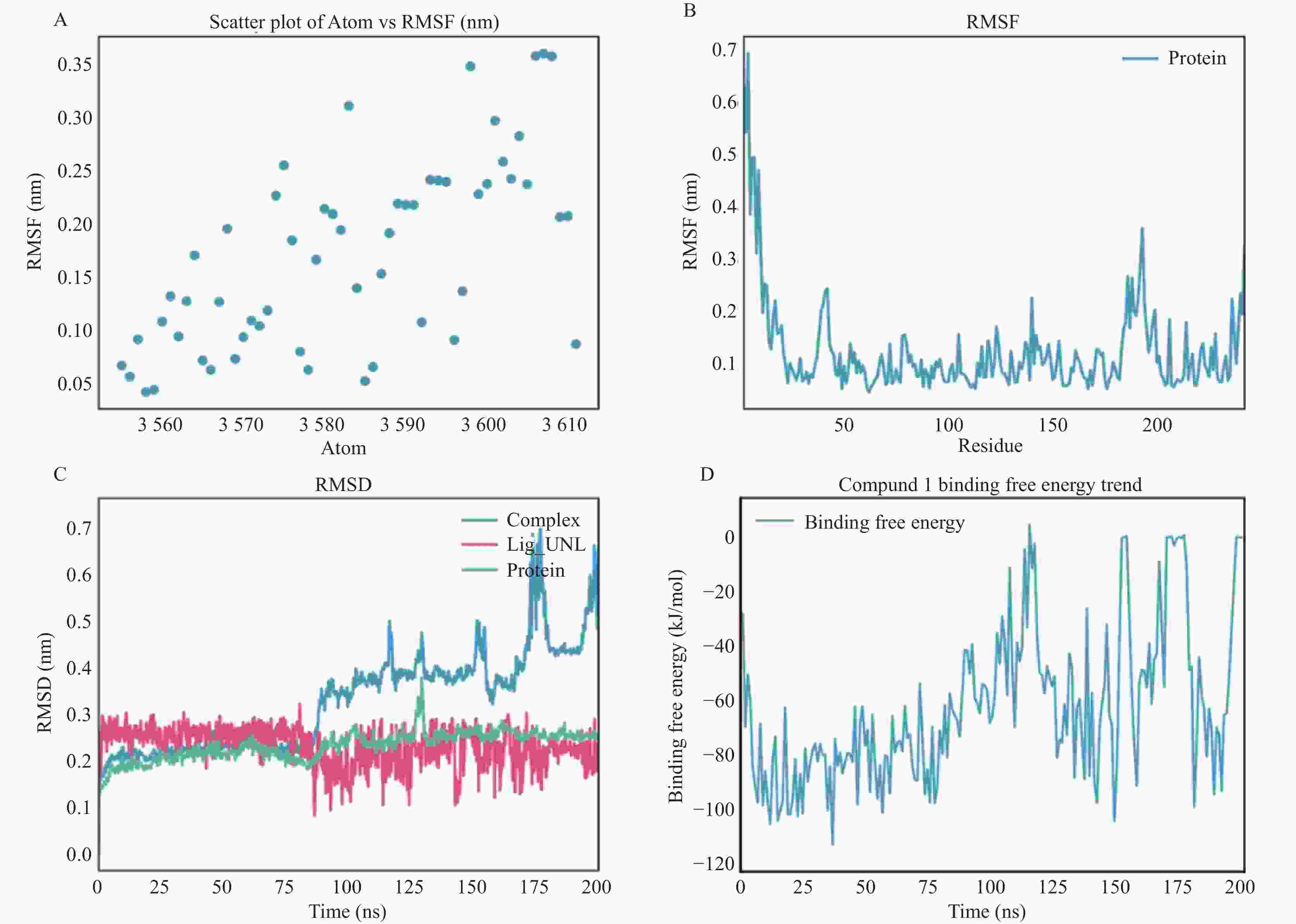
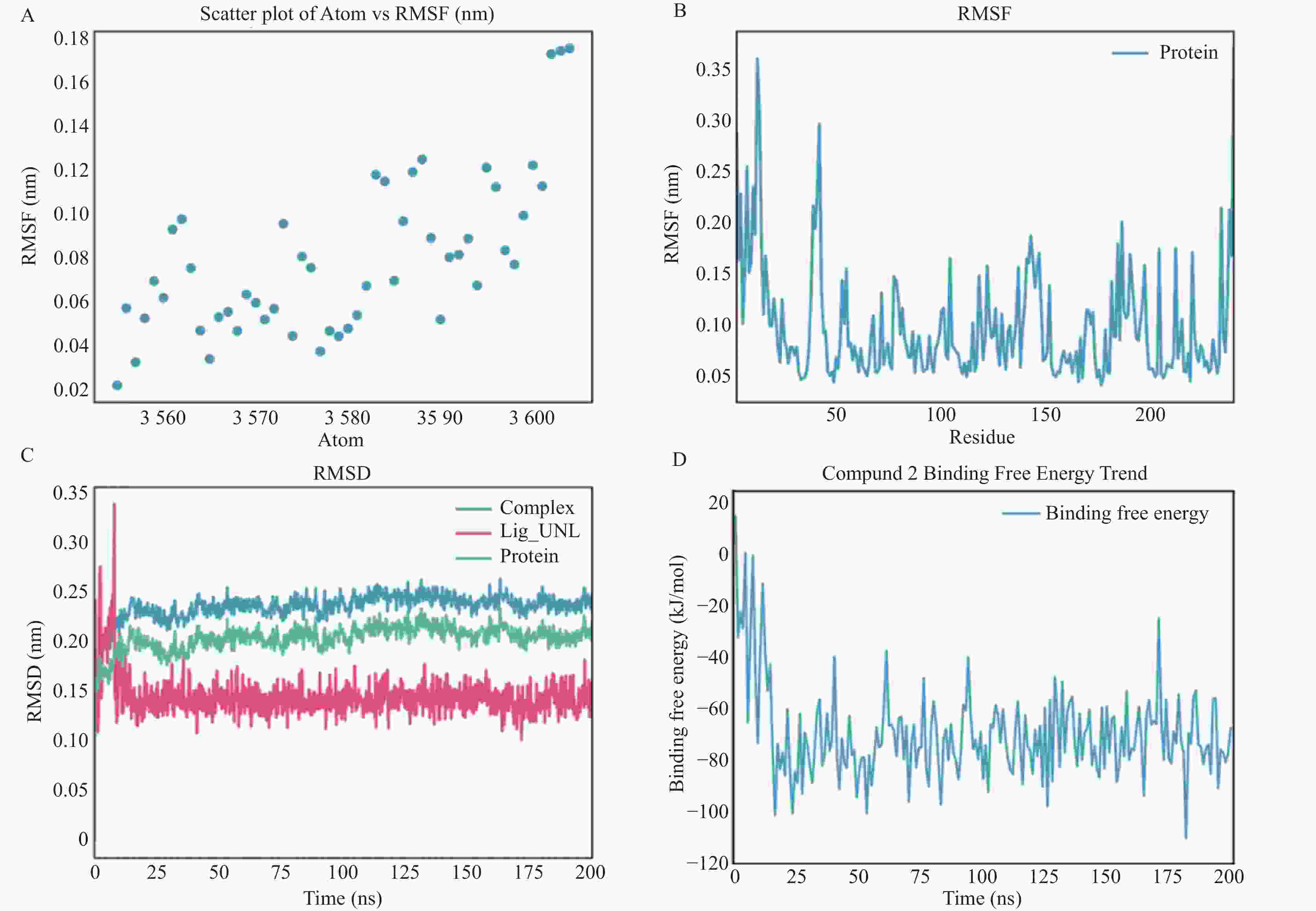
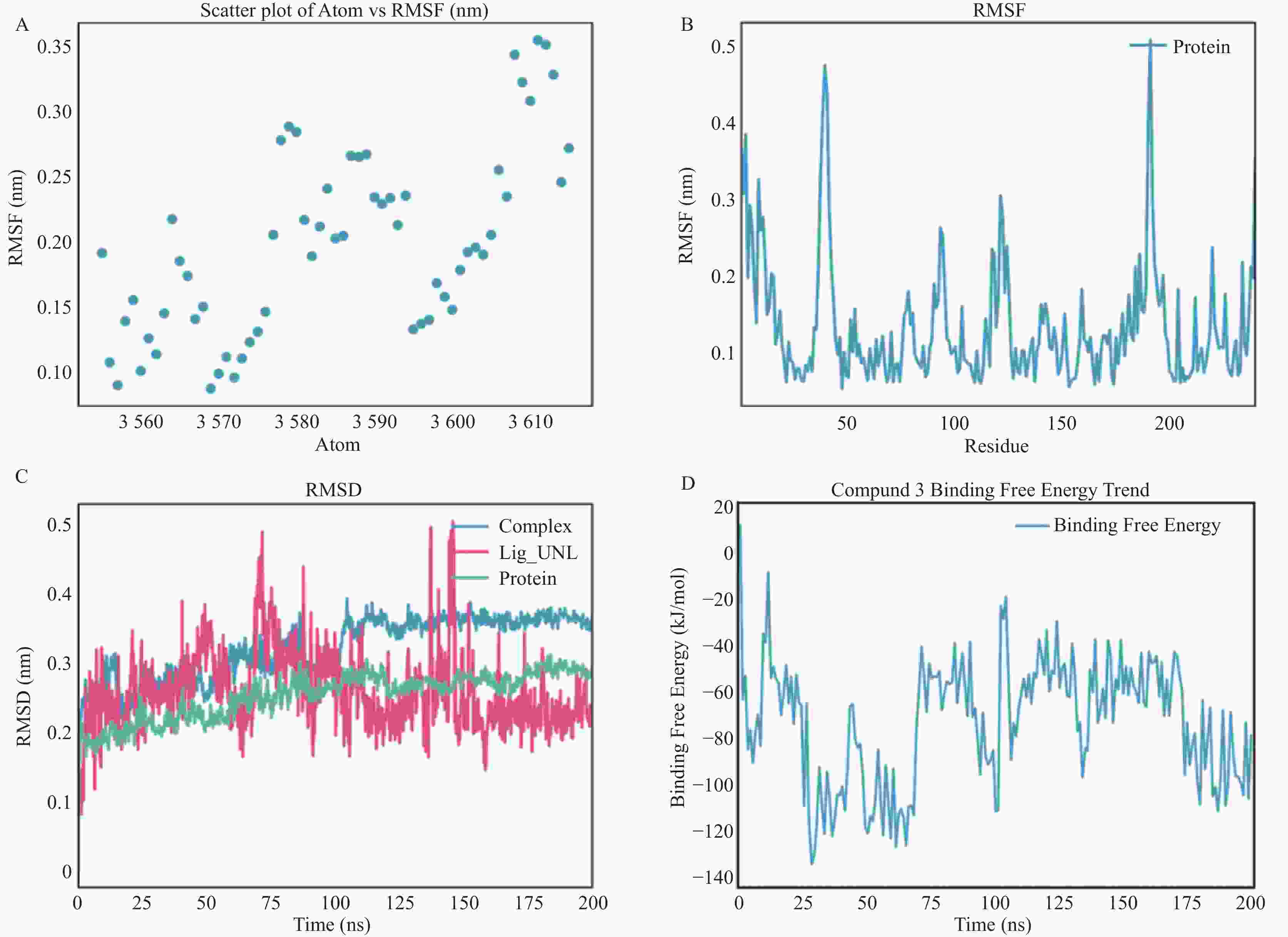
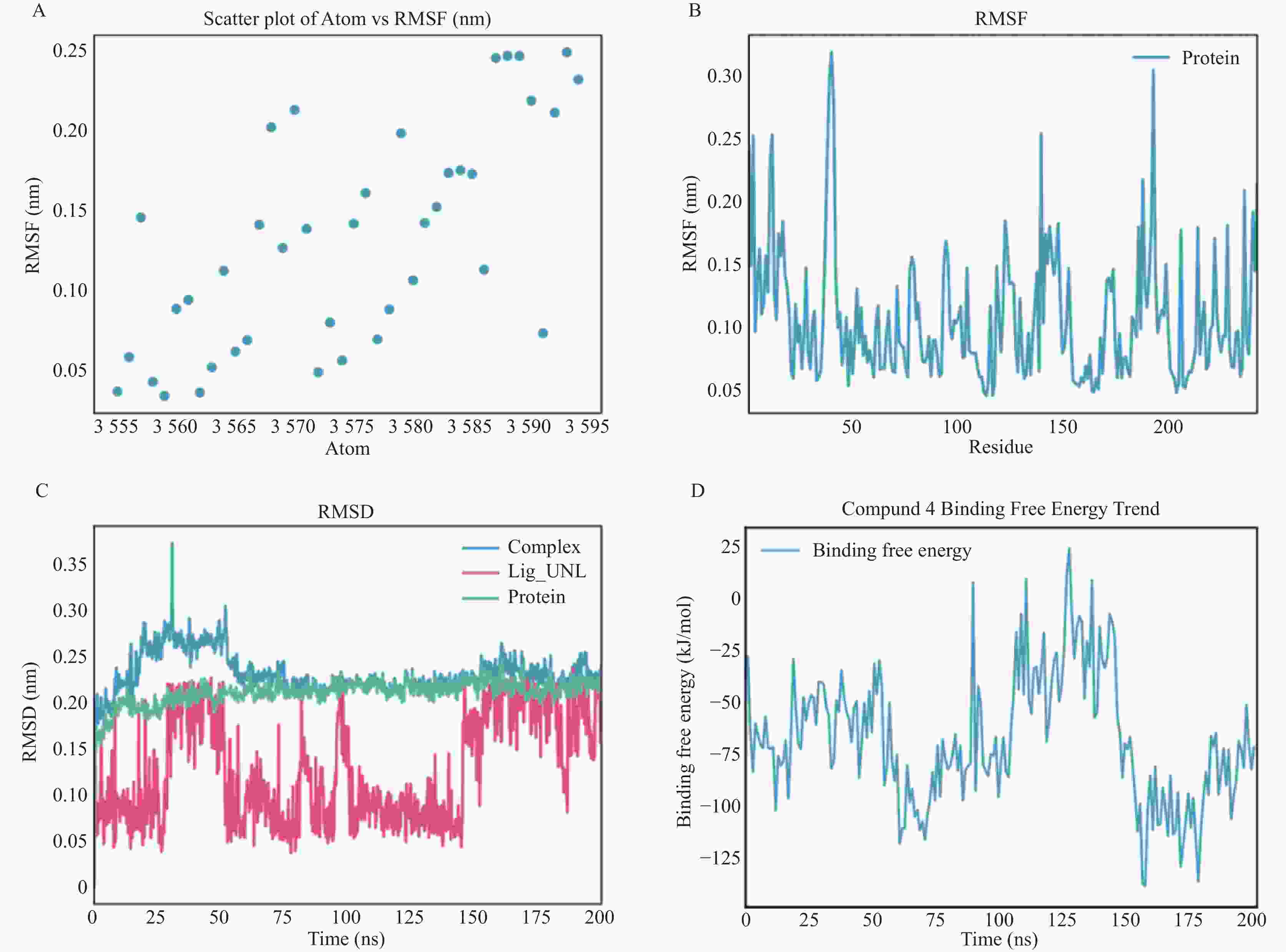
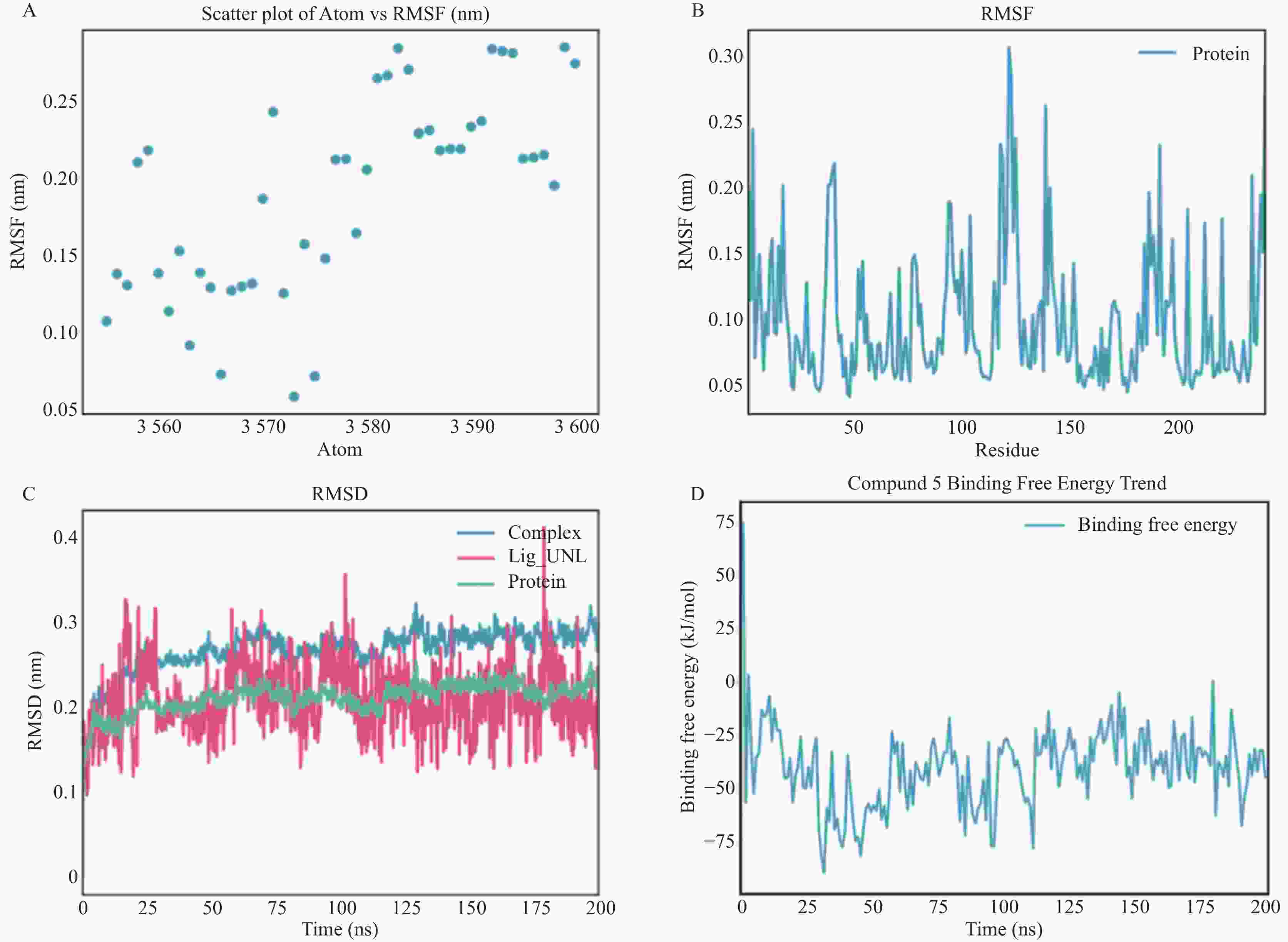
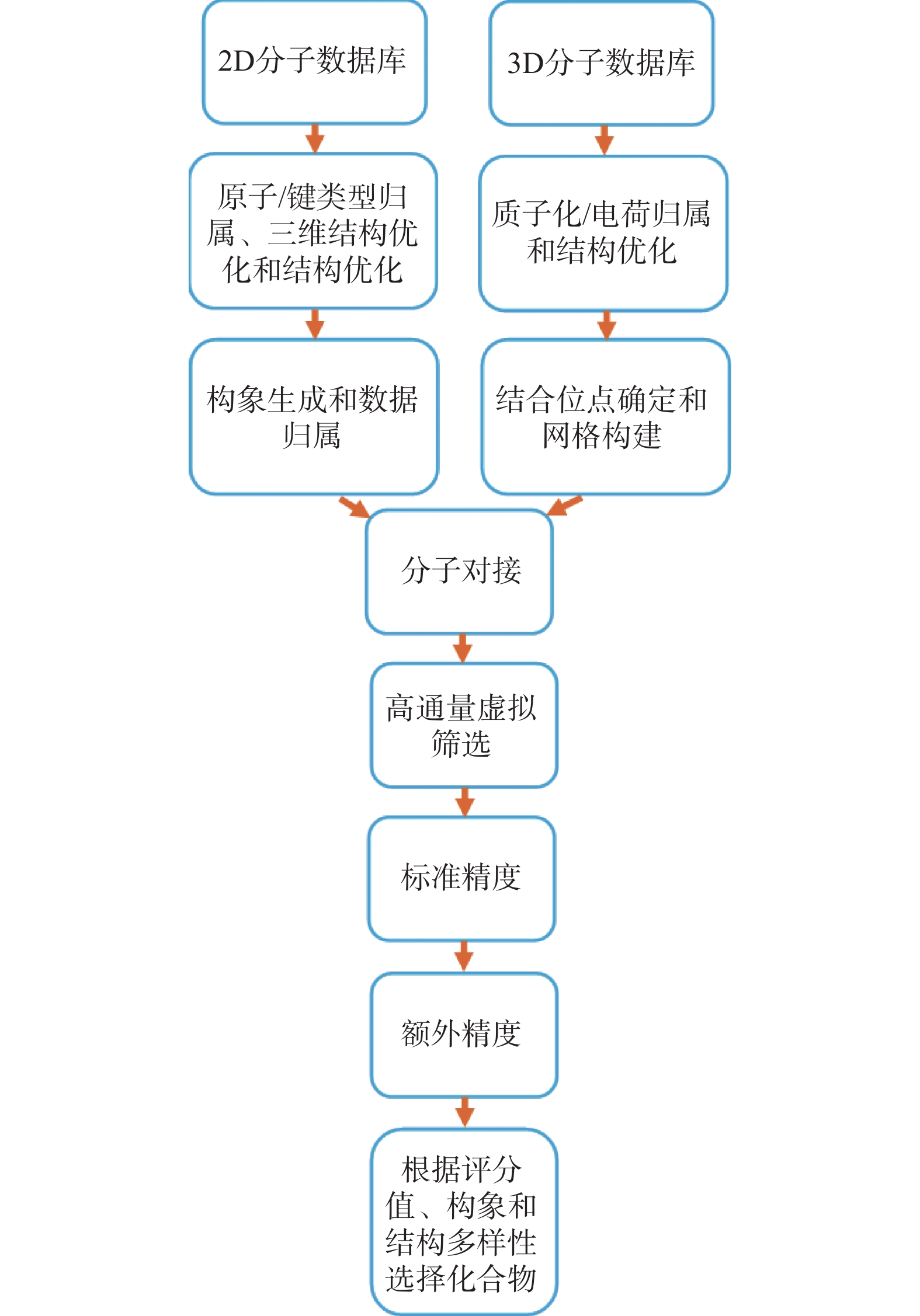
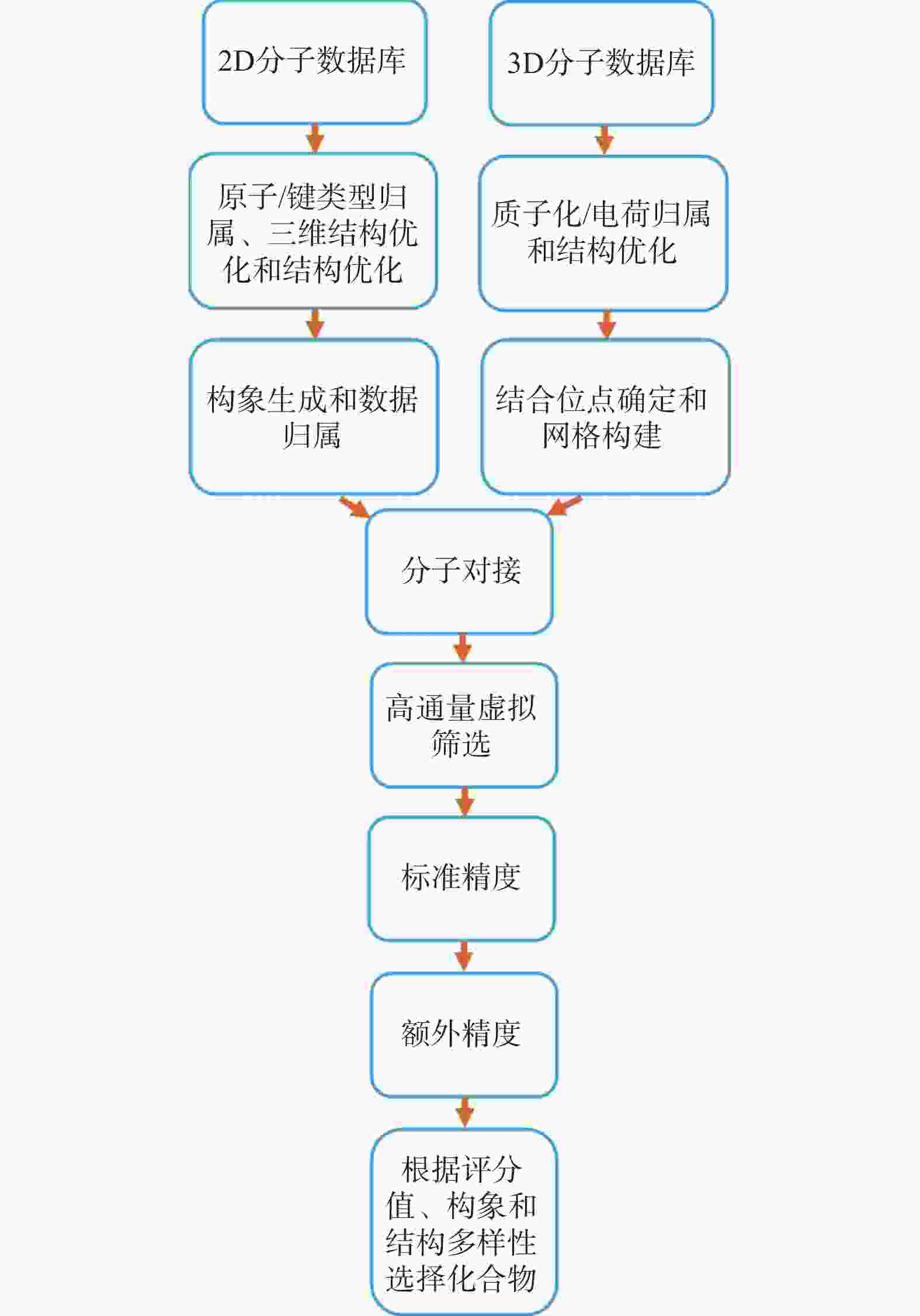
目的 基于NDM-1酶三维结构,筛选潜在抑制剂并进行药效评价。 方法 采用计算机辅助药物设计方法,从50K Diversity Library化合物库中筛选分子对接得分前5的候选化合物。进一步联合美罗培南、亚胺培南及头孢他啶进行最低抑菌浓度(minimum inhibitory concentration,MIC)检测,评估其抑制NDM-1的活性。并通过分子动力学模拟分析候选化合物与NDM-1结合的稳定性。 结果 分子对接得分前5的化合物与三种β-内酰胺类抗生素联合应用时,MIC值未见明显降低,提示其对NDM-1抑制效果有限。分子动力学模拟显示,化合物 1 、 3 、 4 、 5 在反应体系中均方根偏差值(root-mean-square deviation,RMSD)和自由能波动较大,表明其构象不稳定;化合物 2 虽无显著RMSD波动,但结合自由能值偏低,可能导致结合力不足。 结论 5个候选化合物与NDM-1蛋白的结合稳定性存在差异导致其抑制活性整体表现有限。 Abstract:Objective To screen potential inhibitors based on the three-dimensional structure of NDM-1 enzyme and evaluate their pharmacological activity. Methods Computer-aided drug design methods were employed to screen the top 5 candidate compounds with the highest molecular docking scores from the 50K Diversity Library. The minimum inhibitory concentration (MIC) was further determined in combination with meropenem, imipenem, and ceftazidime to assess their inhibitory activity against NDM-1. The stability of the candidate compounds binding to NDM-1 was analyzed through molecular dynamics simulation. Results When the top 5 compounds with the highest molecular docking scores were combined with three β-lactam antibiotics, the MIC values showed no significant reduction, suggesting limited inhibitory effects on NDM-1. Molecular dynamics simulation revealed that compounds 1, 3, 4, and 5 exhibited large root-mean-square deviation (RMSD) and free energy fluctuations in the reaction system, indicating unstable conformations; although compound 2 showed no significant RMSD fluctuation, its binding free energy value was relatively low, potentially resulting in insufficient binding affinity. Conclusion Differences in binding stability between the five candidate compounds and NDM-1 protein led to their overall limited inhibitory activity. -
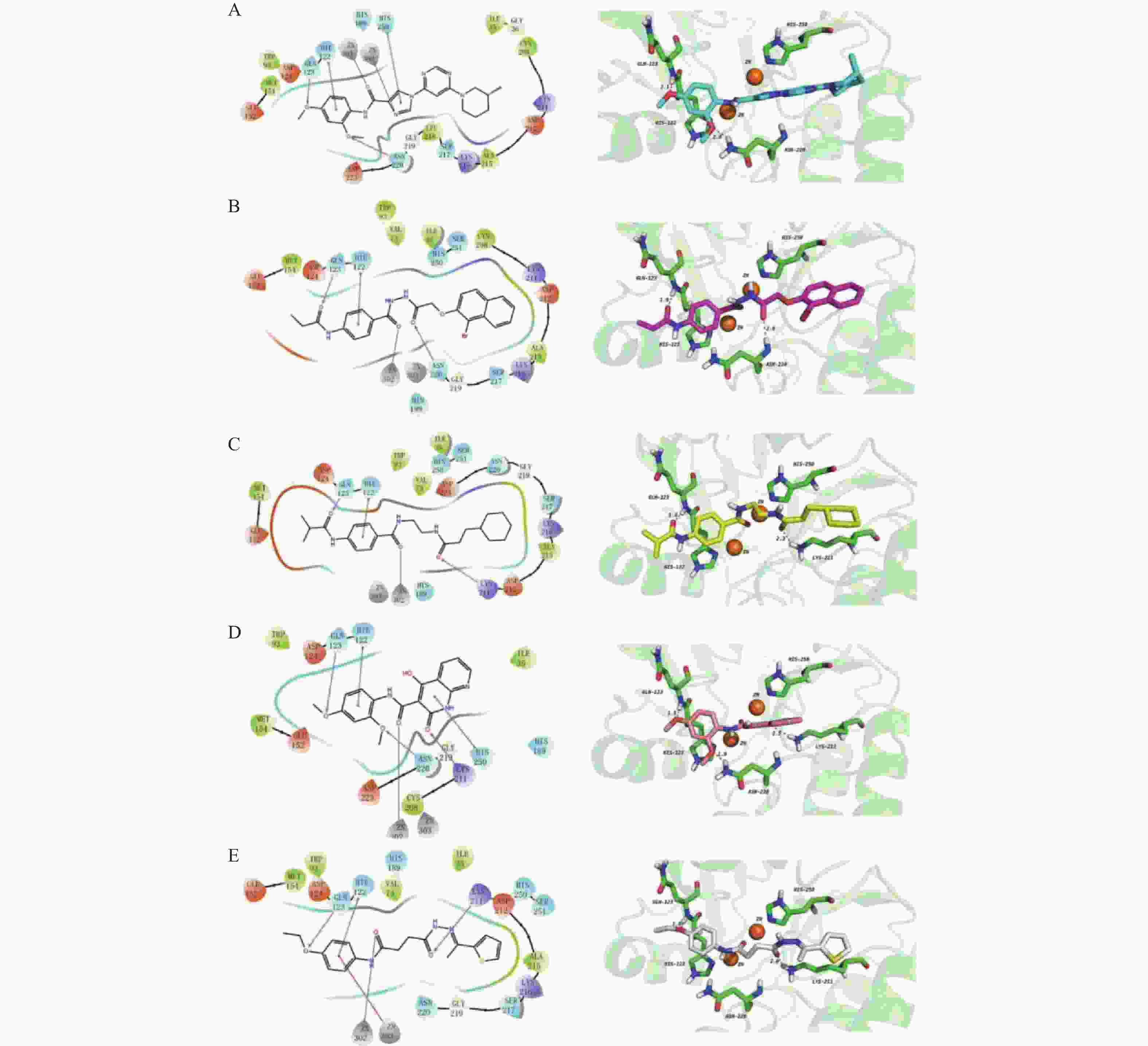
图 3 选定化合物与NDM-1蛋白结合模式的2D和3D图示
A: 化合物1和NDM-1蛋白结合模式的2D、3D作图;B: 化合物2和NDM-1蛋白结合模式的2D、3D作图;C: 化合物3和NDM-1蛋白结合模式的2D、3D作图;D: 化合物4和NDM-1蛋白结合模式的2D、3D作图;E: 化合物5和NDM-1蛋白结合模式的2D、3D作图.3D图中NDM-1蛋白绿色代表C骨架,蓝色代表N原子,大红色代表O原子,白色代表H原子,橘色代表锌离子,氢键-红色虚线,键长越长,氢键作用越弱。5个化合物中的stick依次为淡蓝色、玫红色、黄色、豆沙色、灰白色。
Figure 3. 2D and 3D diagrams of the binding mode of selected compounds with NDM-1 Protein
表 1 抗菌药物敏感性试验结果
Table 1. Antimicrobial susceptibility test results
抗生素名称 MIC(μg/mL) 敏感度 氨曲南(AZM) ≥64.0 R 哌拉西林/他唑巴坦(TZP) ≥128.0 R 替卡西林/克拉维酸(TCC) ≥128.0 R 头孢吡肟(FEP) ≥32.0 R 头孢他啶(TAZ) ≥64.0 R 亚胺培南(IMI) ≥16.0 R 美洛培南(MEM) ≥16.0 R 妥布霉素(TOB) ≦1.0 S 阿米卡星(AN) ≦2.0 S 米诺环素(MN) 4.0 S 环丙沙星(CIP) 1.0 R 左氧氟沙星(LEV) 1.0 I 复方新诺明(SXT) ≥320.0 R 粘菌素(CS) ≦0.5 S 替加环素(TGC) 2.0 S 多西环素(DX) ≥16.0 R 氨苄西林/舒巴坦(AMS) 6 R 头孢曲松(CTR) 6 R 头孢呋辛(ROXH) 6 R 头孢他啶/阿维巴坦(CZA) 17 R 头孢哌酮/舒巴坦(SFP) ≥64.0 R 耐药(R);敏感(S);中介(I)。 表 2 按分子对接评分排名的前5种化合物
Table 2. Top 5 compounds ranked by molecular docking scores
排序 结构 名称 分子对接得分 1 
HY-Q09607 −8.322 2 
HY-Q43565 −8.311 3 
HY-Q26669 −8.273 4 
HY-Q10558 −8.238 5 
HY-Q42293 −8.192 表 3 棋盘试验中药物组合的浓度与布局
Table 3. Concentration and layout of drug combinations in the checkerboard assay
μg/mL 1 2 3 4 5 6 7 8 1 A(128)+B(64) A(64)+B(64) A(32)+B(64) A(16)+B(64) A(8)+B(64) A(4)+B(64) A(2)+B(64) B(64) 2 A(128)+B(32) A(64)+B(32) A(32)+B(32) A(16)+B(32) A(8)+B(32) A(4)+B(32) A(2)+B(32) B(32) 3 A(128)+B(16) A(64)+B(16) A(32)+B(16) A(16)+B(16) A(8)+B(16) A(4)+B(16) A(2)+B(16) B(16) 4 A(128)+B(8) A(64)+B(8) A(32)+B(8) A(16)+B(8) A(8)+B(8) A(4)+B(8) A(2)+B(8) B(8) 5 A(128)+B(4) A(64)+B(4) A(32)+B(4) A(16)+B(4) A(8)+B(4) A(4)+B(4) A(2)+B(4) B(4) 6 A(128)+B(2) A(64)+B(2) A(32)+B(2) A(16)+B(2) A(8)+B(2) A(4)+B(2) A(2)+B(2) B(2) 7 A(128)+B(1) A(64)+B(1) A(32)+B(1) A(16)+B(1) A(8)+B(1) A(4)+B(1) A(2)+B(1) B(1) 8 A(128) A(64) A(32) A(16) A(8) A(4) A(2) 生长对照 A:抗生素;B:酶抑制剂。 表 4 美罗培南与化合物1-5联合对抗Kpn-4
Table 4. Combination of MEM and Compound 1-5 against Kpn-4
化合物 单药MIC(μg/mL) 联用 MIC(μg/mL) FICI 相互作用
类型美罗培南 化合物 美罗培南 化合物 1 64 >64 128 32 2.5 无关 2 64 >64 128 64 3 无关 3 64 >64 128 16 2.25 无关 4 64 >64 128 32 2.5 无关 5 64 >64 128 32 2.5 无关 表 5 亚胺培南与化合物1-5联合对抗Kpn-4
Table 5. Combination of IMI and Compound 1-5 against Kpn-4
化合物 单药MIC(μg/mL) 联用MIC(μg/mL) FICI 相互作用
类型亚胺培南 化合物 亚胺培南 化合物 1 32 >64 32 1 1.016 无关 2 32 >64 32 1 1.016 无关 3 32 >64 32 1 1.016 无关 4 32 >64 32 1 1.016 无关 5 32 >64 32 1 1.016 无关 表 6 头孢他啶/阿维巴坦与化合物1-5联合对抗Kpn-4
Table 6. Combination of CAZ and Compound 1-5 against Kpn-4
化合物 单药MIC(μg/mL) 联用 MIC(μg/mL) FICI 相互作用类型 头孢他啶/
阿维巴坦化合物 头孢他啶/
阿维巴坦化合物 1 >128 >64 >128 >64 >2 无关 2 >128 >64 >128 >64 >2 无关 3 >128 >64 >128 >64 >2 无关 4 >128 >64 >128 >64 >2 无关 5 >128 >64 >128 >64 >2 无关 -
[1] Borchers A, Pieler T. Programming pluripotent precursor cells derived from Xenopus embryos to generate specific tissues and organs[J]. Genes, 2010, 1(3): 413-426. doi: 10.3390/genes1030413 [2] Krco S, Davis S J, Joshi P, et al. Structure, function, and evolution of metallo-β-lactamases from the B3 subgroup-emerging targets to combat antibiotic resistance[J]. Front Chem, 2023, 11: 1196073. doi: 10.3389/fchem.2023.1196073 [3] 金文彬, 李江, 李娇, 等. 超级细菌NDM-1及其抑制剂的研究进展[J]. 药学学报, 2020, 55(6): 1157-1165. doi: 10.16438/j.0513-4870.2019-1049 [4] Philippon A, Jacquier H, Ruppé E, et al. Structure-based classification of class A beta-lactamases, an update[J]. Curr Res Transl Med, 2019, 67(4): 115-122. doi: 10.1016/j.retram.2019.05.003 [5] Tang B, Yang A, Liu P, et al. Outer membrane vesicles transmitting bla (NDM-1) mediate the emergence of carbapenem-resistant hypervirulent Klebsiella pneumoniae[J]. Antimicrob Agents Chemother, 2023, 67(5): e01444-e01422. doi: 10.1128/aac.01444-22 [6] Martínez M M B, Bonomo R A, Vila A J, et al. On the offensive: The role of outer membrane vesicles in the successful dissemination of new Delhi metallo-β-lactamase (NDM-1)[J]. mBio, 2021, 12(5): e01836-e01821. [7] Li L, Zhang Y, Weng L, et al. NDM-5-carried outer membrane vesicles impair the efficacy of antibiotics against bacterial infections[J]. Antimicrob Agents Chemother, 2025, 69(5): e01805-e01824. [8] Wang T, Xu K, Zhao L, et al. Recent research and development of NDM-1 inhibitors[J]. Eur J Med Chem, 2021, 223: 113667. doi: 10.1016/j.ejmech.2021.113667 [9] Linciano P, Cendron L, Gianquinto E, et al. Ten years with New Delhi metallo-β-lactamase-1 (NDM-1): from structural insights to inhibitor design[J]. ACS Infect Dis, 2019, 5(1): 9-34. [10] Newman D J, Cragg G M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019[J]. J Nat Prod, 2020, 83(3): 770-803. doi: 10.1021/acs.jnatprod.9b01285 [11] Jorgensen W L. The many roles of computation in drug discovery[J]. Science, 2004, 303(5665): 1813-1818. doi: 10.1126/science.1096361 [12] 郭宗儒. CADD和AIDD的药物化学刍议[J]. 药学学报, 2023, 58(10): 2931-2941. doi: 10.16438/j.0513-4870.2023-0702 [13] Cerqueira N M F S A, Gesto D, Oliveira E F, et al. Receptor-based virtual screening protocol for drug discovery[J]. Arch Biochem Biophys, 2015, 582: 56-67. doi: 10.1016/j.abb.2015.05.011 [14] 刘伟, 刘艳红, 倪萍, 等. ALK抑制剂HG-14-10-04的抗真菌活性及其作用机制研究[J]. 药学学报, 2024, 59(12): 3282-3290. doi: 10.16438/j.0513-4870.2024-0695 [15] Guo Y, Wang J, Niu G, et al. A structural view of the antibiotic degradation enzyme NDM-1 from a superbug[J]. Protein Cell, 2011, 2(5): 384-394. doi: 10.1007/s13238-011-1055-9 [16] Zhang H, Hao Q. Crystal structure of NDM-1 reveals a common β-lactam hydrolysis mechanism[J]. FASEB J, 2011, 25(8): 2574-2582. doi: 10.1096/fj.11-184036 [17] Daiyasu H, Osaka K, Ishino Y, et al. Expansion of the zinc metallo-hydrolase family of the β-lactamase fold[J]. FEBS Lett, 2001, 503(1): 1-6. [18] 余俊霖, 李国菠. 计算机辅助金属酶靶向药物发现的研究进展[J]. 中国现代应用药学, 2022, 39(21): 2828-2833. doi: 10.13748/j.cnki.issn1007-7693.2022.21.019 [19] King D T, Worrall L J, Gruninger R, et al. New Delhi metallo-β-lactamase: Structural insights into β-lactam recognition and inhibition[J]. J Am Chem Soc, 2012, 134(28): 11362-11365. doi: 10.1021/ja303579d [20] Newman H, Krajnc A, Bellini D, et al. High-throughput crystallography reveals boron-containing inhibitors of a penicillin-binding protein with di-and tricovalent binding modes[J]. J Med Chem, 2021, 64(15): 11379-11394. doi: 10.1021/acs.jmedchem.1c00717 [21] Li T, Wang Q, Chen F, et al. Biochemical characteristics of New Delhi metallo-β-lactamase-1 show unexpected difference to other MBLs[J]. PLoS One, 2013, 8(4): e61914. doi: 10.1371/journal.pone.0061914 -





 下载:
下载: