Advances in Epigenetic Regulatory Mechanisms in HSV1 Infection
-
摘要: 1型单纯疱疹病毒(HSV1)是一类最为常见的人类传染病原体,感染后可导致一系列程度不同的疾病。HSV1在中枢神经系统的潜伏感染及偶发重激活是其病理发生的关键,也为抗病毒治疗带来了巨大的挑战。目前,关于HSV1感染的建立、维持和重激活的机制并未完全阐明,但普遍认为表观遗传调控可能在其中扮演重要作用。越来越多研究表明,病毒裂解期和潜伏感染期的基因组呈现不同的染色质结构,其富含的多种翻译后修饰组蛋白赋予病毒基因转录激活或抑制特征。此外,病毒潜伏相关转录本LATs也可能参与基因组表观遗传修饰调控。Abstract: Herpes simplex viruses type 1(HSV1) is among the most ubiquitous human pathogens that cause a wide variety of disease states. The latent infection of the central nervous system and sporadically reactivation is the central part of HSV1 pathogenesis, which also brings challenges to antiviral therapies. At present, the mechanism of establishing, maintaining and reactivation of HSV1 has not been fully clarified, whereas it has been generally accepted that the epigenetic regulation may play an important role. Accumulating researches have also indicated that the lytic and latent viral genomes exhibit the different chromatin structures, and the accumulation of diverse post-translational modifies the histones endow viral genes with transcriptional activation or repression features. In addition, the latency-associate transcripts of virus may also participate in the genome epigenetic modification. In this review, we summarize the research progress of epigenetic regulation of HSV1 and highlight the critical role of chromatin remodeling in HSV1 lytic proliferation and establishment of latent infection.
-
I型单纯疱疹病毒(herpes simplex virus 1,HSV1)是一种分布广泛的双链DNA病毒,属于疱疹病毒科[1],编码80多个基因,具有高度嗜神经特性,感染一般发生在儿童时期,初始感染部位是黏膜上皮细胞,病毒在其内复制增殖,引起唇疱疹。同时HSV1感染能够引发一系列不同程度的临床病理表现,从轻微的口腔和唇疱疹、角膜感染再到危及生命的病毒性脑炎[1]。
1. HSV1生物学特性及感染疾病
与HIV和乙肝病毒一样,HSV1可以在人体内建立终身潜伏感染,病毒双链DNA持续存在于受感染细胞的细胞核中。其中HIV是一种整合病毒,而疱疹病毒和肝炎病毒不能将自身的双链DNA整合于宿主细胞基因组上,只能以宿主细胞染色体外游离的方式存在[2]。
HSV1的感染一般发生在儿童时期,初始感染部位是黏膜上皮细胞,病毒在其内复制增殖,引起唇疱疹;随后病毒沿神经轴突逆行至三叉神经节(trigeminal ganglia,TG)或进入感觉神经元的神经末梢。在三叉神经节,病毒虽有短暂复制,但大多数病毒最终进入潜伏状态。潜伏期间,病毒DNA复制在很大程度上受到抑制,病毒蛋白也处于极低水平表达,从而逃避宿主免疫监视。HSV1感染能够引发一系列不同程度的临床病理表现,从轻微的口腔和唇疱疹、角膜感染再到危及生命的病毒性脑炎[1]。在免疫功能正常的个体中,HSV1的原发和复发性感染通常是有限的,病毒能被机体免疫系统及时清除;但在免疫功能低下的个体中,更高的复发率往往导致严重的疾病表现。流行病学研究显示,HSV1病毒已是发达国家感染性失明和急性病毒性脑炎的主要原因[3]。
2. HSV1病毒感染机制
2.1 HSV1的裂解感染
裂解感染期,病毒基因组的转录受到严格的时空调控:首先是即早期(immediate early,IE)基因的转录。病毒与靶细胞膜融合后,包括病毒反式激活蛋白VP16在内的一些包膜蛋白会独立于衣壳进入细胞核,VP16与细胞八聚体DNA结合蛋白Oct-1反式作用,结合到IE基因启动子,诱导RNA聚合酶II依赖的IE基因转录[4]。IE基因产物在病毒感染后的2~4 h表达,包括ICP0,ICP4,ICP22,ICP27,ICP47[5],它们参与调控E基因的转录。E基因参与病毒基因组的复制,编码DNA复制所需要的蛋白质,包括7个直接参与病毒DNA复制的蛋白质和许多与核酸有关的蛋白质。E基因表达后,病毒基因组才开始复制[2]。 L基因的转录只有在病毒基因组复制后才被激活,一旦有足够的病毒基因拷贝产生,L基因的产物就会促进DNA的衣壳化[6]。激活L基因的表达需要合成至少3种病毒蛋白:ICP4、ICP27和ICP8[5]。
2.2 HSV1潜伏感染
HSV1在神经元潜伏时,裂解相关基因被沉默,病毒基因组的转录仅局限于一个非编码RNA家族,即潜伏相关转录本LATs[7-8]。初始LAT转录本为1个8.3 Kb的聚腺苷化RNA [9],通过进一步剪接生成一个2.0 Kb的稳定内含子,在神经元中大量积累。此外,在某些神经元中,该2.0 Kb内含子经选择性剪接产生1.5 Kb的内含子[10]。
目前,LATs转录本的功能尚不清楚,多个研究显示其可能通过抑制病毒裂解相关基因表达及抑制宿主细胞凋亡以维持病毒潜伏状态。例如LATs 编码的miR-H2-3p 和miR-H6 分别靶向病毒ICP0 和ICP4,协同神经元特异miR-138 抑制关键病毒基因转录,从而促进病毒潜伏或维持其潜伏状态[11-13],LAT 相关miR-H2 突变能通过上调ICP0 增强病毒神经毒力和重激活水平[14],并且,体外转染LAT表达质粒及LAT突变株动物模型显示LATs 能够抑制Caspase凋亡级联反应,抑制pAKT 去磷酸化水平促进感染细胞存活[15]。 然而,一些研究也显示,LAT缺失的重组病毒仍可以在三叉神经节有效建立潜伏感染[16- 17]。 近期研究发现,人皮肤组织异种移植SCID小鼠模型中,相比于野生型毒株,LAT 突变株(17ΔN/H)并不能在皮肤组织中有效复制并产生皮损表型[18],该研究表明,HSV1 LAT 有助于病毒体内的复制,其作用机制可能依赖于其潜在的表观遗传调控功能。
3. HSV1感染中的表观遗传调控
3.1 表观遗传调控
表观遗传调控是指非DNA编码依赖的基因表达调控方式,如RNA/DNA甲基化或染色质修饰[2]。DNA甲基化是由一系列DNA甲基转移酶(DNA methyltransferases,DNMTs)催化的,将一个甲基从s-腺苷蛋氨酸(SAM)转移到胞嘧啶残基的第5个碳上,形成5mC[19]。而组蛋白修饰通常发生于组蛋白氨基末端,一般包含甲基化、乙酰化和磷酸化,不同修饰的组蛋白通过结合染色质重塑复合物,参与基因组染色质构象调控,最终影响基因表达。例如,组蛋白乙酰化通常发生于转录活性基因上,其主要在启动子和编码区的5’ 端的特定位点富集[20]。
3.2 HSV1裂解感染过程中的表观遗传调控
早期对HSV1表观遗传修饰的研究发现,衣壳包裹的病毒基因组DNA在微球核酸酶(microsphere nuclease,MCN)作用下未产生类似宿主细胞的典型核小体条带,这表明病毒DNA未结合组蛋白形成染色质结构[21],而且,在裂解感染的细胞中,HSV1DNA在MCN酶解下也未能形成核小体条带;相反,潜伏感染的三叉神经节中病毒DNA却呈现出高度浓缩的异染色质化[21-23]。然而随着染色质免疫共沉淀(chromatin immunoprecipitation,ChIP)技术的应用,越来越多的证据表明HSV1裂解感染中也存在着广泛的染色质表观遗传学修饰。Huang和Kent研究[24-25]发现,裂解感染细胞中,病毒DNA能够与组蛋白H3共沉淀,并且病毒ICP0,TK及VP16基因启动子上的H3K9Ac及K3K14c修饰水平与其基因转录水平呈正相关,这充分表明病毒裂解感染受到显著的染色质表观遗传调控。
事实上,初始感染中HSV1双链DNA分子输入宿主细胞核之后,会即刻进行非DNA复制依赖的核小体组装。Paulus等[26]研究表明,这种快速的外源DNA染色质形成可能是一种由细胞的ND10(Nuclear domain 10)介导的固有抗病毒防御机制,促进宿主细胞对外来DNA 的沉默。Lee等[27]使用原代人包皮成纤维细胞裂解感染模型的ChIP分析也显示,HSV1感染的1~2 h内会迅速进行核小体组装,而抑制性异染色质修饰H3K9me3和H3K27me3会迅速增加,其后随感染进程逐渐降低。此外,HSV1裂解感染中,转录开放性染色质修饰如H3K27Ac等随病毒复制进程逐渐增加[28],且药物抑制H3K27Ac能够显著抑制病毒复制[29]。
但HSV1也进化出抵抗宿主表观遗传沉默的机制,主要表现在病毒基因组上异染色质修饰逐渐去除和常染色质修饰增加[30]。而与病毒DNA一同进入宿主细胞核的被膜蛋白VP16和VP22被证实参与抵抗宿主细胞组蛋白介导的表观遗传沉默。如上所述,VP16随病毒基因组进入宿主细胞核后,除参与招募RNA聚合酶外,VP16也能够招募染色质修饰共激活因子如CBP与p300,以及染色质重塑复合物BRG1与BRM到病毒IE基因启动子上,从而参与病毒复制早期的表观遗传调控[5],见图1。VP16蛋白在裂解性感染期间降低IE基因上的总染色质水平,并促进与HSV裂解性基因相关的组蛋白常染色质修饰[5]。此外,病毒即早期基因转录产物,如ICP0与ICP4也被证明可以减少病毒裂解基因启动子上的异染色质并且增加组蛋白乙酰化修饰。ICP0作为E3泛素连接酶,能够降解着丝粒相关抑制性组蛋白变体如CENP-A,CENP-B等。同时ICP0也能够通过与组蛋白去乙酰酶HDAC1/2相互作用,干扰REST/CoREST转录抑制复合物组成,从而促进病毒染色质上组蛋白乙酰化修饰[31]。ICP4作为病毒DNA结合蛋白也能够通过结合或干扰宿主细胞染色质重塑因子NuRD与INO80,调控病毒基因组表观修饰[32]。VP16[33]或ICP0[34]缺陷的病毒突变体会造成病毒裂解相关基因启动子的异染色质增加,表明病毒的被膜蛋白和IE蛋白的表达会在裂解感染过程中促进组蛋白的常染色质修饰从而促进裂解基因的表达。由此可见,HSV1裂解感染中,预先存在的病毒被膜蛋白及早期转录产物对宿主的表观遗传调控是维持感染进程的关键因素。
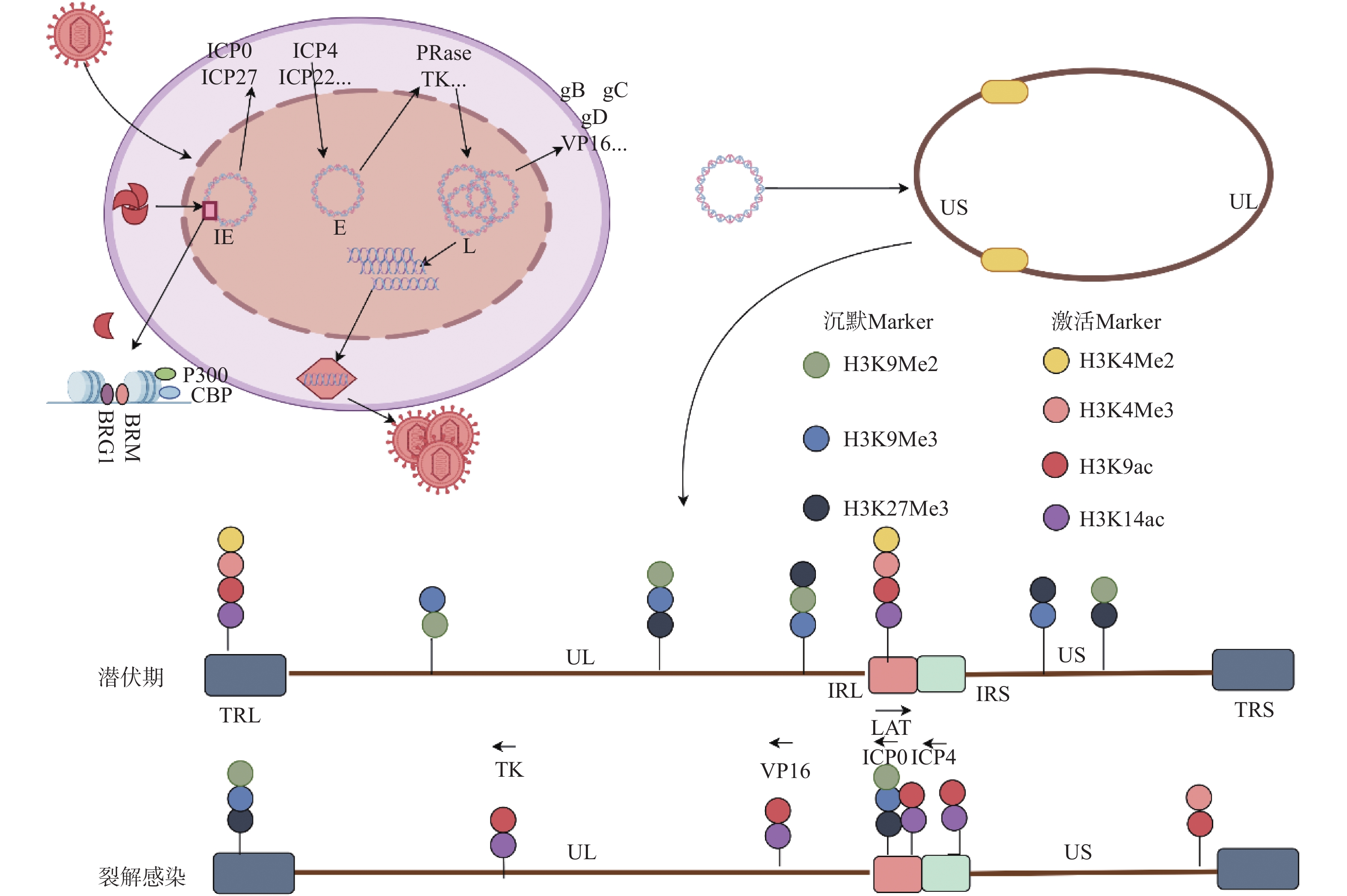 图 1 HSV1基因的级联表达和表观修饰VP16:病毒蛋白16;IE:即早期基因;E:早期基因;L:晚期基因UL:基因组长片段区;US:基因组短片段区;RL:长片段重复区;RS:短片段重复区;图片来源于Figdraw[2] 。Figure 1. Cascade expression and epigenetic modification of HSV1 gene
图 1 HSV1基因的级联表达和表观修饰VP16:病毒蛋白16;IE:即早期基因;E:早期基因;L:晚期基因UL:基因组长片段区;US:基因组短片段区;RL:长片段重复区;RS:短片段重复区;图片来源于Figdraw[2] 。Figure 1. Cascade expression and epigenetic modification of HSV1 gene3.3 潜伏感染中的表观遗传调控
3.3.1 潜伏基因组上组蛋白修饰
在潜伏期,HSV1的显著特点是裂解基因表达沉默和潜伏期相关转录本(LATs)的持续表达[35]。这种将基因组划分为转录活性区和非转录活性区的方式,表明HSV1在潜伏期存在表观遗传控制[36]。先前Nicole J. Kubat[29]通过研究潜伏期小鼠背根神经节分离的DNA,发现在任何区域都没有明显的DNA甲基化,包括转录丰富的LAT启动子和裂解基因ICP4,表明病毒的转录不受DNA甲基化的调节,可能受到翻译后组蛋白修饰的调节。
早在1989年,Deshmane和Fraser发现潜伏感染的神经元中病毒基因组与核小体结合,形成类似于宿主基因组相似的染色质样结构[22,37]。随后Bloom,Neumann和Hill小组通过使用染色质免疫沉淀进一步确认了潜伏基因组上的染色质化[29,37-39]。进一步研究表明,LAT区域是HSV1潜伏基因组中唯一富含转录允许性标记的区域,其含有大量的H3K4Me2,H3K4Ac和H3K14Ac等组蛋白修饰[40]。相比而言,潜伏期裂解基因上存在着大量的异染色质,其通常与转录不活跃的基因有关,包含2种形式:一种是结构异染色质,被认为是不可逆的,如结构异染色质标记H3K9me2大量存在于潜伏期裂解基因上[41];另一种为兼性异染色质,是一种可逆抑制,其标记H3K27Me3和macroH2A也在潜伏期在裂解基因上富集[40],见图1。
3.3.2 潜伏期LAT的表观调控
目前研究认为, HSV1基因组上广泛分布的绝缘子序列元件是维持转录活性区与非转录活性区空间分隔的主要因素;其主要通过与特定结合蛋白如CTCF与Suz12等,形成特定的基因组三维结构,将LAT常染色质活性保持在一个边界内,而将异染色质活性保持在另一边界外[42]。Amelio等[42]通过软件预测HSV1基因组存在7个独立的CTCF结合位点,包括CTRL1,CTRL2,CTa’ m,CTRS1,CTRS2,CTRS3与CTUS1,其中大部分位点位于基因组重复区内的ICP0,ICP4和LAT等关键基因附近,见图2。而Lang等[43]应用CTCF靶向的ChIP-seq鉴定出HSV1裂解感染中病毒基因组至少存在25个CTCF结合位点,且上述预测位点如CTRL2与CTRS3并未结合CTCF蛋白。这充分表明,HSV1裂解与潜伏感染中,病毒基因组上的绝缘子元件与CTCF的结合存在着动态调节,此外,研究显示,AAV介导的小鼠三叉神经节CTCF敲低促进了潜伏病毒ICP0转录,而ICP0恰位于CTRL2与CTa’ m绝缘子之间[44],并且,敲除HSV1 CTRL2绝缘子的突变株表现出增强的毒性,并且更难维持潜伏状态[44]。三叉神经节潜伏的突变株病毒出现明显的ICP0和ICP27表达,以及病毒IE基因启动子转录抑制性组蛋白修饰H3K27Me3显著减少[45],这表明,病毒自身的绝缘子元件的组装参与调控裂解-潜伏感染中的病毒基因组结构改变及表观遗传修饰调控。
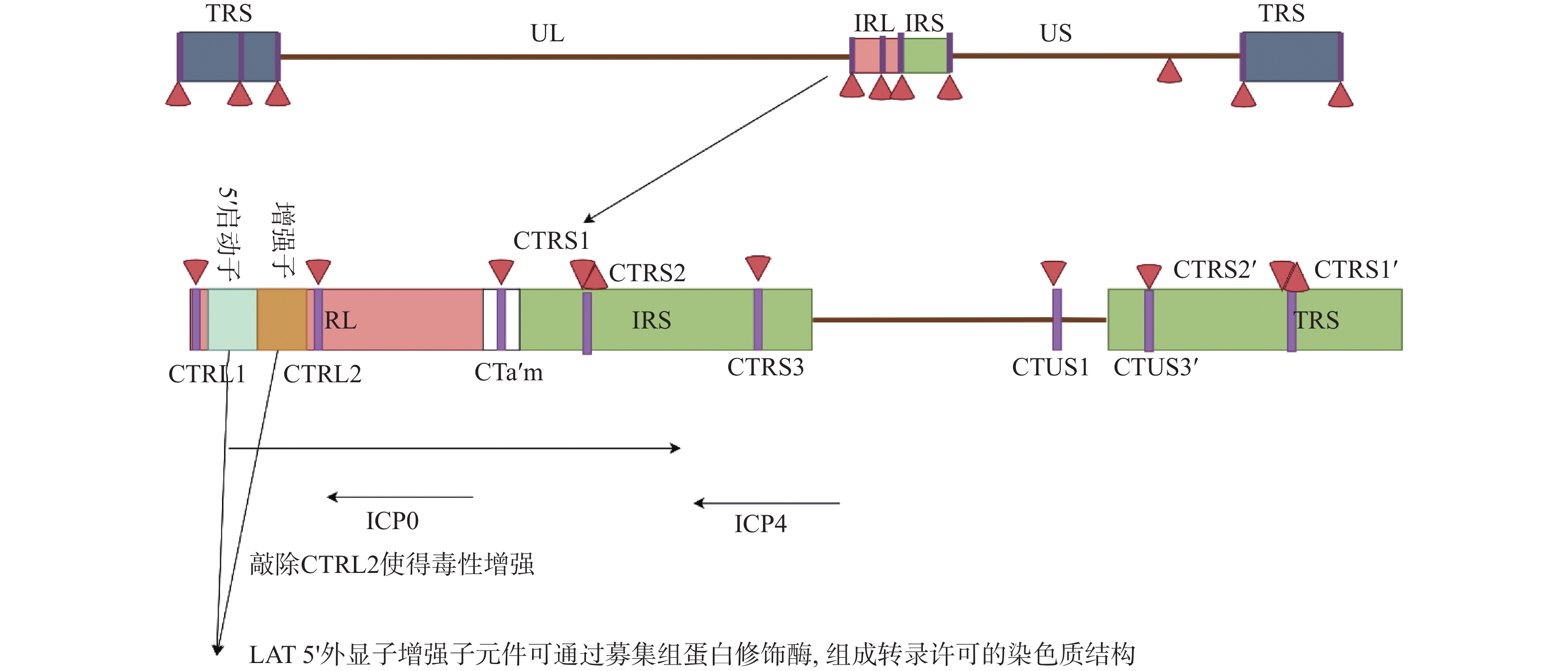 图 2 LAT上CTCF结合位点和LAT的表观调控CTCF结合位点,包括CTRL1,CTRL2,CTa’ m,CTRS1,CTRS2,CTRS3与CTUS1[42]Figure 2. CTCF binding sites and epigenetic regulation of LAT on LAT
图 2 LAT上CTCF结合位点和LAT的表观调控CTCF结合位点,包括CTRL1,CTRL2,CTa’ m,CTRS1,CTRS2,CTRS3与CTUS1[42]Figure 2. CTCF binding sites and epigenetic regulation of LAT on LAT除绝缘子外,LAT启动子区的不同调控元件也可能参与病毒潜伏-重激活过程中的表观遗传调控。LAT 5’ 外显子增强子元件可通过募集组蛋白修饰酶,组成转录许可的染色质结构[39]。此外,LAT启动子调控区也存在甲状腺激素响应元件TRE。在体外感染的神母细胞瘤细胞系中,甲状腺激素受体TR能够直接结合TRE元件,促进CTCF蛋白与高乙酰化组蛋白H4在LAT区富集,进而激活LAT转录并抑制ICP0表达[46]。
另一方面,病毒潜伏期LAT转录本自身是否直接参与基因组表观遗传修饰仍存在较大争议。Bloom等[40]使用17 syn+株的LAT启动子缺失病毒17△Pst(未影响绝缘子序列)发现,LAT的缺失会导致潜伏病毒基因组H3K27me3水平增加。相比之下,Knipe等[7]使用KOS株LAT启动子缺失病毒KOS△Pst观察到潜伏基因组上H3K27me3水平降低。此外,运用体外神经元潜伏-重激活的模型研究也显示,对比2种HSV1野生型菌株及其各自的LAT启动子缺失病毒,LAT以一种菌株特异性的方式影响染色质标记水平、病毒转录和病毒蛋白产生[47]。
4. 小结
HSV1因其可以建立潜伏期并具有从潜伏期重激活的能力,成为一种在人体内建立终身潜伏感染的病毒。目前对于HSV1的治疗主要是采用小分子抑制剂抑制裂解期病毒的复制。但不可否认,目前治疗药物并不能靶向潜伏感染中病毒;而低免疫人群中HSV1的反复发作也可能促进病毒耐药性突变的积累,从而成为HSV1彻底根除的一大障碍。考虑到表观遗传调控其在HSV1裂解和潜伏感染维持中扮演的关键作用,可以推测表观修饰相关小分子药物如DNA甲基转移酶抑制剂,组蛋白乙酰化/甲基化酶抑制剂等有希望成为全新的HSV1抗病毒药物,同时靶向裂解/潜伏中的病毒。
目前对裂解/潜伏中的HSV1病毒基因组完整的染色质组成及组蛋白修饰类型的理解尚不充分。随着新型高通量表观组学测序与空间组学技术的开发及应用,有望解析HSV1感染中基因组结构组成的全貌及动态调控。此外,除组蛋白修饰外,其他表观遗传修饰如RNAm6A以及DNA水平的5mC修饰是否参与调控HSV1感染进程等问题仍待进一步研究。
-
图 1 HSV1基因的级联表达和表观修饰
VP16:病毒蛋白16;IE:即早期基因;E:早期基因;L:晚期基因UL:基因组长片段区;US:基因组短片段区;RL:长片段重复区;RS:短片段重复区;图片来源于Figdraw[2] 。
Figure 1. Cascade expression and epigenetic modification of HSV1 gene
图 2 LAT上CTCF结合位点和LAT的表观调控
CTCF结合位点,包括CTRL1,CTRL2,CTa’ m,CTRS1,CTRS2,CTRS3与CTUS1[42]
Figure 2. CTCF binding sites and epigenetic regulation of LAT on LAT
-
[1] Arduino P G,Porter S R. Herpes simplex virus type 1 infection: Overview on relevant clinico-pathological features[J]. Journal of Oral Pathology & Medicine:Official Publication of the International Association of Oral Pathologists and the American Academy of Oral Pathology,2008,37(2):107-121. [2] Schang L M,Hu M,Cortes E F,et al. Chromatin-mediated epigenetic regulation of HSV-1 transcription as a potential target in antiviral therapy[J]. Antiviral Research,2021,192(1):105103-105139. [3] Steiner I,Benninger F. Update on herpes virus infections of the nervous system[J]. Current Neurology and Neuroscience Reports,2013,13(12):414-421. doi: 10.1007/s11910-013-0414-8 [4] Lehman I R,Boehmer P E. Replication of herpes simplex virus DNA[J]. The Journal of Biological Chemistry,1999,274(40):28059-28062. doi: 10.1074/jbc.274.40.28059 [5] Knipe D M,Cliffe A. Chromatin control of herpes simplex virus lytic and latent infection[J]. Nature Reviews Microbiology,2008,6(3):211-221. doi: 10.1038/nrmicro1794 [6] Zhu S,Viejo-Borbolla A. Pathogenesis and virulence of herpes simplex virus[J]. Virulence,2021,12(1):2670-2702. doi: 10.1080/21505594.2021.1982373 [7] Cliffe A R,Garber D A,Knipe D M. Transcription of the herpes simplex virus latency-associated transcript promotes the formation of facultative heterochromatin on lytic promoters[J]. J Virol,2009,83(16):8182-8190. doi: 10.1128/JVI.00712-09 [8] Stevens J G,Wagner E K,Devi-rao G B,et al. RNA complementary to a herpesvirus alpha gene mRNA is prominent in latently infected neurons[J]. Science (New York,NY),1987,235(4792):1056-1059. [9] Zwaagstra J C,Ghiasi H,Slanina S M,et al. Activity of herpes simplex virus type 1 latency-associated transcript (LAT) promoter in neuron-derived cells: Evidence for neuron specificity and for a large LAT transcript[J]. J Virol,1990,64(10):5019-5028. doi: 10.1128/jvi.64.10.5019-5028.1990 [10] Farrell M J,Dobson A T,Feldman L T. Herpes simplex virus latency-associated transcript is a stable intron[J]. Proceedings of the National Academy of Sciences of the United States of America,1991,88(3):790-794. [11] Pan D,Flores O,Umbach J L,et al. A neuron-specific host microRNA targets herpes simplex virus-1 ICP0 expression and promotes latency[J]. Cell Host & Microbe,2014,15(4):446-456. [12] Umbach J L,Nagel M A,Cohrs R J,et al. Analysis of human alphaherpesvirus microRNA expression in latently infected human trigeminal ganglia[J]. J Virol,2009,83(20):10677-10683. doi: 10.1128/JVI.01185-09 [13] Umbach J L,Kramer M F,Jurak I,et al. MicroRNAs expressed by herpes simplex virus 1 during latent infection regulate viral mRNAs[J]. Nature,2008,454(7205):780-783. doi: 10.1038/nature07103 [14] Jiang X,Brown D,Osorio N,et al. Increased neurovirulence and reactivation of the herpes simplex virus type 1 latency-associated transcript (LAT)-negative mutant dLAT2903 with a disrupted LAT miR-H2[J]. Journal of Neurovirology,2016,22(1):38-49. doi: 10.1007/s13365-015-0362-y [15] Perng G C,Jones C,Ciacci-Zanella J,et al. Virus-induced neuronal apoptosis blocked by the herpes simplex virus latency-associated transcript[J]. Science (New York,NY),2000,287(5457):1500-1503. [16] Javier R T,Stevens J G,Dissette V B,et al. A herpes simplex virus transcript abundant in latently infected neurons is dispensable for establishment of the latent state[J]. Virology,1988,166(1):254-257. doi: 10.1016/0042-6822(88)90169-9 [17] Leib D A,Bogard C L,Kosz-Vnenchak M,et al. A deletion mutant of the latency-associated transcript of herpes simplex virus type 1 reactivates from the latent state with reduced frequency[J]. J Virol,1989,63(7):2893-2900. doi: 10.1128/jvi.63.7.2893-2900.1989 [18] Vanni E A H,Foley J W,Davison A J,et al. The latency-associated transcript locus of herpes simplex virus 1 is a virulence determinant in human skin[J]. PLoS Pathog,2020,16(12):e1009166-e1009196. doi: 10.1371/journal.ppat.1009166 [19] Moore L D,Le T,Fan G. DNA methylation and its basic function[J]. Neuropsychopharmacology:Official Publication of the American College of Neuropsychopharmacology,2013,38(1):23-38. doi: 10.1038/npp.2012.112 [20] Kouzarides T. Chromatin modifications and their function[J]. Cell,2007,128(4):693-705. doi: 10.1016/j.cell.2007.02.005 [21] Leinbach S S, Summers W C. The structure of herpes simplex virus type 1 DNA as probed by micrococcal nuclease digestion [J]. The Journal of General Virology, 1980, 51(Pt 1): 45-59. [22] Deshmane S L,Fraser N W. During latency,herpes simplex virus type 1 DNA is associated with nucleosomes in a chromatin structure[J]. J Virol,1989,63(2):943-947. doi: 10.1128/jvi.63.2.943-947.1989 [23] Muggeridge M I,Fraser N W. Chromosomal organization of the herpes simplex virus genome during acute infection of the mouse central nervous system[J]. J Virol,1986,59(3):764-767. doi: 10.1128/jvi.59.3.764-767.1986 [24] Huang J,Kent J R,Placek B,et al. Trimethylation of histone H3 lysine 4 by set1 in the lytic infection of human herpes simplex virus 1[J]. J Virol,2006,80(12):5740-5746. doi: 10.1128/JVI.00169-06 [25] Kent J R,Zeng P Y,Atanasiu D,et al. During lytic infection herpes simplex virus type 1 is associated with histones bearing modifications that correlate with active transcription[J]. J Virol,2004,78(18):10178-10186. doi: 10.1128/JVI.78.18.10178-10186.2004 [26] Paulus C,Nitzszhe A,Nevels M. Chromatinisation of herpesvirus genomes[J]. Reviews in Medical Virology,2010,20(1):34-50. doi: 10.1002/rmv.632 [27] Lee J S,Raja P,Knipe D M. Herpesviral ICP0 protein promotes wwo waves of heterochromatin removal on an early viral promoter during lytic infection[J]. mBio,2016,7(1):e02007-e02015. [28] Gao C,Chen L,Tang S B,et al. The epigenetic landscapes of histone modifications on HSV-1 genome in human THP-1 cells[J]. Antiviral Research,2020,176(1):104730-104741. doi: 10.1016/j.antiviral.2020.104730 [29] Kubat N J,Tran R K,Mcanany P,et al. Specific histone tail modification and not DNA methylation is a determinant of herpes simplex virus type 1 latent gene expression[J]. J Virol,2004,78(3):1139-1149. doi: 10.1128/JVI.78.3.1139-1149.2004 [30] Oh J,Fraser N W. Temporal association of the herpes simplex virus genome with histone proteins during a lytic infection[J]. J Virol,2008,82(7):3530-3537. doi: 10.1128/JVI.00586-07 [31] Gross S,Catez F,Masumoto H,et al. Centromere architecture breakdown induced by the viral E3 ubiquitin ligase ICP0 protein of herpes simplex virus type 1[J]. PloS One,2012,7(9):e44227-e44240. doi: 10.1371/journal.pone.0044227 [32] Wagner L M,Deluca N A. Temporal association of herpes simplex virus ICP4 with cellular complexes functioning at multiple steps in PolII transcription[J]. PloS One,2013,8(10):e78242-e78254. doi: 10.1371/journal.pone.0078242 [33] Herrera F J,Triezenberg S J. VP16-dependent association of chromatin-modifying coactivators and underrepresentation of histones at immediate-early gene promoters during herpes simplex virus infection[J]. J Virol,2004,78(18):9689-9696. doi: 10.1128/JVI.78.18.9689-9696.2004 [34] Cliffe A R,Knipe D M. Herpes simplex virus ICP0 promotes both histone removal and acetylation on viral DNA during lytic infection[J]. J Virol,2008,82(24):12030-12038. doi: 10.1128/JVI.01575-08 [35] Arbuckle J H,Vogel J L,Efstathiou S,et al. Deletion of the transcriptional coactivator HCF-1 in vivo impairs the removal of repressive heterochromatin from latent HSV genomes and suppresses the initiation of viral reactivation[J]. mBio,2023,14(1):e0354222-e0354238. doi: 10.1128/mbio.03542-22 [36] Bloom D C,Giordani N V,Kwiakkowski D L. Epigenetic regulation of latent HSV-1 gene expression[J]. Biochim Biophys Acta,2010,1799(3-4):246-256. doi: 10.1016/j.bbagrm.2009.12.001 [37] Neumann D M,Bhattacharjee P S,GIORDANI N V,et al. In vivo changes in the patterns of chromatin structure associated with the latent herpes simplex virus type 1 genome in mouse trigeminal ganglia can be detected at early times after butyrate treatment[J]. J Virol,2007,81(23):13248-13253. doi: 10.1128/JVI.01569-07 [38] Amelio A L,Giordani N V,Kubat N J,et al. Deacetylation of the herpes simplex virus type 1 latency-associated transcript (LAT) enhancer and a decrease in LAT abundance precede an increase in ICP0 transcriptional permissiveness at early times postexplant[J]. J Virol,2006,80(4):2063-2068. doi: 10.1128/JVI.80.4.2063-2068.2006 [39] Kubat N J,Amelio A L,Giordani N V,et al. The herpes simplex virus type 1 latency-associated transcript (LAT) enhancer/rcr is hyperacetylated during latency independently of LAT transcription[J]. J Virol,2004,78(22):12508-12518. doi: 10.1128/JVI.78.22.12508-12518.2004 [40] Kwiatkowski D L,Thompson H W,Bloom D C. The polycomb group protein Bmi1 binds to the herpes simplex virus 1 latent genome and maintains repressive histone marks during latency[J]. J Virol,2009,83(16):8173-8181. doi: 10.1128/JVI.00686-09 [41] Wang Q Y,Zhou C,Johnson K E,et al. Herpesviral latency-associated transcript gene promotes assembly of heterochromatin on viral lytic-gene promoters in latent infection[J]. Proceedings of the National Academy of Sciences of the United States of America,2005,102(44):16055-16059. [42] Amelio A L,Mcanany P K,Bloom D C. A chromatin insulator-like element in the herpes simplex virus type 1 latency-associated transcript region binds CCCTC-binding factor and displays enhancer-blocking and silencing activities[J]. J Virol,2006,80(5):2358-2368. doi: 10.1128/JVI.80.5.2358-2368.2006 [43] Lang F,Li X,Vladimirova O,et al. CTCF interacts with the lytic HSV-1 genome to promote viral transcription[J]. Scientific Reports,2017,7(1):39861-39876. doi: 10.1038/srep39861 [44] Washington S D,Musarrat F,ERTEL M K,et al. CTCF binding sites in the herpes simplex virus 1 genome display site-specific CTCF occupation,protein recruitment,and insulator function[J]. J Virol,2018,92(8):e00156-e00171. [45] Washington S D,Singh P,Johns R N,et al. The CCCTC binding factor,CTRL2,modulates heterochromatin deposition and the establishment of herpes shimplex virus 1 latency in vivo[J]. J Virol,2019,93(13):e00415-e00419. [46] Bedadala G R,Pinnoji R C,Palem J R,et al. Thyroid hormone controls the gene expression of HSV-1 LAT and ICP0 in neuronal cells[J]. Cell Res,2010,20(5):587-598. doi: 10.1038/cr.2010.50 [47] Grams T R,Edwards T G,Bloom D C. HSV-1 LAT promoter deletion viruses exhibit strain-specific and LAT-dependent epigenetic regulation of latent viral genomes in human neurons[J]. J Virol,2023,97(2):e01935-e01951. 期刊类型引用(1)
1. 周磊,吕雯雯,段永忠,钱雯,程继帅. 靶向抗HSV-1的siRNA研究进展. 昆明医科大学学报. 2024(09): 1-8 .  本站查看
本站查看其他类型引用(0)
-






 下载:
下载:
 下载:
下载:

