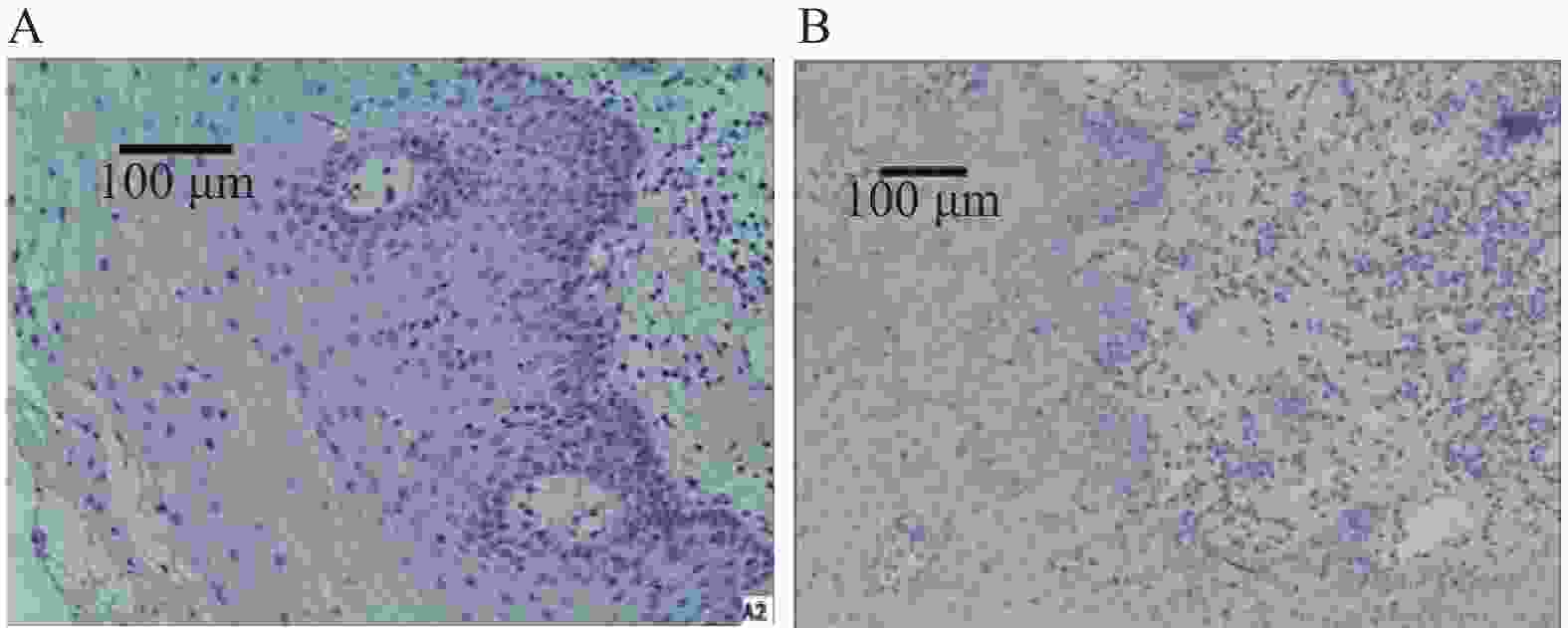
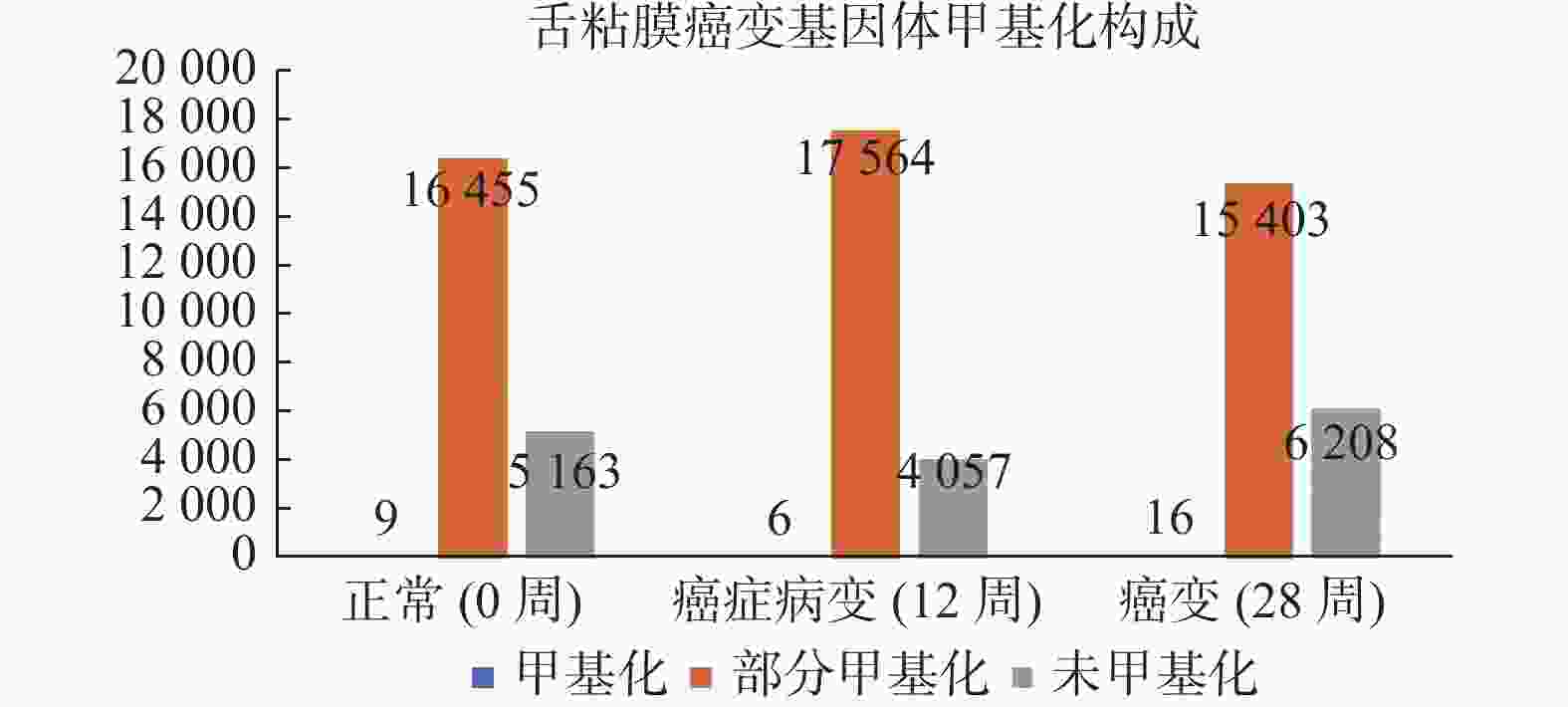
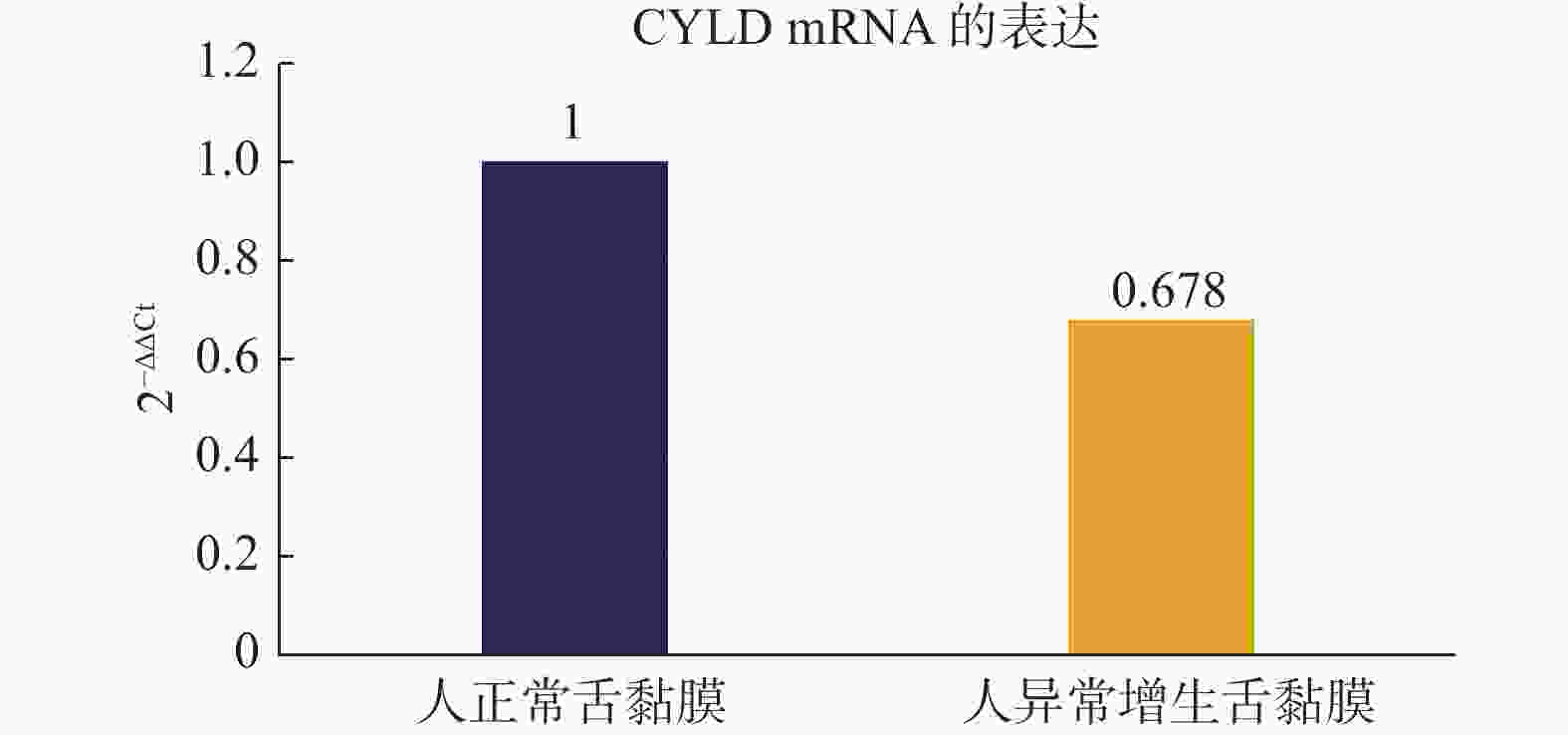
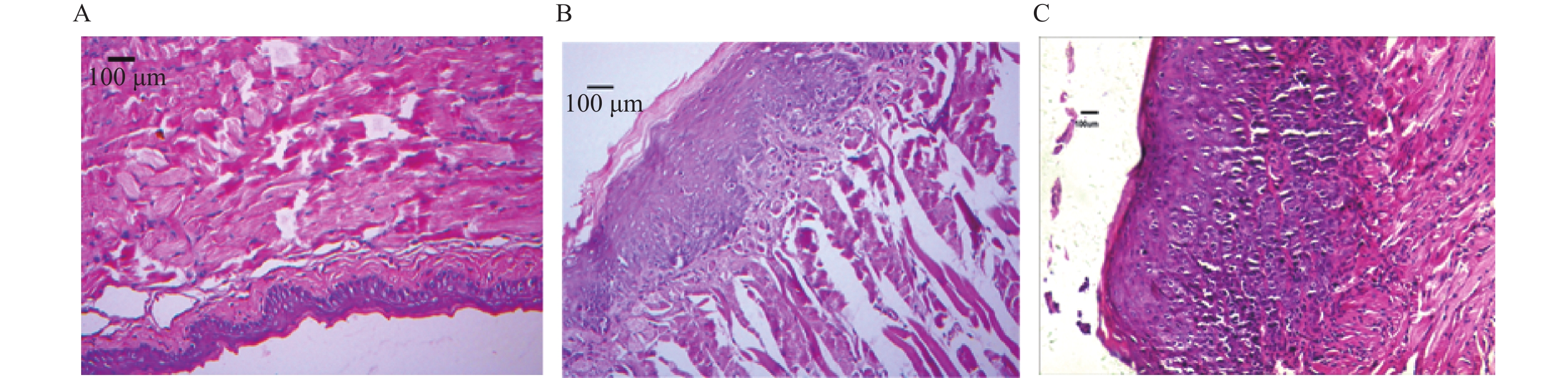
Changes of Genebody Methylation in Lingual Carcinogenesis
-
摘要:
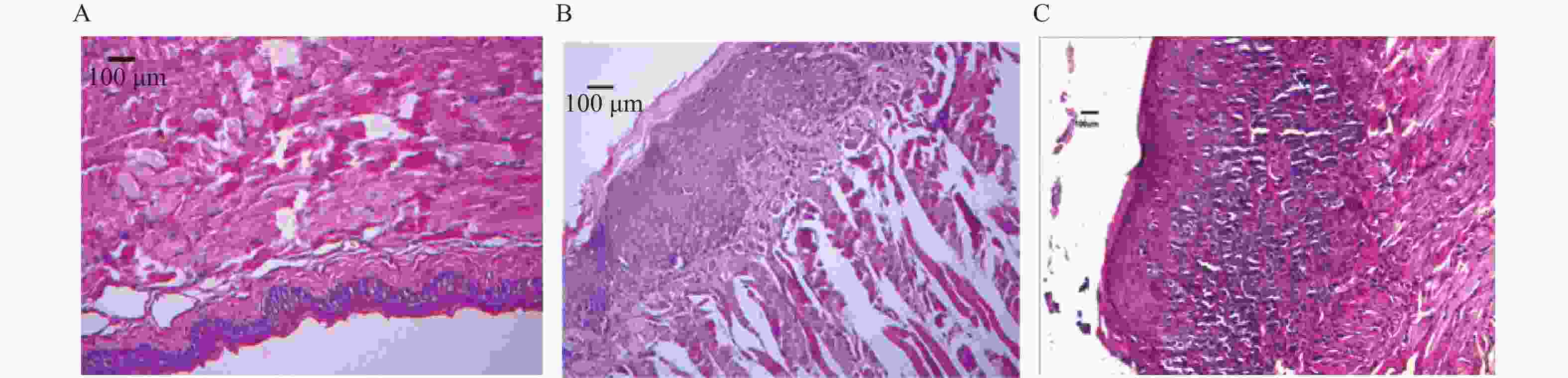
目的 了解舌粘膜癌变中基因体甲基化的差异表达基因。 方法 用浓度50 mg/L的4-硝基喹啉-1-氧化物(4NQO)诱导C57BL/6小鼠舌癌。基因芯片和甲基化DNA免疫沉淀测序(MeDIP-Seq)检测0周、12周、28周(代表正常、癌前、癌变)的舌粘膜的基因表达和基因组DNA甲基化。在人正常舌粘膜和癌前病变组织中用qRT-PCR和飞行质谱检测泛素羧基水解酶CYLD的mRNA表达和基因体甲基化。 结果 正常、癌前和舌癌3组间基因体MeDIP-score(mean±s)分别是(5.58±14.80)、(5.79±13.35)、(5.83±17.25),差异无统计学意义(H = 100.75,P > 0.05)。3组间145个差异表达基因伴基因体甲基化改变。CYLD在人癌前病变组织中表达降低伴有基因体甲基化降低(P < 0.05)。 结论 舌粘膜癌变有多个基因体甲基化异常的差异表达基因,CYLD在人癌前病变中低表达可能与基因体甲基化有关。 Abstract:Objective To evaluate differentially expressed genes with correlation of gene body methylation . Methods The lingual carcinogenesis was induced through the delivery of drinking water of 4-nitroquinoline-1-oxide(4NQO) with a density of 50 mg/L. The lingual mucosae specimens were harvested at 0, 12, 28 weeks, which represented normal, pre-cancer, and cancer individually. The microarray and methylated DNA immunoprecipitation sequencing (MeDIP-Seq) were used for the evaluation of gene expression and global DNA methylation in these specimens. The mRNA expression of Ubiquitin carboxyl-terminal hydrolase CYLD was tested with qRT-PCR in human normal and dysplasia mucosa. Results There were no statistical differences in gene body methylation among normal(C), pre-cancer(M), and cancer(E)(mean±sofMeDIP-score were 5.58±14.80, 5.79±13.35, 5.83±17.25 individually, H = 100.75, P > 0.05)There presented 145 differentially expressed genes with positive correlation to gene body methylation changes.The expression of CYLD mRNA was decreased in human dysplasia compared to normal mucosa with downregulation of gene body methylation. Conclusions There are many differentially expressed genes with a positive correlation to gene body methylations. The expression of CYLD is downregulated with the downregulation of gene body methylation in earlier stage during tongue carcinogenesis. -
Key words:
- Tongue cancer /
- Animal model /
- Methylation /
- Gene body /
- Differentially expressed gene
-
表 1 基因体甲基化异常的差异表达基因
Table 1. Differentially expressed genes with positive correlation to gene body methylation
序号 癌前vs正常 癌变vs正常 癌变vs癌前 上调 下调 上调 下调 上调 下调 1 Gm12657 Ddx25 Hps3 Irf3 Odf2 Msln 2 Rnf14 Cyld Mreg Zfp939 Uba1 Slc35a5 3 Rsbn1l 2610507B11Rik Gm12657 Tesk1 2010012O05Rik Csprs 4 Celf2 5330434G04Rik Edaradd Gab3 Wdyhv1 Ywhaz 5 Mapk3 Dclk2 Gm7120 Ntng1 Cdca5 Ada 6 Rdh9 Ecsit Pemt Sema3b Fbxo24 Bpifb6 7 Ache Il20 Plekhb2 Ptges3l Tmem37 A730008H23Rik 8 Stard10 AI846148 Nop14 Bace2 Spp1 Stard10 9 Klhl7 Iars2 Hivep1 Gm3086 Plekhg4 Casc4 10 Mfsd1 Gm3086 Fam98b Kcnn4 Fam81a Igsf5 11 Arf4 Zfp763 Oas2 a Fubp1 12 Zbtb18 Pptc7 Tbx22 Ewsr1 Cdkl5 13 Kcnj2 Cdc42se2 Mfsd2a Fut2 14 Ap1s2 Slfn4 Shc4 H2-T9 15 Setd1b Npl Tspan32 16 Cyth3 Exoc6 Car4 17 Nek1 Son Mapkapk2 18 Cpn1 Rsbn1l Slc25a39 19 Aqr Nacad Eif5a 20 Abca5 Ddx20 Bace2 21 Pomgnt1 Atf1 Hira 22 Nfkb1 Vps37c Azgp1 23 Kalrn Nfkbiz Hmgcll1 24 Dsg1b Prkaa1 Cfl1 25 Sec62 Pik3c2a Mamstr 26 Camk2a Ankfn1 Celf2 27 Ube2q2 Mfsd1 Gfra4 28 Ppp4r2 Cdk1 Papss2 29 Lrrc58 Bcas2 Smurf2 30 Tnpo1 Rad21 Cat 31 Zfp763 Zfand1 Myoz3 32 Il2rg Mmgt1 Tmed3 33 Slc25a39 Trip13 Prb1 34 Nfe2l1 Dyrk1b 35 Tob1 Prrt2 36 H2-T10 37 Gyk 38 Tmem55b 39 Zfp386 40 Akr1b3 41 Tnfaip6 -
[1] Siegel R L,Giaquinto A N,Jemal A. Cancer statistics,2024[J]. CA Cancer J Clin,2024,74(1):12-49. doi: 10.3322/caac.21820 [2] 代晓明,刘华,左志斌,等. 4-硝基喹啉-1-氧化物诱导C57BL/6小鼠舌黏膜癌变的研究[J]. 华西口腔医学杂志,2015,33(4):357-360. doi: 10.7518/hxkq.2015.04.007 [3] Lai Z L,Tsou Y A,Fan S R,et al. Methylation-associated gene silencing of RARB in areca carcinogens induced mouse oral squamous cell carcinoma[J]. Biomed Res Int,2014,2014:378358. [4] Human D. Declaration of helsinki[J]. Lancet,2001,357(9251):236. [5] Kundaje A,Meuleman W,Ernst J,et al. Integrative analysis of 111 reference human epigenomes[J]. Nature,2015,518(7539):317-330. doi: 10.1038/nature14248 [6] Lorincz M C,Dickerson D R,Schmitt M,et al. Intragenic DNA methylation alters chromatin structure and elongation efficiency in mammalian cells[J]. Nat Struct Mol Biol,2004,11(11):1068-1075. doi: 10.1038/nsmb840 [7] Naftelberg S,Schor I E,Ast G,et al. Regulation of alternative splicing through coupling with transcription and chromatin structure[J]. Annu Rev Biochem,2015,84:165-198. doi: 10.1146/annurev-biochem-060614-034242 [8] Bell J S K,Vertino P M. Orphan CpG islands define a novel class of highly active enhancers[J]. Epigenetics,2017,12(6):449-464. doi: 10.1080/15592294.2017.1297910 [9] Huang K K,Ramnarayanan K,Zhu F,et al. Genomic and epigenomic profiling of high-risk intestinal metaplasia reveals molecular determinants of progression to gastric cancer[J]. Cancer Cell,2018,33(1):137-150e5. doi: 10.1016/j.ccell.2017.11.018 [10] Zhang F F,Zhang X,Zhang H K,et al. Pan-precancer and cancer DNA methylation profiles revealed significant tissue specificity of interrupted biological processes in tumorigenesis[J]. Epigenetics,2023,18(1):2231222. doi: 10.1080/15592294.2023.2231222 [11] Hernández H G,Aranzazu-Moya G C,Pinzón-Reyes E H. Aberrant AHRR,ADAMTS2 and FAM184 DNA methylation: Candidate biomarkers in the oral rinse of heavy smokers[J]. Biomedicines,2023,1(7):1797. [12] Li X,Li Z,Gao Q,et al. Correlation of DNA methylation of DNMT3A and TET2 with oral squamous cell carcinoma[J]. Discov Oncol,2024,15(1):15. doi: 10.1007/s12672-024-00866-9 [13] Gabusi A,Gissi D B,Grillini S,et al. Shared epigenetic alterations between oral cancer and periodontitis: A preliminary study[J]. Oral Dis,2023,9(5):2052-2060. [14] Aghiorghiesei O,Irimie AI,Braicu C,et al. Epigenetic methylation changes: Implication as biomarkers in oral and maxillofacial area cancers[J]. Med Pharm Rep,2023,96(3):310-317. [15] Okoturo E,Green D,Clarke K,et al. Whole genome DNA methylation and mutational profiles identify novel changes in proliferative verrucous leukoplakia[J]. Oral Surg Oral Med Oral Pathol Oral Radiol,2023,135(6):893-903. doi: 10.1016/j.oooo.2023.03.004 [16] Rivera-Peña B,Folawiyo O,Turaga N,et al. Promoter DNA methylation patterns in oral,laryngeal and oropharyngeal anatomical regions are associated with tumor differentiation,nodal involvement and survival[J]. Oncol Lett,2024,27(3):89. doi: 10.3892/ol.2024.14223 [17] Dean K A,von Ahsen O,Görlich D,et al. Signal recognition particle protein 19 is imported into the nucleus by importin 8 (RanBP8) and transportin[J]. J Cell Sci,2001,114(Pt 19): 3479-3485. [18] Giussani P,Maceyka M,Le Stunff H,et al. Sphingosine-1-phosphate phosphohydrolase regulates endoplasmic reticulum-to-golgi trafficking of ceramide[J]. Mol Cell Biol,2006,26(13):5055-5069. doi: 10.1128/MCB.02107-05 [19] Lv X,Yu X. Signatures and prognostic values of related immune targets in tongue cancer[J]. Front Surg,2023,9:952389. doi: 10.3389/fsurg.2022.952389 [20] Thakore V P,Patel K D,Vora H H,et al. Up-regulation of extracellular-matrix and inflammation related genes in oral squamous cell carcinoma[J]. Arch Oral Biol,2024,161:105925. doi: 10.1016/j.archoralbio.2024.105925 -





 下载:
下载: