Association of Polymorphisms in the 3' UTR of Genes in the ERK1/2 Signaling Pathway with Non-small Cell Lung Cancer
-
摘要:
目的 探究4个ERK1/2信号通路的基因3'UTR区域的单核苷酸多态性(single nucleotide polymorphism,SNP)位点(MAPK1基因中的rs9340,NRAS基因中的rs14804,KRAS基因中的rs712和rs7973450)与非小细胞肺癌(non-small cell lung cancer,NSCLC)的相关性。 方法 纳入了478名NSCLC患者及480名健康对照者,利用TaqMan探针法对其进行基因分型检测,并分析上述4个SNP与NSCLC的相关性。 结果 rs9340位点的等位基因在对照组与非小细胞鳞状细胞癌组( squamous cell carcinoma,SCC)中分布频率的差异具有统计学意义(P = 0.009),该结果表明rs9340 位点的G等位基因可能是非小细胞肺鳞癌的保护性因素(OR = 0.67,95% CI: 0.50~0.91)。同时,在 < 50岁年龄组中,rs9340位点的等位基因在对照组和NSCLC组中的分布频率差异具有统计学意义(P = 5.07 × 10-4),该结果表明rs9340等位基因G可能是NSCLC的保护性因素(OR = 0.46,95% CI: 0.29~0.72)。 结论 MAPK1基因SNP位点rs9340可能与NSCLC的发生风险相关。 -
关键词:
- 非小细胞肺癌 /
- ERK1/2信号通路 /
- 单核苷酸多态性 /
- 3'UTR /
- MAPK1
Abstract:Objective To investigate the association between four single nucleotide polymorphisms(SNP)(rs9340 in MAPK1, rs14804 in NRAS, rs712 and rs7973450 in KRAS) in the 3'UTR of ERK1/2 signaling pathway-related genes and non-small cell lung cancer(NSCLC). Methods A total of 478 NSCLC patients and 480 healthy controls were enrolled in this study. Four SNPs were genotyped by using TaqMan assays. The association between the four SNPs and NSCLC was analyzed. Results The distribution frequency difference of the allele of rs9340 was statistically significant between the control group and the non-small cell squamous cell carcinoma(SCC) group(P = 0.009), suggesting that the G allele of rs9340 may be a protective factor for non-small cell lung squamous cell carcinoma(OR = 0.67, 95%CI: 0.50~0.91). In addition, in the < 50 years age group, the distribution frequency difference of the allele of rs9340 was statistically significant between the control group and the NSCLC group(P = 5.07 × 10-4), indicating that the G allele of rs9340 may be a protective factor for NSCLC(OR = 0.46, 95%CI: 0.29~0.72). Conclusion The SNP rs9340 in MAPK1 may be associated with the risk of NSCLC. -
Key words:
- Non-small cell lung cancer /
- ERK1/2 signaling pathway /
- SNP /
- 3'UTR /
- MAPK1
-
2020年,全球估计有220万肺癌新发病例和180万死亡病例,约占癌症诊断的十分之一(11.4%)和死亡的五分之一(18.0%)[1]。2016年我国癌症统计报告显示,肺癌是男、女性癌症死亡的最常见原因,约占男性癌症死亡的29.71%和女性癌症死亡的22.92%[2],这主要是因为肺癌早期症状隐匿,难以实现早期诊治[3]。肺癌的发生受多种因素影响[4],而其中遗传因素是影响个体间肺癌易感性差异的最主要因素[5]。非小细胞肺癌(non-small cell lung cancer,NSCLC)是常见的肺癌类型,约占肺癌的85%[6],NSCLC预后较差,且近年来发病趋于年轻化[7]。研究发现,有多条信号通路参与了NSCLC的发生发展过程,如CDKN2A/RB1、NFE2L2/KEAP1/CUL3、PI3K/AKT、SOX2/TP63/NOTCH1等[8-10]。这些信号通路相关基因中的单核苷酸多态性位点(single nucleotide polymorphisms,SNPs)可能与NSCLC的发生发展有相关性[11]。
丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)信号通路在肿瘤发生过程中起关键作用,与肿瘤发生的多个方面有关,包括细胞分化、增殖、侵袭、血管生成、凋亡和转移[12]。细胞外信号调节激酶(ERK)是3个MAPK家族(JNK,p38/SAPK和ERK)之一[13-15]。ERK1/2通路也被称为经典的MAPK信号传导途径,约在34%的肿瘤中发生改变[16]。研究发现,位于基因3'非翻译区(3'untranslated region,3'UTR)的SNP可能会影响miRNA-mRNA相互作用[17-18],进而影响靶基因的表达,可能在肿瘤的发生中发挥关键作用[19]。本研究选取了4个ERK1/2信号通路基因3'UTR区域的SNP位点(MAPK1基因中的rs9340,NRAS基因中的rs14804,KRAS基因中的rs712和rs7973450),探讨其与NSCLC遗传易感性的相关性,以期为肺癌的分子病理机制的探究提供数据支持。
1. 资料与方法
1.1 研究对象
随机选取在昆明医科大学第三附属医院诊断的478名NSCLC患者,纳入标准为:患者经活检或手术诊断为NSCLC、临床资料完整、具有较好的依从性、无其他合并肿瘤、无其他肿瘤既往史、未接受放疗或化疗等抗肿瘤治疗。诊断标准依据《中华医学会肺癌临床诊疗指南(2018版)》[20]。同时随机选取480名同期健康体检人员作为健康对照组。采集所有受试者的乙二胺四乙酸(EDTA)抗凝全血4 mL。所有样本收集均获得患者及家人的知情同意,并经昆明医科大学第三附属医院伦理委员会批准(审查编号:KYLX2022180)。为了响应我国对肺癌筛查与早诊早治的倡议,根据《中国肺癌筛查与早诊早治指南(2021,北京)》[21]以及《老年晚期肺癌内科治疗中国专家共识(2022版)》[22]的推荐,本研究将受试者按照年龄划分为 < 50岁组,50~65岁组以及>65岁组。
1.2 DNA提取
使用全血基因组DNA提取试剂盒QIAamp DNA Mini Kit(购自Qiagen公司,Qiagen NV,Venlo,Netherlands),根据试剂说明书完成基因组DNA提取。使用Multiskan GO全波长酶标仪测定DNA浓度及纯度。将提取到的DNA样本稀释到25 ng/μL的工作浓度,用于随后的基因分型检测。
1.3 SNP选取及分型
使用CAPD数据库(http://bio-bigdata.hrbmu.edu.cn/CPAD/index.jsp)选取与肺癌相关的候选信号通路,利用GEPIA数据库(http://gepia.cancer-pku.cn/)对信号通路中的候选基因进行表达验证,使用Ensembl数据库(https://www.ensembl.org/index.html)及UCSC数据库(https://genome.ucsc.edu/)查找位于候选基因3'UTR区域的SNP位点。同时以中国南方汉族人群次要等位基因频率(minor allele frequency,MAF)高于0.04作为筛选标准。最终选取4个SNP作为本次研究的候选SNP,分别是位于MAPK1基因3'UTR区域的rs9340,位于NRAS基因3'UTR区域的rs14804,位于KRAS基因3'UTR区域的rs712和rs7973450,见表1。
表 1 所选SNP位点信息Table 1. The information of selected SNPs in the current studySNPs 基因 功能 位置 等位基因 中国南方汉族人群MAF rs9340 MAPK1 3'UTR突变 Chr 22:21761064 G > A 0.21 rs14804 NRAS 3'UTR突变 Chr 1:114707222 G > A 0.04 rs712 KRAS 3'UTR突变 Chr 12:25209618 A > C 0.48 rs7973450 KRAS 3'UTR突变 Chr 12:25208208 A > G 0.09 SNP,单核苷酸多态性;MAF,次要等位基因频率;Chr,染色体。 使用TaqMan探针法对上述SNP进行基因分型检测,所用试剂及探针均购自美国ABI公司,具体分型方法参见笔者之前的研究[23]。为验证基因分型结果的准确性,本研究随机挑选5%的样本进行重复检测,并随机挑选了3%的样本进行测序验证。
1.4 生物信息学分析
利用GTEx数据库(https://www.gtexportal.org/home/)评估SNP和基因表达的相关性,使用miRNASNP数据库(http://bioinfo.life.hust.edu.cn/miRNASNP/#!/)预测基因3'UTR变异对miRNA-mRNA相互作用的影响。
1.5 统计学处理
使用Microsoft Excel软件整理所有基因分型数据以及临床信息,采用Graphpad Prism软件和在线软件SHEsis[24]进行统计学分析。研究对象年龄以($\bar x \pm s$)表示,性别、病理类型、临床分期、SNP位点等位基因及基因型以例数和百分比[n(%)]表示。使用独立样本t检验分析病例组和对照组间样本年龄差异;使用χ2检验分析2组间性别分布差异;采用哈迪温伯格平衡(hardy-weinberg equilibrium,HWE)检验分析纳入样本的群体代表性;采用χ2检验分析各SNP位点等位基因及基因型在各组间的分布频率的差异及优势比(odd ratio,OR),检验水准α为0.05。对于多重比较,使用Bonferroni校正P值,本次研究设定P值为0.012(0.05/n,n = 4)。假阳性报告概率(false positive report probability,FPRP)分析[25]用于评估研究结果中假阳性发生的概率。
2. 结果
2.1 临床特征
本研究共纳入958名受试者,包括478名NSCLC患者,平均年龄为(55.94 ± 10.79)岁,其中男性305名,女性173名。健康对照组包含480名受试者,平均年龄为(55.16 ± 11.35)岁,其中男性277名,女性203名。2组受试者之间的年龄和性别分布比较差异无统计学意义(t = 1.09,P = 0.275; χ2 = 3.74,P = 0.053),见表2。
表 2 受试者临床信息[$ x \pm s $/n(%)]Table 2. The clinical characteristics of the subjects enrolled in the current study [$ x \pm s $/n(%)]项目 肺癌组 对照组 t/χ2 P 样本数 478 480 年龄(岁) 55.94 ± 10.79 55.16 ± 11.35 1.09 0.275 性别 男 305(63.8) 277(57.7) 3.74 0.053 女 173(36.2) 203(42.3) 病理类型 腺癌 327(68.4) 8.00 < 0.001* 鳞状细胞癌 151(31.6) 临床分期 Ⅰ期 126(26.3) 26.45 < 0.001* Ⅱ期 74(15.5) Ⅲ期 151(31.6) Ⅳ期 127(26.6) 2.2 4个SNP与肺癌的相关性分析
位于ERK1/2信号通路相关基因3'UTR区域的4个SNP的基因型在对照组中的分布特征均符合HWE(P > 0.05),表明本研究纳入样本是具有群体代表性的随机样本。对4个SNP位点等位基因和基因型在对照组与NSCLC组中分布频率的差异进行分析,结果显示,4个SNP的等位基因和基因型在对照组与NSCLC组中分布频率的差异经Bonferroni校正后,差异无统计学意义(P > 0.012),见表3。
表 3 在健康对照组和NSCLC组中4个SNP位点等位基因及基因型频率分布结果[n(%)]Table 3. Allele and genotype frequencies of four SNPs between the control group and NSCLC group [n(%)]SNPs 等位基因/基因型 对照组 肺癌组 对照组HWE 肺癌组 vs 对照组 χ2 P OR (95%CI) χ2 P rs9340 G 773(80.5) 728(76.2) 0.77(0.62~0.96) 5.39 0.020 A 187(19.5) 228(23.8) G/G 308(64.2) 273(57.1) 0.87 0.350 5.63 0.060 G/A 157(32.7) 182(38.1) A/A 15(3.1) 23(4.8) rs14804 G 936(97.5) 935(97.8) 1.14(0.63~2.06) 0.19 0.661 A 24(2.5) 21(2.2) G/G 456(95.0) 458(95.8) 0.32 0.574 1.58 0.454 G/A 24(5.0) 19(4.0) A/A 0 1(0.2) rs712 A 197(20.5) 202(21.1) 1.04(0.83~1.29) 1.04 0.743 C 763(79.5) 754(78.9) A/A 21(4.4) 29(6.1) 0.05 0.826 1.68 0.431 A/C 155(32.3) 144(30.1) C/C 304(63.3) 305(63.8) rs7973450 A 873(90.9) 876(91.6) 1.09(0.79~1.50) 1.09 0.590 G 87(9.1) 80(8.4) A/A 399(83.1) 401(83.9) 1.30 0.254 1.01 0.604 A/G 75(15.6) 74(15.5) G/G 6(1.3) 3(0.6) HWE,哈迪温伯格平衡。*P < 0.012(经Bonferroni校正,n = 4)。 2.3 4个SNP与不同病理类型的肺癌相关性分析
为了探究ERK1/2信号通路相关基因3'UTR区域的4个SNP与不同病理类型NSCLC的相关性,本研究按照病理类型将NSCLC组分为腺癌组(adenocarcinoma,AC)和鳞状细胞癌组(squamous cell carcinoma,SCC),并分析4个SNP位点的等位基因和基因型在对照组与AC组以及对照组与SCC组中分布频率的差异。结果显示,rs9340位点的等位基因在对照组与SCC组中分布频率的差异具有统计学意义(P = 0.009),在先验概率水平为0.25时,rs9340与SCC的相关性仍有意义(FPRP = 0.045),该结果表明rs9340位点的G等位基因可能是非小细胞肺鳞癌的保护性因素(OR = 0.67,95% CI: 0.50~0.91),而其余位点的等位基因和基因型在对照组与各病理类型组间分布频率的差异经Bonferroni校正后均无统计学意义(P > 0.012),见表4。
表 4 在健康对照组和不同病理类型肺癌组中4个SNP位点等位基因及基因型频率分布结果[n(%)]Table 4. Allele and genotype frequencies of four SNPs between different pathological types of NSCLC [n(%)]SNPs 等位基因/基因型 对照组 AC组 SCC组 AC组 vs 对照组 SCC组 vs 对照组 SCC组 vs AC组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 773(80.5) 506(77.4) 222(73.5) 0.83(0.65~1.05) 2.35 0.125 0.67(0.50~0.91) 6.77 0.009* 0.81(0.59~1.11) 1.70 0.193 A 187(19.5) 148(22.6) 80(26.5) G/G 308(64.2) 192(58.7) 81(53.7) 2.53 0.282 7.26 0.027 2.13 0.345 G/A 157(32.7) 122(37.3) 60(39.7) A/A 15(3.1) 13(4.0) 10(6.6) rs14804 G 936(97.5) 640(97.9) 295(97.7) 1.17(0.60~2.28) 0.22 0.640 1.08(0.46~2.53) 0.03 0.858 0.92(0.37~2.31) 0.03 0.862 A 24(2.5) 14(2.1) 7(2.3) G/G 456(95.0) 313(95.7) 145(96.0) 0.22 0.636 3.90 0.142 2.41 0.300 G/A 24(5.0) 14(4.3) 5(3.3) A/A 0 0 1(0.7) rs712 A 197(20.5) 140(21.4) 62(20.5) 1.05(0.83~1.35) 0.18 0.667 1.00(0.73~1.38) 1.10 × 10−5 0.997 0.95(0.68~1.33) 0.09 0.758 C 763(79.5) 514(78.6) 240(79.5) A/A 21(4.4) 20(6.1) 9(6.0) 1.34 0.512 1.01 0.602 0.12 0.943 A/C 155(32.3) 100(30.6) 44(29.1) C/C 304(63.3) 207(63.3) 98(64.9) rs7973450 A 873(90.9) 597(91.3) 279(92.4) 1.04(0.74~1.48) 0.06 0.810 1.21(0.75~1.95) 0.60 0.437 1.16(0.70~1.92) 0.32 0.568 G 87(9.1) 57(8.7) 23(7.6) A/A 399(83.1) 273(83.5) 128(84.8) 0.20 0.907 1.94 0.380 1.41 0.493 A/G 75(15.6) 51(15.6) 23(15.2) G/G 6(1.3) 3(0.9) 0 AC,肺腺癌;SCC,肺鳞状细胞癌。*P < 0.012(经Bonferroni校正,n = 4)。 2.4 4个SNP与不同分期的肺癌相关性分析
为分析ERK1/2信号通路相关基因3'UTR区域的4个SNP位点与NSCLC不同临床分期的相关性,本研究按临床分期将NSCLC组分为I+II期组以及III+IV期组,并分析4个SNP位点等位基因和基因型在对照组与I+II期组以及对照组与III+IV期组中分布频率的差异。结果显示,4个SNP位点的等位基因和基因型在对照组与I+II期组以及对照组与III+IV期组中分布频率的差异经Bonferroni校正后差异无统计学意义(P > 0.012),见表5。
表 5 健康对照组和不同分期肺癌组中4个SNP位点等位基因及基因型频率分布结果[n(%)]Table 5. Allele and genotype frequencies of four SNPs between different stages of NSCLC [n(%)]SNPs 等位基因/基因型 对照组 I+II期组 III+IV期组 I+II期组 vs 对照组 III+IV期组 vs 对照组 III+IV期组 vs I+II期组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 773(80.5) 303(75.7) 425(76.4) 0.76(0.57~1.00) 3.89 0.049 0.78(0.61~1.01) 3.54 0.060 1.04(0.77~1.40) 0.06 0.805 A 187(19.5) 97(24.3) 131(23.6) G/G 308(64.2) 114(57.0) 159(57.2) 4.20 0.122 3.78 0.151 0.37 0.831 G/A 157(32.7) 75(37.5) 107(38.5) A/A 15(3.1) 11(5.5) 12(4.3) rs14804 G 936(97.5) 394(98.5) 541(97.3) 1.68(0.68~4.15) 1.31 0.253 0.92(0.48~1.78) 0.05 0.815 0.55(0.21~1.43) 1.55 0.213 A 24(2.5) 6(1.5) 15(2.7) G/G 456(95.0) 194(97.0) 264(95.0) 1.34 0.247 1.76 0.414 1.59 0.451 G/A 24(5.0) 6(3.0) 13(4.6) A/A 0 0 1(0.4) rs712 A 197(20.5) 79(19.8) 123(22.1) 0.95(0.71~1.28) 0.10 0.747 1.10(0.85~1.42) 0.54 0.462 1.15(0.84~1.59) 0.79 0.375 C 763(79.5) 321(80.2) 433(77.9) A/A 21(4.4) 9(4.5) 20(7.2) 0.21 0.901 2.92 0.232 1.48 0.476 A/C 155(32.3) 61(30.5) 83(29.9) C/C 304(63.3) 130(65.0) 175(62.9) rs7973450 A 873(90.9) 370(92.5) 506(91.0) 1.23(0.80~1.89) 0.88 0.349 1.01(0.70~1.45) 2.08 × 10−3 0.964 0.82(0.51~1.32) 0.68 0.411 G 87(9.1) 30(7.5) 50(9.0) A/A 399(83.1) 173(86.5) 228(82.0) 1.53 0.465 4.10 0.129 7.14 0.028 A/G 75(15.6) 24(12.0) 50(18.0) G/G 6(1.3) 3(1.5) 0 *P < 0.012(经Bonferroni校正,n = 4)。 2.5 4个SNP在不同年龄段人群中与肺癌的相关性分析
为了分析ERK1/2信号通路相关基因3'UTR区域的4个SNP位点在不同的年龄组中与NSCLC的相关性,本研究分析了4个SNP位点等位基因和基因型在 < 50岁组、50~60岁组以及 > 65岁组中分布频率的差异。结果显示,在 < 50岁年龄组中,rs9340位点的等位基因在对照组和NSCLC组中的分布频率差异具有统计学意义(P = 5.07×10−4),在先验概率水平为0.25时,rs9340与 < 50岁年龄组中NSCLC的相关性仍有意义(FPRP = 2.52 × 10−4),该结果表明rs9340等位基因G可能是NSCLC的保护性因素(OR = 0.46,95% CI: 0.29~0.72),见表6。
表 6 不同年龄组中4个SNP位点等位基因及基因型频率分布结果[n(%)] (1)Table 6. Allele and genotype frequencies of four SNPs between different age groups [n(%)] (1)SNPs 等位基因/
基因型< 50岁组 50~65岁组 > 65岁组 < 50岁
对照组vs肺癌组50~65岁
对照组vs肺癌组> 65岁
对照组vs肺癌组对照组 肺癌组 对照组 肺癌组 对照组 肺癌组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 220(85.9) 196
(73.7)387
(79.3)380
(75.4)166(76.9) 152(81.7) 0.46(0.29~0.72) 12.10 5.07 × 10−4* 0.80(0.59~1.08) 2.16 0.142 1.35
(0.83~2.19)1.43 0.231 A 36(14.1) 70
(26.3)101
(20.7)124
(24.6)50
(23.1)34
(18.3)G/G 92(71.9) 70
(52.6)151
(61.9)144
(57.2)65
(60.2)59
(63.4)14.24 8.15 × 10−4 2.98 0.225 6.26 0.044 G/A 36(28.1) 56
(42.1)85
(34.8)92
(36.5)36
(33.3)34
(36.6)A/A 0 7
(5.3)8
(3.3)16
(6.3)7
(6.5)0 rs14804 G 248(96.9) 260
(97.7)475
(97.3)490
(97.2)213
(98.6)185
(99.5)1.40
(0.48~4.09)0.38 0.539 0.96
(0.45~2.06)0.01 0.912 2.61
(0.27~25.27)0.74 0.391 A 8(3.1) 6
(2.3)13
(2.7)14
(2.8)3
(1.4)1
(0.5)G/G 120(93.8) 127
(95.5)231
(94.7)239
(94.8)105
(97.2)92
(98.9)0.39 0.533 1.04 0.592 0.74 0.389 G/A 8(6.2) 6
(4.5)13
(5.3)12
(4.8)3
(2.8)1
(1.1)A/A 0 0 0 1
(0.4)0
(0.0)0 表 6 不同年龄组中4个SNP位点等位基因及基因型频率分布结果[n(%)] (2)Table 6. Allele and genotype frequencies of four SNPs between different age groups [n(%)] (2)SNPs 等位基因/
基因型< 50岁组 50~65岁组 > 65岁组 < 50岁
对照组vs肺癌组50~65岁
对照组vs肺癌组> 65岁
对照组vs肺癌组对照组 肺癌组 对照组 肺癌组 对照组 肺癌组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs712 A 53(20.7) 50
(18.8)95
(19.5)111
(22.0)49
(22.7)41
(22.0)0.89
(0.58~1.36)0.30 0.584 1.17
(0.86~1.59)0.98 0.321 0.96
(0.60~1.54)0.02 0.878 C 203(79.3) 216
(81.2)393
(80.5)393
(78.0)167
(77.3)145
(78.0)A/A 8(6.3) 4
(3.0)6
(2.5)19
(7.5)7
(6.5)6
(6.4)1.65 0.439 7.35 0.025 0.04 0.982 A/C 37(28.9) 42
(31.6)83
(34.0)73
(29.0)35
(32.4)29
(31.2)C/C 83(64.8) 87
(65.4)155
(63.5)160
(63.5)66
(61.1)58
(62.4)rs7973450 A 233(91.0) 246
(92.5)441
(90.4)459
(91.1)199
(92.1)171
(91.9)1.21
(0.65~2.27)0.37 0.543 1.09
(0.71~1.67)0.15 0.703 0.97
(0.47~2.01)5.14 × 10−3 0.943 G 23(9.0) 20
(7.5)47
(9.6)45
(8.9)17
(7.9)15
(8.1)A/A 108(84.4) 114
(85.7)198
(81.2)209
(82.9)93
(86.1)78
(83.9)1.10 0.578 0.69 0.709 2.35 0.309 A/G 17(13.3) 18
(13.5)45
(18.4)41
(16.3)13
(12.0)15
(16.1)G/G 3(2.3) 1
(0.8)1
(0.4)2
(0.8)2
(1.9)0 *P < 0.012(经Bonferroni校正,n = 4)。 2.6 rs9340功能预测分析
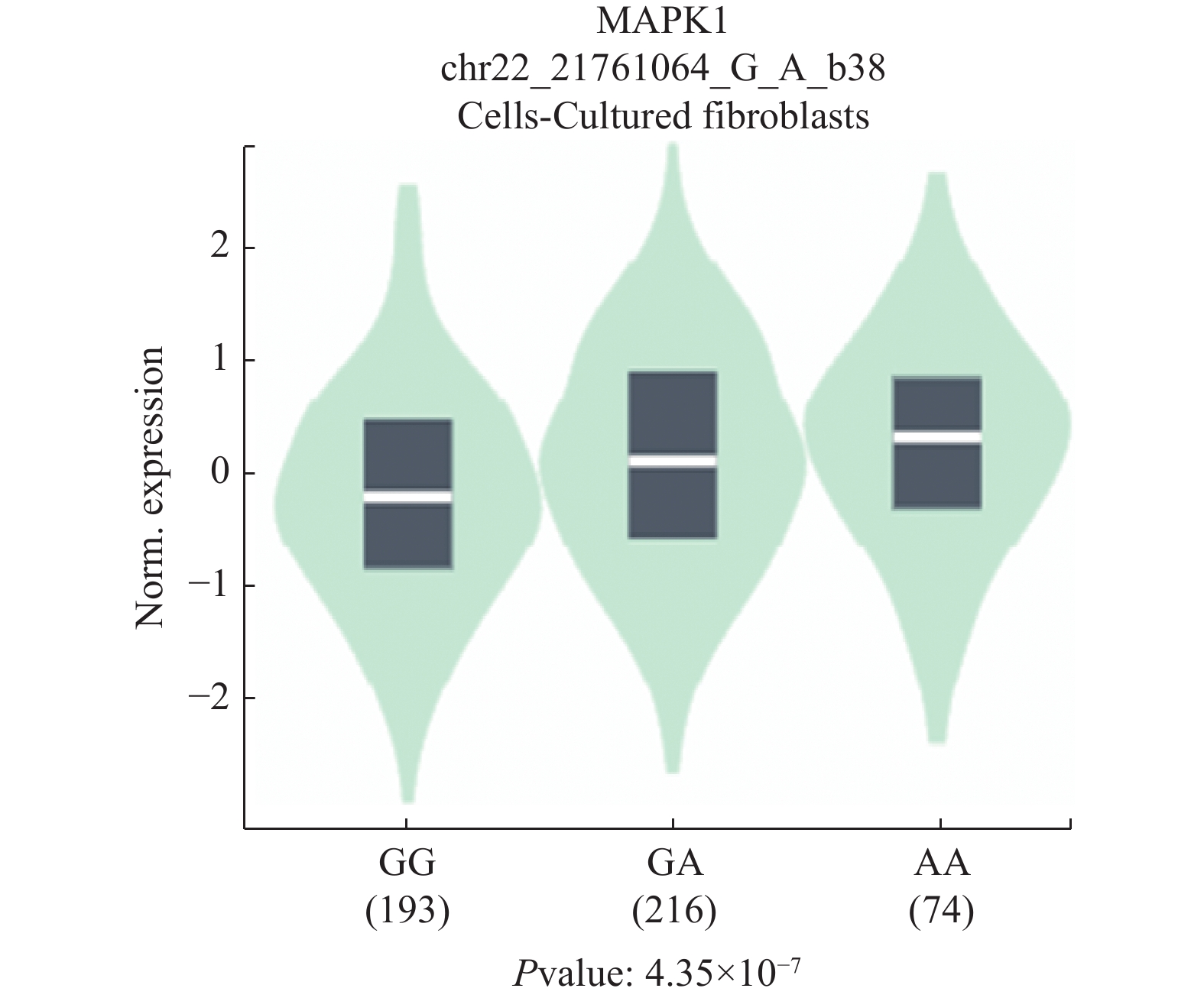
在获得rs9340位点可能与NSCLC发生风险具有相关性的结果后,本研究利用生物信息学工具对rs9340位点的功能进行初步预测,结果显示,rs9340位点的A等位基因可使培养的成纤维细胞中MAPK1基因表达升高,见图1。在miRNA-mRNA相互作用方面,rs9340位点的A等位基因可能在MAPK1基因3'UTR区域增加了与hsa-miR-153-3p、hsa-miR-448的结合位点,同时影响了与hsa-miR-521的结合。该结果表明,rs9340位点可能是通过影响MAPK1基因的表达而在NSCLC的发生过程中发挥作用。
3. 讨论
ERK1/2信号通路参与多个细胞生物学进程的调控,包括增殖、分化和转化[26],异常激活的ERK1/2信号通路可以促进癌细胞的增殖、抑制凋亡、促进血管生成和细胞迁移等,从而促进肿瘤的生长和转移[27-28]。更重要的是,ERK1/2信号通路深度参与癌症治疗中的放射抵抗[29],可能与肿瘤不良预后相关。研究发现,肿瘤相关信号通路基因中的突变在肿瘤发生发展过程中发挥着重要作用[30-31],这些突变通常与信号通路基因表达及功能的改变相关[32-33],其中位于基因3'UTR区域的SNP可能会通过影响miRNA-mRNA的相互作用[34],而影响信号通路基因的表达[35],进而产生级联反应[36],并在疾病的发生发展过程中发挥作用[37]。因此,本研究选取了4个位于ERK1/2信号通路的基因3'UTR区域的SNP位点,研究其与NSCLC发生风险的相关性。
丝裂原活化蛋白激酶1(MAPK1)是研究广泛的MAP激酶家族成员,也被称为细胞外信号调节激酶2(ERK2)[38]。在正常生理条件下,MAPK1在大多数组织中呈中度持续表达,但在乳腺癌、膀胱癌、肺癌和胃癌等多种恶性肿瘤中表达会发生显著变化[39-40]。Wang 等[41]的实验证实,MAPK1可以双向调控靶基因,从而促进胃腺癌细胞的侵袭和迁移。Zhu 等[42]报道,MAPK1在宫颈癌患者中表达上调,MAPK1逆转了miR-217对细胞活力、迁移、侵袭和凋亡的抑制。Gagliardi 等[43]的研究结果表明,ERK2可以促进三阴性乳腺癌细胞的迁移和侵袭、肿瘤干细胞表型形成和转移。以上研究资料表明MAPK1基因的表达异常在肿瘤发生中发挥重要作用。而rs9340位点位于MAPK1基因的3'UTR,可能参与MAPK1的转录后调控,而与疾病的发生相关。Guo 等[44]对MAPK1基因中的rs9340的研究结果显示,rs9340位点的G等位基因与降低围绝经期冠心病的易感性显著相关。Insodaite 等[45]的结果表明,rs9340位点基因型A/A是喉鳞癌患者发生远处转移的风险因素。同样,本研究结果显示rs9340可能与非小细胞肺鳞癌的发生风险具有相关性,其中等位基因G可能是非小细胞肺鳞癌的保护性因素。本研究生物信息学相关分析结果提示rs9340位点可能会影响miRNA与MAPK1基因3'UTR 区域的相互作用,并与MAPK1基因的表达相关,这可能是该位点与非小细胞肺鳞癌发生风险相关的机制。非小细胞肺鳞癌与其他类型肺癌相比,预后不理想,并且缺乏可用的靶向药物,往往对免疫治疗的敏感性较低[20,46-47],结合本研究的发现,未来应继续在更大规模人群中分析rs9340位点与非小细胞肺鳞癌的相关性,并通过功能研究探究相关机制,为该位点在非小细胞肺鳞癌早期诊断及预后分析中的应用提供必要的指导。
此外,本研究结果还显示,在 < 50岁人群中,rs9340位点与NSCLC发生风险相关。肺癌是全球及我国60岁以上人群发病率及死亡率最高的恶性肿瘤[48],考虑到我国肺癌发病年龄较早,《中国肺癌筛查与早诊早治指南》建议我国的肺癌筛查起始年龄为50岁[21]。因此,未来有必要开展更多研究探讨MAPK1基因3'UTR 区域的rs9340位点在50岁以下人群的NSCLC风险预测中的应用。
综上所述,本研究分析了4个位于ERK1/2信号通路基因3'UTR区域的SNP位点与NSCLC发生风险的相关性。研究结果初步揭示了位于MAPK1基因3'UTR 区域的rs9340位点与NSCLC发生风险的相关性及其可能的机制,未来应在此基础上进一步开展更大样本量的相关性研究以及功能研究,阐明该位点在肺癌发生过程中发挥的作用及其机制,为该位点在肺癌的早期诊断及治疗中的应用提供研究基础。
-
表 1 所选SNP位点信息
Table 1. The information of selected SNPs in the current study
SNPs 基因 功能 位置 等位基因 中国南方汉族人群MAF rs9340 MAPK1 3'UTR突变 Chr 22:21761064 G > A 0.21 rs14804 NRAS 3'UTR突变 Chr 1:114707222 G > A 0.04 rs712 KRAS 3'UTR突变 Chr 12:25209618 A > C 0.48 rs7973450 KRAS 3'UTR突变 Chr 12:25208208 A > G 0.09 SNP,单核苷酸多态性;MAF,次要等位基因频率;Chr,染色体。 表 2 受试者临床信息[$ x \pm s $/n(%)]
Table 2. The clinical characteristics of the subjects enrolled in the current study [$ x \pm s $/n(%)]
项目 肺癌组 对照组 t/χ2 P 样本数 478 480 年龄(岁) 55.94 ± 10.79 55.16 ± 11.35 1.09 0.275 性别 男 305(63.8) 277(57.7) 3.74 0.053 女 173(36.2) 203(42.3) 病理类型 腺癌 327(68.4) 8.00 < 0.001* 鳞状细胞癌 151(31.6) 临床分期 Ⅰ期 126(26.3) 26.45 < 0.001* Ⅱ期 74(15.5) Ⅲ期 151(31.6) Ⅳ期 127(26.6) 表 3 在健康对照组和NSCLC组中4个SNP位点等位基因及基因型频率分布结果[n(%)]
Table 3. Allele and genotype frequencies of four SNPs between the control group and NSCLC group [n(%)]
SNPs 等位基因/基因型 对照组 肺癌组 对照组HWE 肺癌组 vs 对照组 χ2 P OR (95%CI) χ2 P rs9340 G 773(80.5) 728(76.2) 0.77(0.62~0.96) 5.39 0.020 A 187(19.5) 228(23.8) G/G 308(64.2) 273(57.1) 0.87 0.350 5.63 0.060 G/A 157(32.7) 182(38.1) A/A 15(3.1) 23(4.8) rs14804 G 936(97.5) 935(97.8) 1.14(0.63~2.06) 0.19 0.661 A 24(2.5) 21(2.2) G/G 456(95.0) 458(95.8) 0.32 0.574 1.58 0.454 G/A 24(5.0) 19(4.0) A/A 0 1(0.2) rs712 A 197(20.5) 202(21.1) 1.04(0.83~1.29) 1.04 0.743 C 763(79.5) 754(78.9) A/A 21(4.4) 29(6.1) 0.05 0.826 1.68 0.431 A/C 155(32.3) 144(30.1) C/C 304(63.3) 305(63.8) rs7973450 A 873(90.9) 876(91.6) 1.09(0.79~1.50) 1.09 0.590 G 87(9.1) 80(8.4) A/A 399(83.1) 401(83.9) 1.30 0.254 1.01 0.604 A/G 75(15.6) 74(15.5) G/G 6(1.3) 3(0.6) HWE,哈迪温伯格平衡。*P < 0.012(经Bonferroni校正,n = 4)。 表 4 在健康对照组和不同病理类型肺癌组中4个SNP位点等位基因及基因型频率分布结果[n(%)]
Table 4. Allele and genotype frequencies of four SNPs between different pathological types of NSCLC [n(%)]
SNPs 等位基因/基因型 对照组 AC组 SCC组 AC组 vs 对照组 SCC组 vs 对照组 SCC组 vs AC组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 773(80.5) 506(77.4) 222(73.5) 0.83(0.65~1.05) 2.35 0.125 0.67(0.50~0.91) 6.77 0.009* 0.81(0.59~1.11) 1.70 0.193 A 187(19.5) 148(22.6) 80(26.5) G/G 308(64.2) 192(58.7) 81(53.7) 2.53 0.282 7.26 0.027 2.13 0.345 G/A 157(32.7) 122(37.3) 60(39.7) A/A 15(3.1) 13(4.0) 10(6.6) rs14804 G 936(97.5) 640(97.9) 295(97.7) 1.17(0.60~2.28) 0.22 0.640 1.08(0.46~2.53) 0.03 0.858 0.92(0.37~2.31) 0.03 0.862 A 24(2.5) 14(2.1) 7(2.3) G/G 456(95.0) 313(95.7) 145(96.0) 0.22 0.636 3.90 0.142 2.41 0.300 G/A 24(5.0) 14(4.3) 5(3.3) A/A 0 0 1(0.7) rs712 A 197(20.5) 140(21.4) 62(20.5) 1.05(0.83~1.35) 0.18 0.667 1.00(0.73~1.38) 1.10 × 10−5 0.997 0.95(0.68~1.33) 0.09 0.758 C 763(79.5) 514(78.6) 240(79.5) A/A 21(4.4) 20(6.1) 9(6.0) 1.34 0.512 1.01 0.602 0.12 0.943 A/C 155(32.3) 100(30.6) 44(29.1) C/C 304(63.3) 207(63.3) 98(64.9) rs7973450 A 873(90.9) 597(91.3) 279(92.4) 1.04(0.74~1.48) 0.06 0.810 1.21(0.75~1.95) 0.60 0.437 1.16(0.70~1.92) 0.32 0.568 G 87(9.1) 57(8.7) 23(7.6) A/A 399(83.1) 273(83.5) 128(84.8) 0.20 0.907 1.94 0.380 1.41 0.493 A/G 75(15.6) 51(15.6) 23(15.2) G/G 6(1.3) 3(0.9) 0 AC,肺腺癌;SCC,肺鳞状细胞癌。*P < 0.012(经Bonferroni校正,n = 4)。 表 5 健康对照组和不同分期肺癌组中4个SNP位点等位基因及基因型频率分布结果[n(%)]
Table 5. Allele and genotype frequencies of four SNPs between different stages of NSCLC [n(%)]
SNPs 等位基因/基因型 对照组 I+II期组 III+IV期组 I+II期组 vs 对照组 III+IV期组 vs 对照组 III+IV期组 vs I+II期组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 773(80.5) 303(75.7) 425(76.4) 0.76(0.57~1.00) 3.89 0.049 0.78(0.61~1.01) 3.54 0.060 1.04(0.77~1.40) 0.06 0.805 A 187(19.5) 97(24.3) 131(23.6) G/G 308(64.2) 114(57.0) 159(57.2) 4.20 0.122 3.78 0.151 0.37 0.831 G/A 157(32.7) 75(37.5) 107(38.5) A/A 15(3.1) 11(5.5) 12(4.3) rs14804 G 936(97.5) 394(98.5) 541(97.3) 1.68(0.68~4.15) 1.31 0.253 0.92(0.48~1.78) 0.05 0.815 0.55(0.21~1.43) 1.55 0.213 A 24(2.5) 6(1.5) 15(2.7) G/G 456(95.0) 194(97.0) 264(95.0) 1.34 0.247 1.76 0.414 1.59 0.451 G/A 24(5.0) 6(3.0) 13(4.6) A/A 0 0 1(0.4) rs712 A 197(20.5) 79(19.8) 123(22.1) 0.95(0.71~1.28) 0.10 0.747 1.10(0.85~1.42) 0.54 0.462 1.15(0.84~1.59) 0.79 0.375 C 763(79.5) 321(80.2) 433(77.9) A/A 21(4.4) 9(4.5) 20(7.2) 0.21 0.901 2.92 0.232 1.48 0.476 A/C 155(32.3) 61(30.5) 83(29.9) C/C 304(63.3) 130(65.0) 175(62.9) rs7973450 A 873(90.9) 370(92.5) 506(91.0) 1.23(0.80~1.89) 0.88 0.349 1.01(0.70~1.45) 2.08 × 10−3 0.964 0.82(0.51~1.32) 0.68 0.411 G 87(9.1) 30(7.5) 50(9.0) A/A 399(83.1) 173(86.5) 228(82.0) 1.53 0.465 4.10 0.129 7.14 0.028 A/G 75(15.6) 24(12.0) 50(18.0) G/G 6(1.3) 3(1.5) 0 *P < 0.012(经Bonferroni校正,n = 4)。 表 6 不同年龄组中4个SNP位点等位基因及基因型频率分布结果[n(%)] (1)
Table 6. Allele and genotype frequencies of four SNPs between different age groups [n(%)] (1)
SNPs 等位基因/
基因型< 50岁组 50~65岁组 > 65岁组 < 50岁
对照组vs肺癌组50~65岁
对照组vs肺癌组> 65岁
对照组vs肺癌组对照组 肺癌组 对照组 肺癌组 对照组 肺癌组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs9340 G 220(85.9) 196
(73.7)387
(79.3)380
(75.4)166(76.9) 152(81.7) 0.46(0.29~0.72) 12.10 5.07 × 10−4* 0.80(0.59~1.08) 2.16 0.142 1.35
(0.83~2.19)1.43 0.231 A 36(14.1) 70
(26.3)101
(20.7)124
(24.6)50
(23.1)34
(18.3)G/G 92(71.9) 70
(52.6)151
(61.9)144
(57.2)65
(60.2)59
(63.4)14.24 8.15 × 10−4 2.98 0.225 6.26 0.044 G/A 36(28.1) 56
(42.1)85
(34.8)92
(36.5)36
(33.3)34
(36.6)A/A 0 7
(5.3)8
(3.3)16
(6.3)7
(6.5)0 rs14804 G 248(96.9) 260
(97.7)475
(97.3)490
(97.2)213
(98.6)185
(99.5)1.40
(0.48~4.09)0.38 0.539 0.96
(0.45~2.06)0.01 0.912 2.61
(0.27~25.27)0.74 0.391 A 8(3.1) 6
(2.3)13
(2.7)14
(2.8)3
(1.4)1
(0.5)G/G 120(93.8) 127
(95.5)231
(94.7)239
(94.8)105
(97.2)92
(98.9)0.39 0.533 1.04 0.592 0.74 0.389 G/A 8(6.2) 6
(4.5)13
(5.3)12
(4.8)3
(2.8)1
(1.1)A/A 0 0 0 1
(0.4)0
(0.0)0 表 6 不同年龄组中4个SNP位点等位基因及基因型频率分布结果[n(%)] (2)
Table 6. Allele and genotype frequencies of four SNPs between different age groups [n(%)] (2)
SNPs 等位基因/
基因型< 50岁组 50~65岁组 > 65岁组 < 50岁
对照组vs肺癌组50~65岁
对照组vs肺癌组> 65岁
对照组vs肺癌组对照组 肺癌组 对照组 肺癌组 对照组 肺癌组 OR(95%CI) χ2 P OR(95%CI) χ2 P OR(95%CI) χ2 P rs712 A 53(20.7) 50
(18.8)95
(19.5)111
(22.0)49
(22.7)41
(22.0)0.89
(0.58~1.36)0.30 0.584 1.17
(0.86~1.59)0.98 0.321 0.96
(0.60~1.54)0.02 0.878 C 203(79.3) 216
(81.2)393
(80.5)393
(78.0)167
(77.3)145
(78.0)A/A 8(6.3) 4
(3.0)6
(2.5)19
(7.5)7
(6.5)6
(6.4)1.65 0.439 7.35 0.025 0.04 0.982 A/C 37(28.9) 42
(31.6)83
(34.0)73
(29.0)35
(32.4)29
(31.2)C/C 83(64.8) 87
(65.4)155
(63.5)160
(63.5)66
(61.1)58
(62.4)rs7973450 A 233(91.0) 246
(92.5)441
(90.4)459
(91.1)199
(92.1)171
(91.9)1.21
(0.65~2.27)0.37 0.543 1.09
(0.71~1.67)0.15 0.703 0.97
(0.47~2.01)5.14 × 10−3 0.943 G 23(9.0) 20
(7.5)47
(9.6)45
(8.9)17
(7.9)15
(8.1)A/A 108(84.4) 114
(85.7)198
(81.2)209
(82.9)93
(86.1)78
(83.9)1.10 0.578 0.69 0.709 2.35 0.309 A/G 17(13.3) 18
(13.5)45
(18.4)41
(16.3)13
(12.0)15
(16.1)G/G 3(2.3) 1
(0.8)1
(0.4)2
(0.8)2
(1.9)0 *P < 0.012(经Bonferroni校正,n = 4)。 -
[1] Sung H,Ferlay J,Siegel R L,et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin,2021,71(3):209-249. doi: 10.3322/caac.21660 [2] Zheng R,Zhang S,Zeng H,et al. Cancer incidence and mortality in China,2016[J]. Journal of the National Cancer Center,2022,2(1):1-9. doi: 10.1016/j.jncc.2022.02.002 [3] Nooreldeen R,Bach H. Current and future development in lung cancer diagnosis[J]. Int J Mol Sci,2021,22(16):8661. doi: 10.3390/ijms22168661 [4] Cao W,Chen H D,Yu Y W,et al. Changing profiles of cancer burden worldwide and in China: A secondary analysis of the global cancer statistics 2020[J]. Chin Med J (Engl),2021,134(7):783-791. doi: 10.1097/CM9.0000000000001474 [5] Saller J J,Boyle T A. Molecular pathology of lung cancer[J]. Cold Spring Harb Perspect Med,2022,12(3):a037812. doi: 10.1101/cshperspect.a037812 [6] Duma N,Santana-Davila R,Molina JR. Non-small cell lung cancer: Epidemiology,screening,diagnosis,and treatment[J]. Mayo Clin Proc,2019,94(8):1623-1640. doi: 10.1016/j.mayocp.2019.01.013 [7] Zhang J,Chen S F,Zhen Y,et al. Multicenter analysis of lung cancer patients younger than 45 years in Shanghai[J]. Cancer,2010,116(15):3656-3662. doi: 10.1002/cncr.25100 [8] Heist R S,Sequist L V,Engelman J A. Genetic changes in squamous cell lung cancer: a review[J]. J Thorac Oncol,2012,7(5):924-933. doi: 10.1097/JTO.0b013e31824cc334 [9] Kim Y,Hammerman P S,Kim J,et al. Integrative and comparative genomic analysis of lung squamous cell carcinomas in East Asian patients[J]. J Clin Oncol,2014,32(2):121-128. doi: 10.1200/JCO.2013.50.8556 [10] Niu Z,Jin R,Zhang Y,et al. Signaling pathways and targeted therapies in lung squamous cell carcinoma: mechanisms and clinical trials[J]. Signal Transduct Target Ther,2022,7(1):353. doi: 10.1038/s41392-022-01200-x [11] Shi Q,Ruan J,Yang Y C,et al. rs66651343 and rs12909095 confer lung cancer risk by regulating CCNDBP1 expression[J]. PLoS One,2023,18(4):e0284347. doi: 10.1371/journal.pone.0284347 [12] Anjum J,Mitra S,Das R,et al. A renewed concept on the MAPK signaling pathway in cancers: polyphenols as a choice of therapeutics[J]. Pharmacol Res,2022,184(2022):106398. [13] Qi M, Elion E A. MAP kinase pathways [J]. J Cell Sci, 2005, 118(Pt 16): 3569-3572. [14] Morrison D K. MAP kinase pathways[J]. Cold Spring Harb Perspect Biol,2012,4(11):a011254. [15] Keshet Y, Seger R. The MAP kinase signaling cascades: A system of hundreds of components regulates a diverse array of physiological functions[M]. //Seger R. MAP kinase signaling protocols. Second Edition. Totowa, NJ: Humana Press, 2010: 3-38. [16] Sinkala M,Nkhoma P,Mulder N,et al. Integrated molecular characterisation of the MAPK pathways in human cancers reveals pharmacologically vulnerable mutations and gene dependencies[J]. Commun Biol,2021,4(1):9. doi: 10.1038/s42003-020-01552-6 [17] Brennecke J,Stark A,Russell RB,et al. Principles of microRNA-target recognition[J]. PLoS Biol,2005,3(3):e85. doi: 10.1371/journal.pbio.0030085 [18] Liu C J,Fu X,Xia M,et al. miRNASNP-v3: a comprehensive database for SNPs and disease-related variations in miRNAs and miRNA targets[J]. Nucleic Acids Research,2021,49(D1):D1276-D1281. doi: 10.1093/nar/gkaa783 [19] Wu W,Wu L,Zhu M,et al. miRNA mediated noise making of 3'UTR mutations in cancer[J]. Genes (Basel),2018,9(11):545. doi: 10.3390/genes9110545 [20] 中华医学会,中华医学会肿瘤学分会,中华医学会杂志社. 中华医学会肺癌临床诊疗指南(2018版)[J]. 中华肿瘤杂志,2018,40(12):30. [21] 赫捷,李霓,陈万青,等. 中国肺癌筛查与早诊早治指南(2021,北京)[J]. 中国肿瘤,2021,30(02):81-111. [22] 黄鼎智, 李琳, 李旭, 等.老年晚期肺癌内科治疗中国专家共识(2022版)[J].中国肺癌杂志, 2022, 25(06): 363-384. [23] Yang J,Yan Z,Wang Y,et al. Association study of relationships of polymorphisms in the miR-21,miR-26b,miR-221/222 and miR-126 genes with cervical intraepithelial neoplasia and cervical cancer[J]. BMC Cancer,2021,21(1):997. doi: 10.1186/s12885-021-08743-2 [24] Shi Y Y,He L. SHEsis,a powerful software platform for analyses of linkage disequilibrium,haplotype construction,and genetic association at polymorphism loci[J]. Cell Res,2005,15(2):97-98. doi: 10.1038/sj.cr.7290272 [25] Lee J,Son M J,Son C Y,et al. Genetic Variation and Autism: A Field Synopsis and Systematic Meta-Analysis[J]. Brain Sci,2020,10(10):692. doi: 10.3390/brainsci10100692 [26] Lake D,Corrêa S A,Müller J. Negative feedback regulation of the ERK1/2 MAPK pathway[J]. Cell Mol Life Sci,2016,73(23):4397-413. doi: 10.1007/s00018-016-2297-8 [27] Balmanno K,Cook S J. Tumour cell survival signalling by the ERK1/2 pathway[J]. Cell Death & Differentiation,2009,16(3):368-377. [28] Yan Z,Ohuchida K,Fei S,et al. Inhibition of ERK1/2 in cancer-associated pancreatic stellate cells suppresses cancer-stromal interaction and metastasis[J]. BioMed Central,2019,38(1):221. [29] Marampon F,Ciccarelli C,Zani B M. Biological rationale for targeting MEK/ERK pathways in anti-cancer therapy and to potentiate tumour responses to radiation[J]. Int J Mol Sci,2019,20(10):2530. doi: 10.3390/ijms20102530 [30] Zhou B,Lin W,Long Y,et al. Notch signaling pathway: architecture,disease,and therapeutics[J]. Signal Transduct Target Ther,2022,7(1):95. doi: 10.1038/s41392-022-00934-y [31] Pino M S,Chung D C. The chromosomal instability pathway in colon cancer[J]. Gastroenterology,2010,138(6):2059-2072. doi: 10.1053/j.gastro.2009.12.065 [32] Müller M F,Ibrahim A E,Arends M J. Molecular pathological classification of colorectal cancer[J]. Virchows Arch,2016,469(2):125-134. doi: 10.1007/s00428-016-1956-3 [33] Ding L,Getz G,Wheeler D A,et al. Somatic mutations affect key pathways in lung adenocarcinoma[J]. Nature,2008,455(7216):1069-1075. doi: 10.1038/nature07423 [34] Hill M,Tran N. miRNA interplay: mechanisms and consequences in cancer[J]. Dis Model Mech,2021,14(4):dmm047662. doi: 10.1242/dmm.047662 [35] Zhu Z,Zhang F,Hu H,et al. Integration of summary data from GWAS and eQTL studies predicts complex trait gene targets[J]. Nat Genet,2016,48(5):481-487. doi: 10.1038/ng.3538 [36] Fabian M R,Sonenberg N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC[J]. Nat Struct Mol Biol,2012,19(6):586-593. doi: 10.1038/nsmb.2296 [37] Chan J J,Tabatabaeian H,Tay Y. 3'UTR heterogeneity and cancer progression[J]. Trends Cell Biol,2023,33(7):568-582. doi: 10.1016/j.tcb.2022.10.001 [38] Sturgill T W,Ray L B,Erikson E,et al. Insulin-stimulated MAP-2 kinase phosphorylates and activates ribosomal protein S6 kinase II[J]. Nature,1988,334(6184):715-718. doi: 10.1038/334715a0 [39] AACR Project GENIE Consortium. AACR project GENIE: powering precision medicine through an international consortium[J]. Cancer Discov,2017,7(8):818-831. doi: 10.1158/2159-8290.CD-17-0151 [40] Rubio K,Romero-Olmedo A J,Sarvari P,et al. Non-canonical integrin signaling activates EGFR and RAS-MAPK-ERK signaling in small cell lung cancer[J]. Theranostics,2023,13(8):2384-2407. doi: 10.7150/thno.79493 [41] Wang Y,Guo Z,Tian Y,et al. MAPK1 promotes the metastasis and invasion of gastric cancer as a bidirectional transcription factor[J]. BMC Cancer,2023,23(1):959. doi: 10.1186/s12885-023-11480-3 [42] Zhu L,Yang S,Wang J. miR-217 inhibits the migration and invasion of HeLa cells through modulating MAPK1[J]. Int J Mol Med,2019,44(5):1824-1832. [43] Gagliardi M,Pitner M K,Park J,et al. Differential functions of ERK1 and ERK2 in lung metastasis processes in triple-negative breast cancer[J]. Sci Rep,2020,10(1):8537. doi: 10.1038/s41598-020-65250-3 [44] Guo N,Zhang N,Yan L,et al. Correlation between genetic polymorphisms within the MAPK1/HIF-1/HO-1 signaling pathway and risk or prognosis of perimenopausal coronary artery disease[J]. Clin Cardiol,2017,40(8):597-604. doi: 10.1002/clc.22708 [45] Insodaite R,Smalinskiene A,Liutkevicius V,et al. Associations of polymorphisms localized in the 3'UTR regions of the KRAS,NRAS,MAPK1 genes with laryngeal squamous cell carcinoma[J]. Genes (Basel),2021,12(11):1679. doi: 10.3390/genes12111679 [46] Hirsch F R,Scagliotti G V,Mulshine J L,et al. Lung cancer: current therapies and new targeted treatments[J]. Lancet,2017,389(10066):299-311. doi: 10.1016/S0140-6736(16)30958-8 [47] Kulasingam V,Diamandis E P. Strategies for discovering novel cancer biomarkers through utilization of emerging technologies[J]. Nat Clin Pract Oncol,2008,5(10):588-599. doi: 10.1038/ncponc1187 [48] Chen W,Zheng R,Baade P D,et al. Cancer statistics in China,2015[J]. CA Cancer J Clin,2016,66(2):115-132. doi: 10.3322/caac.21338 -






 下载:
下载:
 下载:
下载:

