An Analysis of PLA2G1B Expression and Prognosis in Patients with Lung Adenocarcinoma
-
摘要:
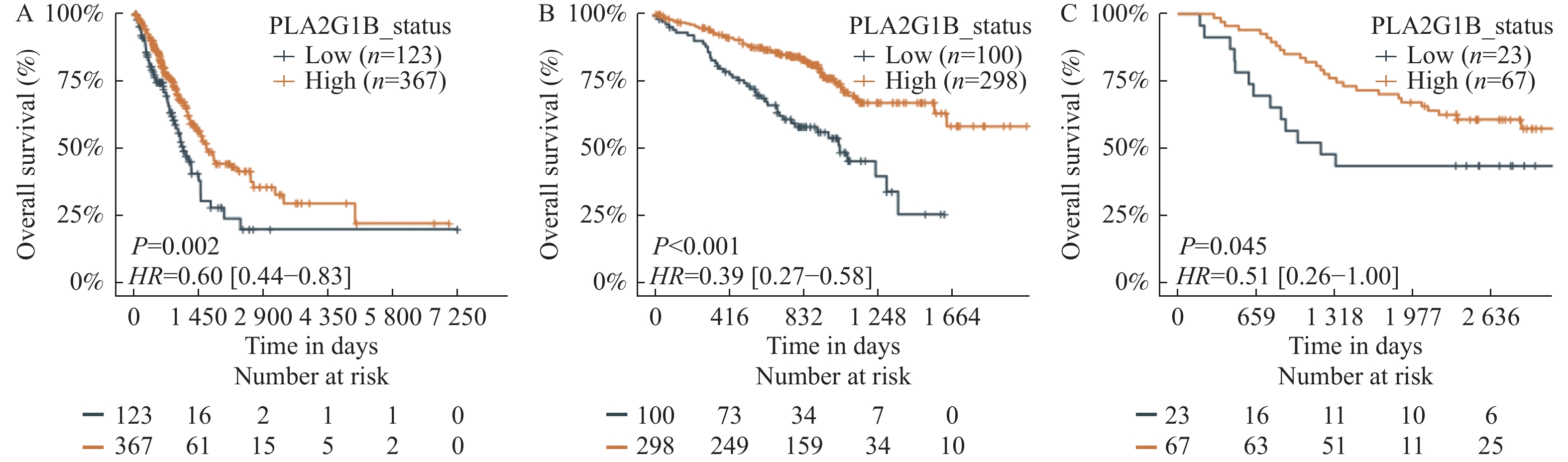
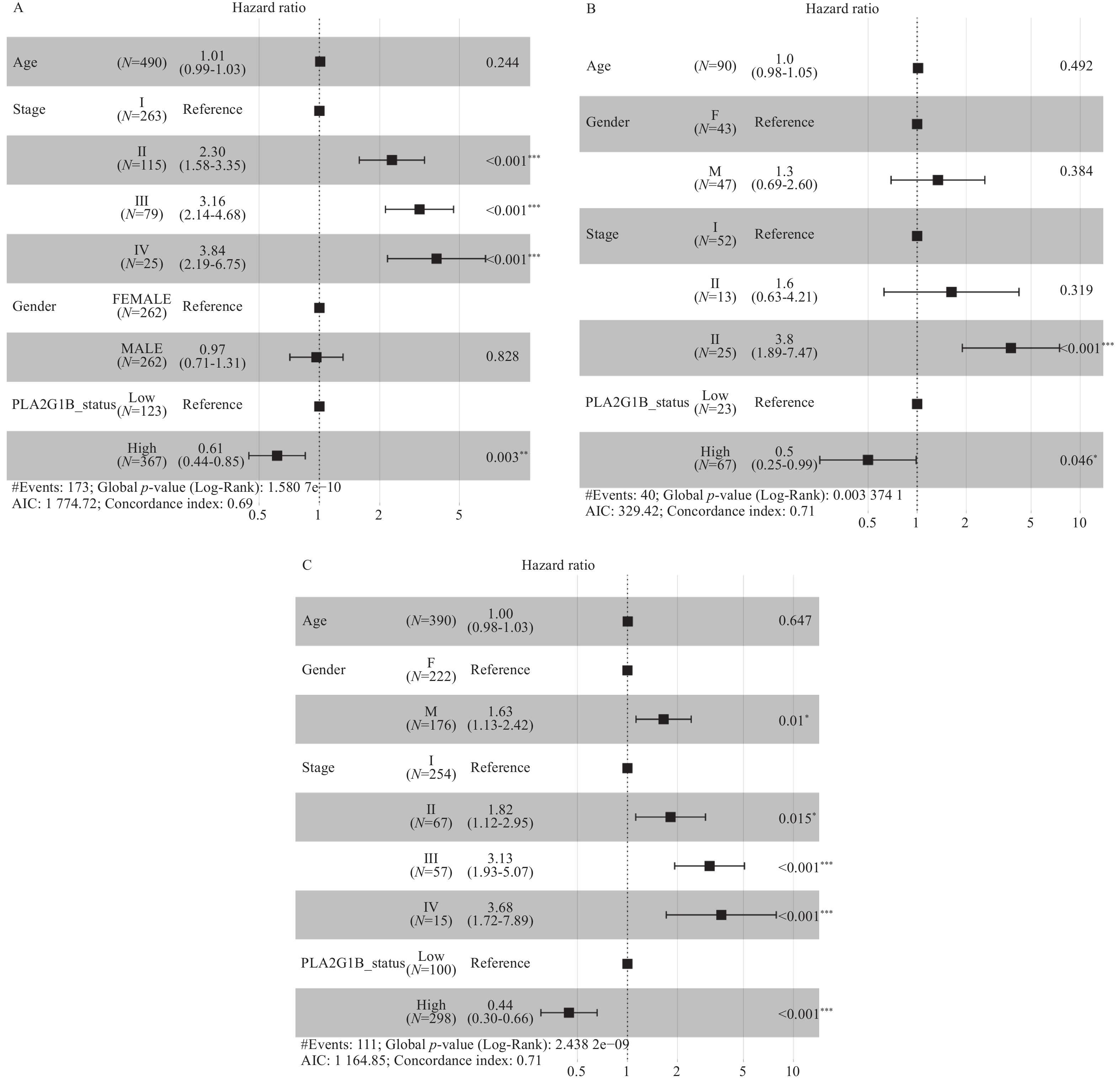
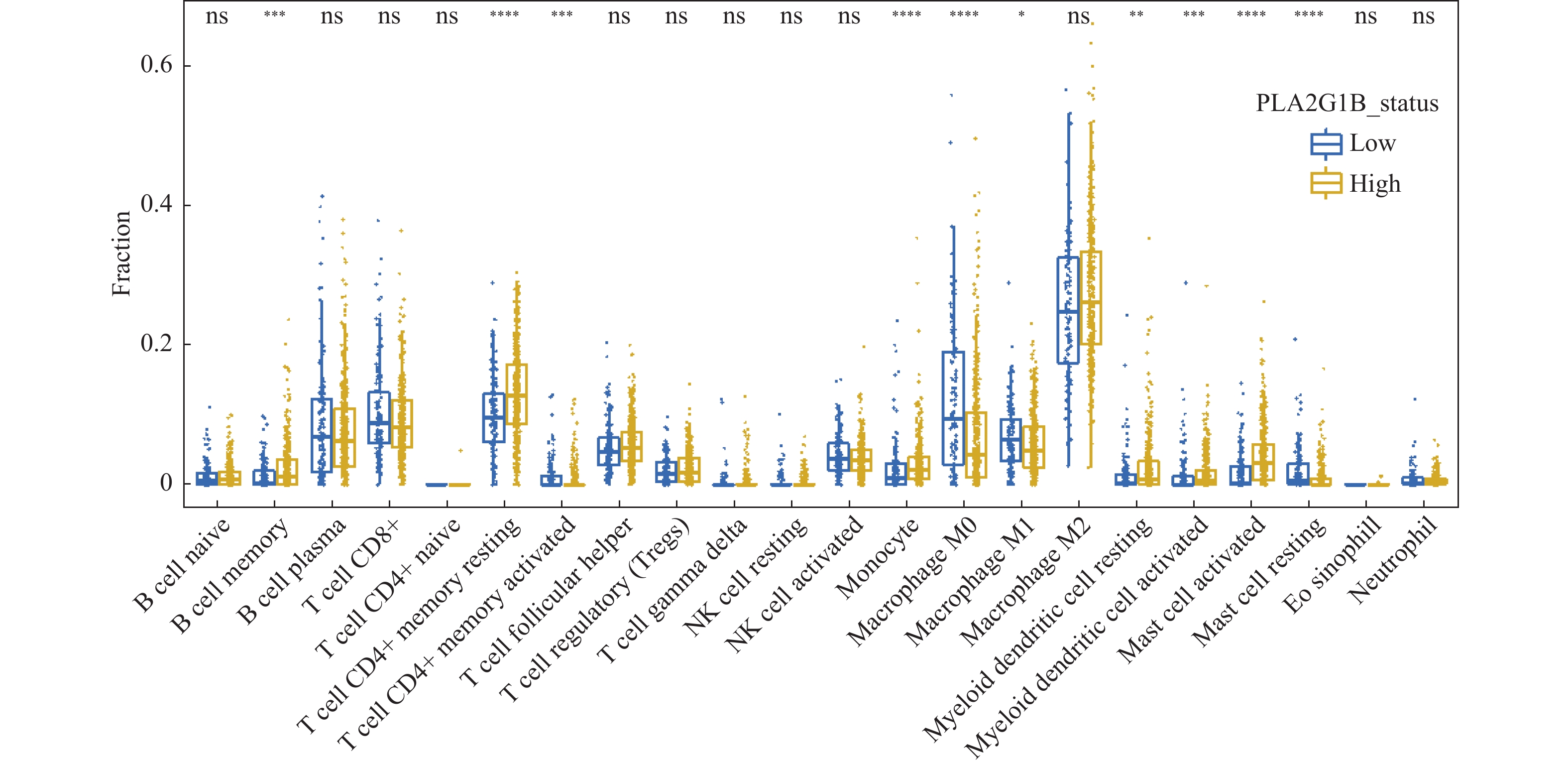
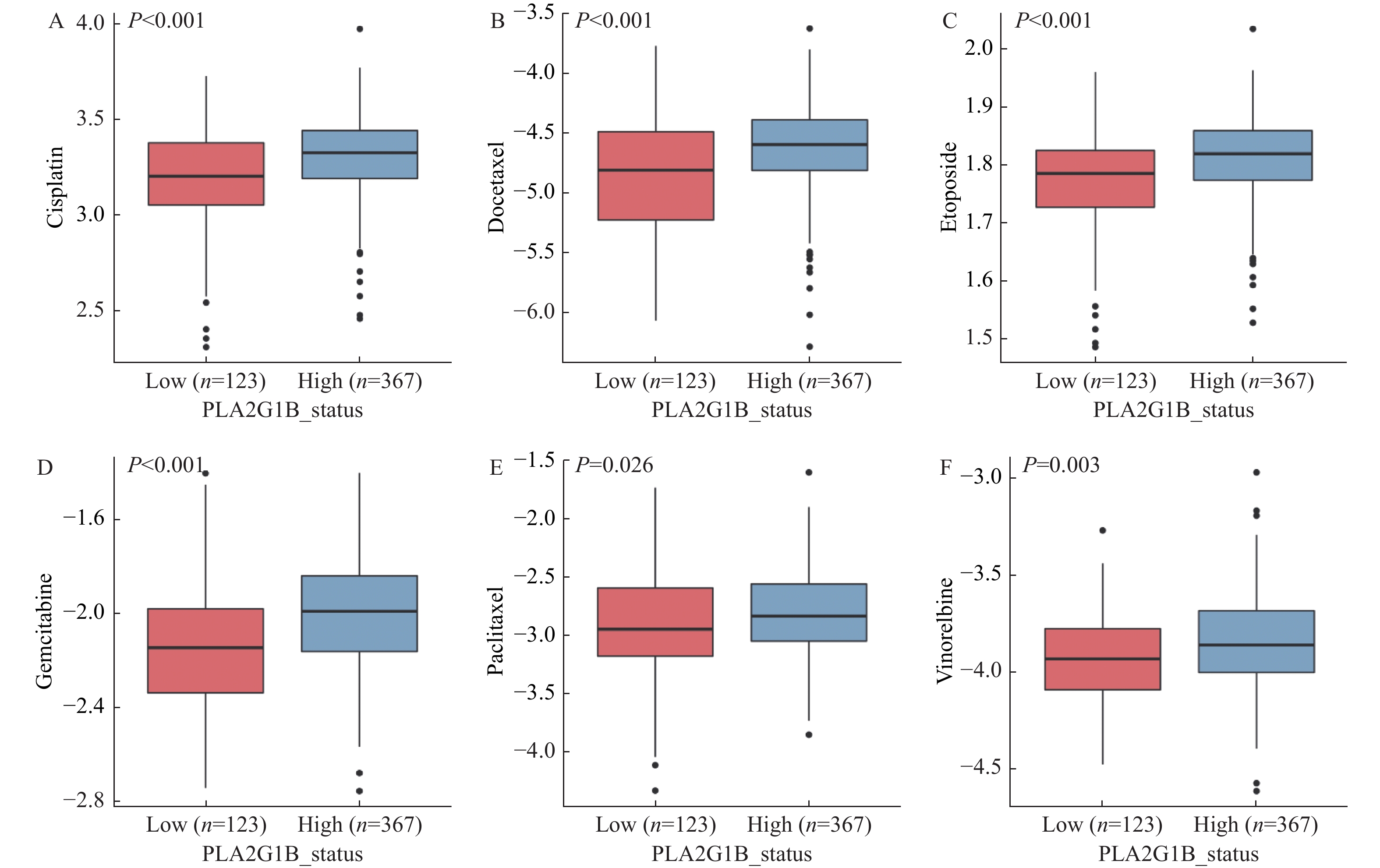
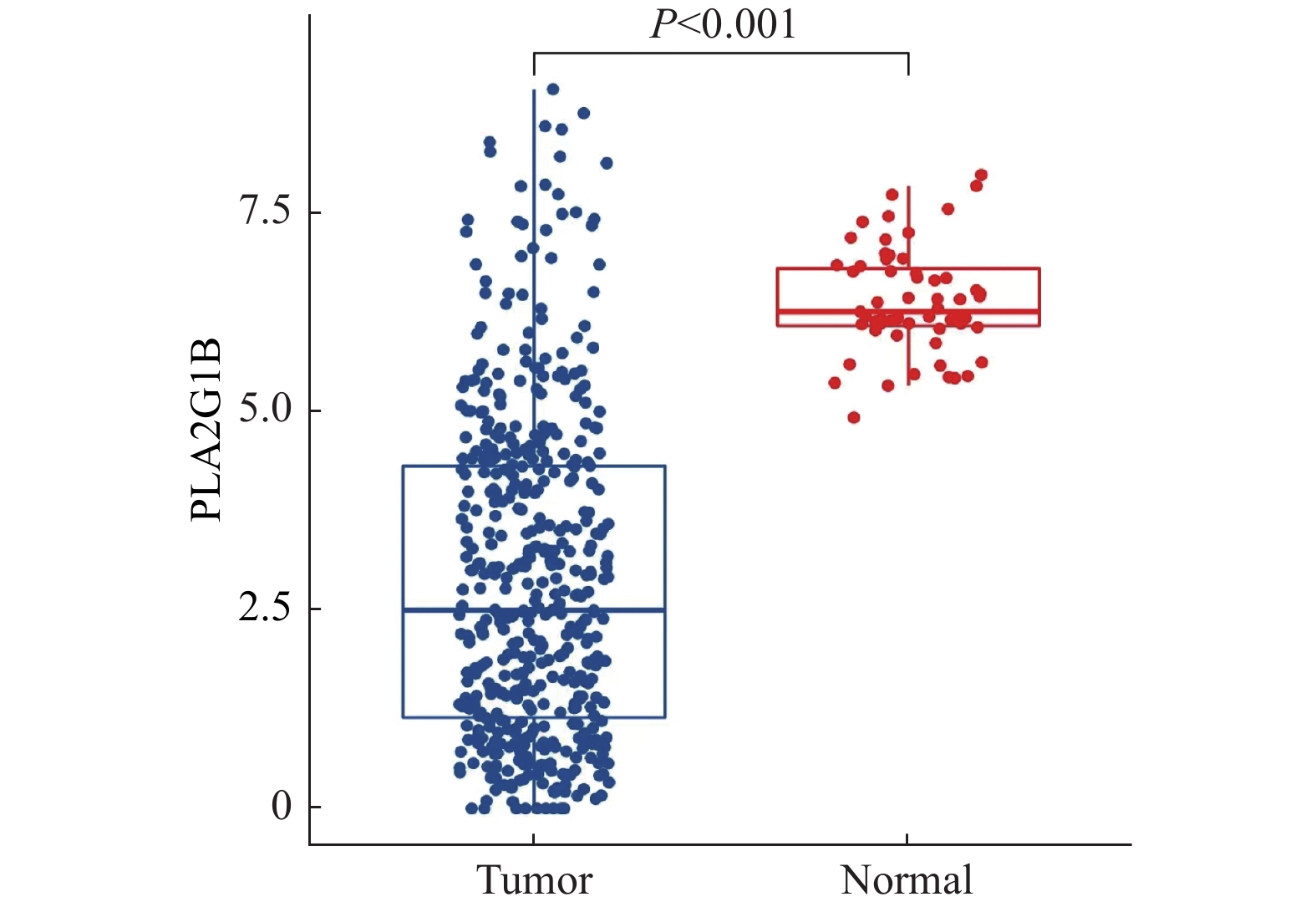
目的 基于数据挖掘分析PLA2G1B基因在肺腺癌患者中的表达特征、预后特征、免疫特征以及潜在的治疗方案影响。 方法 通过对TCGA以及GEO数据库中的肺腺癌患者RNAseq表达谱数据以及临床信息进行分析,对比PLA2G1B高表达和低表达在肺腺癌患者的预后差异,并通过CIBERSORT计算患者的免疫细胞浸润程度,对比PLA2G1B高低表达组之间的浸润程度差异。并通过pRRophetic算法分析2组之间的药物敏感性差异。 结果 PLA2G1B基因在肺腺癌组织中相比癌旁表达量显著降低,且表达量越低其患者的预后总生存越差,在TCGA以及2个GEO的独立验证数据集中均表现出一致的统计学差异,经多因素校正之后证明PLA2G1B低表达为差预后的独立因素(P < 0.05),且在预后较差的样本中其免疫浸润水平更低,主要表现为Bcellmemory,Tcell CD4+ memoryresting,Monocyte,Myeloiddendriticcell,Mastcellactivated细胞类型。但差预后的样本对多种化疗药物的敏感性要显著更好。 结论 PLA2G1B低表达为肺腺癌独立的差预后风险因素,且低表达患者中整体免疫浸润水平显著更低。 Abstract:Objective To analyze PLA2G1B gene expression, prognosis, immunologic features and potential therapeutic effects in patients with lung adenocarcinoma, based on the data-mining of public databases. Methods Data from TCGA and GEO database of LUAD were collected, including gene expression matrix and clinical data. The overall survival (OS) rate was compared between these two groups of PLA2G1B high expressed and low expressed. CIBERSORT algorithm was introduced to calculate totally 22 immune cells infiltration level and their difference was also compared between PLA2G1B high and low groups. Finally, pRRophetic was used to find some drugs that were probably sensitive to the worse OS groups. Results PLA2G1B was down regulated in LUAD samples and the lower expression of PLA2G1B led to a worse OS in LUAD, this result was validated in two independent GEO cohorts. After adjusting three main clinical elements, PLA2G1B was proved to be an independent prognosis biomarker (P < 0.05). For the worse OS group, we also found its immune infiltration level was lower than the better OS group, especially for B cell memory, T cell CD4+ memory resting, Monocyte, Myeloid dendritic cell and Mast activated cells. For the worse OS group, we found they might be more sensitive to several chemo drugs including Cisplatin, Docetaxel, Etoposide, Gemcitabine, Paclitaxeland Vinorelbine. Conclusion PLAG21B is an independent prognosis biomarker in LUAD and the ones low expressed showed worse OS and lower immune infiltration level. -
Key words:
- Lung adenocarcinoma /
- PLA2G1B /
- Prognosis /
- Data mining /
- Immune infiltration
-
根据国际糖尿病联盟的分析,预计到2045年,糖尿病患者的数量将从2030年的5.78亿增加到7.00亿人,这意味着世界各地的公共卫生威胁日益增加[1]。其中2型糖尿病(type 2 diabetes mellitus,T2DM)约占全球所有糖尿病患者的90%, 是一种受遗传和环境风险综合影响的复杂疾病,越来越多的研究表明微生物失调在T2DM 发病机制中的作用[2]。前期研究发现:恒古骨伤愈合剂具有降血糖功效,可改善2型糖尿病db/db小鼠肠道微生态平衡[3]。广谱抗生素引起宿主肠道菌群失调。因此,本实验进一步探索恒古骨伤愈合剂联合广谱抗生素对糖尿病小鼠的作用,为进一步的药物开发提供理论依据。
1. 材料与方法
1.1 实验动物
SPF级10周龄雄性 db/db小鼠和野生型小鼠,购于北京斯贝福公司[SCXK(京)2019-0010],在昆明医科大学[许可证SYXK(滇)2020-0006]饲养:室温(23±2)℃,湿度(52±10)%,明暗交替时间为12 h。小鼠自由采食和饮水。相关操作遵守动物福利要求,试验方案通过伦理委员会批准(KMMU20220903)。
1.2 药物和仪器
恒古骨伤愈合剂(批号:20211211)来自克雷斯制药公司。抗生素(万古霉素,氨苄西林,甲硝唑,新霉素)来自美国Sigma公司。小鼠胰岛素酶联免疫吸附测定试剂盒购自齐一生物科技(上海)公司。血糖检测仪为三诺生物公司制造。
1.3 实验方法
1.3.1 分组与样本采集
适应性饲养2 w后,野生型小鼠为对照组(wt),24只db/db小鼠随机分为模型组(db组)、恒古骨伤愈合剂组(OK组)、广谱抗生素组(ABX组)、恒古骨伤愈合剂与抗生素联合干预组(AO组)(n = 6)。OK组以临床成人常用剂量(按60 kg计算),并按人和动物体表面积换算法折算等效剂量为15.3 g/(kg.2 d),ABX组灌胃等体积广谱抗生素(甲硝唑1 g/L、氨苄西林1 g/L、0.5 g/L 万古霉素和1 g/L新霉素)、对照组、模型组灌胃等体积0.9 %氯化钠溶液,每2 d给药1次。AO组给与恒古骨伤愈合剂与抗生素交替灌胃,共11周。禁食 12 h,测量小鼠体重、空腹血糖,3% 戊巴比妥钠麻醉,采心脏血离心,收集血清检测胰岛素,计算胰岛素抵抗指数(HOMA-IR)。公示为HOMA-IR = 空腹血糖 (mmol/L)× 空腹胰岛素(μU/mL )/ 22.5(校正因子)。取盲结肠内容物0.5 g~1.0 g,分装至无菌 EP 管,过液氮,-80 ℃保存。
1.3.2 肠道菌群16S rDNA测序
由上海百趣公司利用Illumina NovaSeq测序平台测序进行。具体使用Hipure Stool DNA Kits 提取DNA,对16S rDNA V3 + V4区扩增,用AxyPrep DNA Gel Extraction Kit纯化PCR产物,ABI StepOnePlus System定量。原始数据提交SRA数据库,获得ASVs[4],物种注释。并进行菌群多样性分析、差异分析及功能预测。
1.4 统计学处理
采用SPSS 22.0分析数据,计量数据用平均数±标准差表示,组间比较采用单因素方差分析(方差齐) 或秩和检验(方差不齐),多重比较用LSD法或Nemenyi法,P < 0.05为差异有统计学意义。
2. 结果
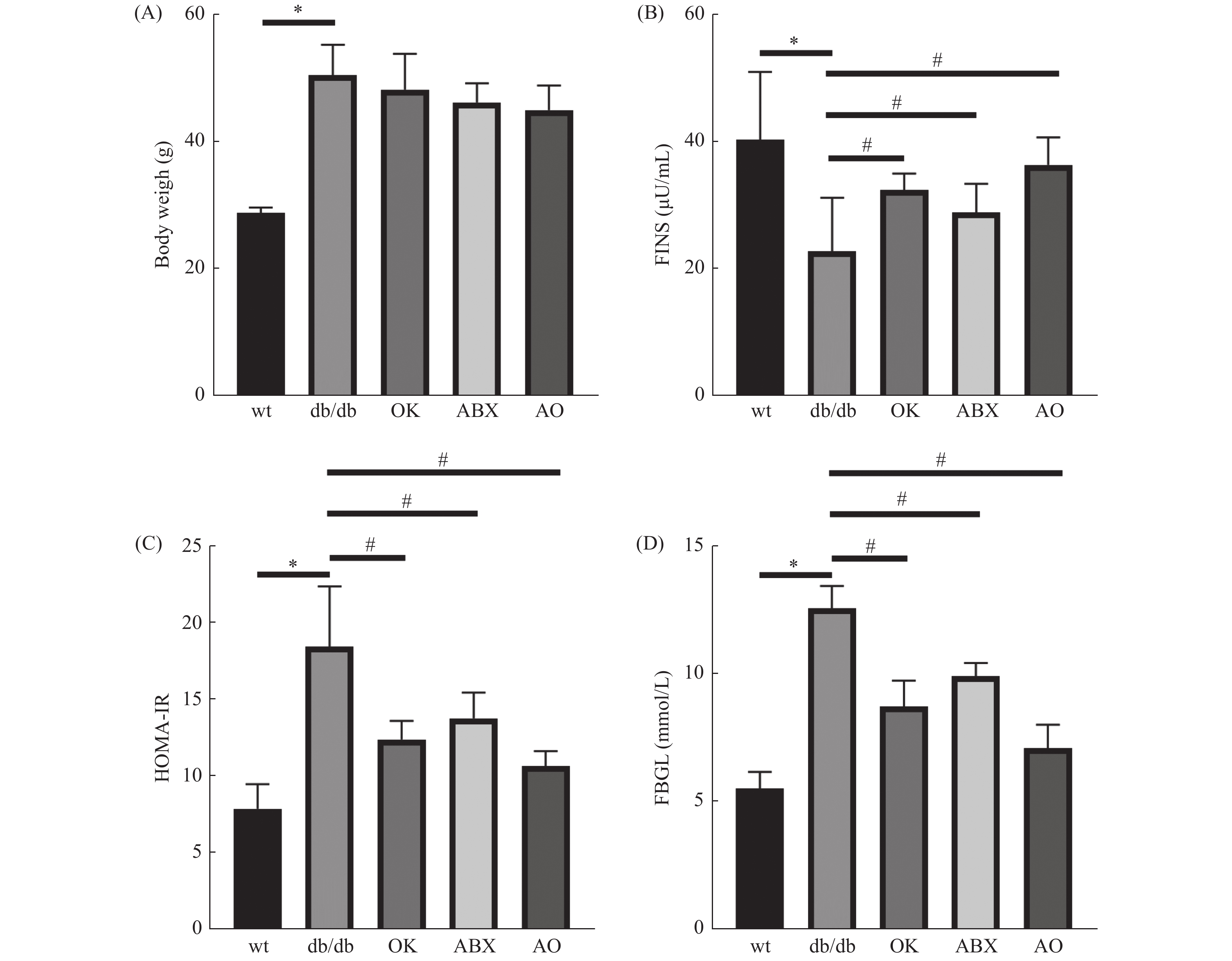
2.1 广谱抗生素、恒古骨伤愈合剂单独和联合使用改善db/db小鼠胰岛素抵抗
与野生型比较,db/db 小鼠体重(图1A)、空腹血糖(图1B)和胰岛素抵抗指数(图1C)升高,血清胰岛素(图1D)含量降低,差异具有统计学意义(P < 0.05),提示db/db 小鼠出现T2DM的病理症状。与模型组(db/db )比较,广谱抗生素组、恒古骨伤愈合剂组和联合用药组空腹血糖和胰岛素抵抗指数降低,血清胰岛素含量升高,差异具有统计学意义( P < 0.05),但各处理组体重与模型组差异无统计学意义( P > 0.05)( 图1A)。
2.2 肠道菌群测序ASVs和韦恩图
5组小鼠韦恩图呈现小鼠间共有、特有的ASVs,其中对照组
1283 个ASVs;ABX、db/db、OK、AO组的ASVs依次增加(824个、878个、946个、1 330个),ABX组肠道菌群ASVs最少而AO组最多。提示抗生素抑制肠道菌群,恒古骨伤愈合剂改善这种抑制作用。2.3 肠道菌群丰富度和均匀性
与对照组wt小鼠比较,db/db组肠道菌群丰富度和均匀性相关指标chao1、shannon、simpson和pielou-e指数降低(P < 0.05)。与db/db组比较,OK组菌群丰富度和均匀性增加( P < 0.05);ABX组菌群丰富度和均匀性最低,差异具有统计学意义( P < 0.01);AO组菌群丰富度和均匀性介于db/db与ABX组之间,其中chao1和simpson指数与db/db组无统计学差异( P > 0.05)。提示恒古骨伤愈合剂改善抗生素作用下的db/db菌群的丰富度和均匀性。各组样本覆盖度大于99.7%,提示样本中的物种均被检测到( 表1)。
表 1 各组小鼠肠道菌群丰富度和均匀性指数( $ \bar x \pm s $)Table 1. Alpha-diversity index of gut microbiota in the 5 groups ( $ \bar x \pm s $)组别 chao1指数 shannon指数 simpson指数 Pielou-e指数 样本覆盖度 对照组(wt) 557.81 ± 78.59 7.16 ± 0.47 0.98 ± 0.0044 0.79 ± 0.039 1.00 模型组(db/db) 363.52 ± 28.91* 5.87 ± 0.64* 0.92 ± 0.0087 *0.69 ± 0.047* 1.00 广谱抗生素组(ABX) 193.04 ± 32.41## 3.70 ± 0.14## 0.84 ± 0.0091 ##0.49 ± 0.018## 1.00 恒古骨伤愈合剂组(OK) 467.39 ± 51.07# 6.84 ± 0.82# 0.96 ± 0.041# 0.78 ± 0.080# 1.00 联合用药组(AO) 311.28 ± 33.87 4.50 ± 0.50# 0.91 ± 0.013 0.59 ± 0.032# 1.00 F 4.01 15.75 13.41 11.69 — P 0.0132 < 0.0001 < 0.0001 < 0.0001 — 与对照组比较,*P < 0.05;与模型组比较, #P < 0.05, ##P < 0.01。 2.4 门与科水平的肠道菌群丰度
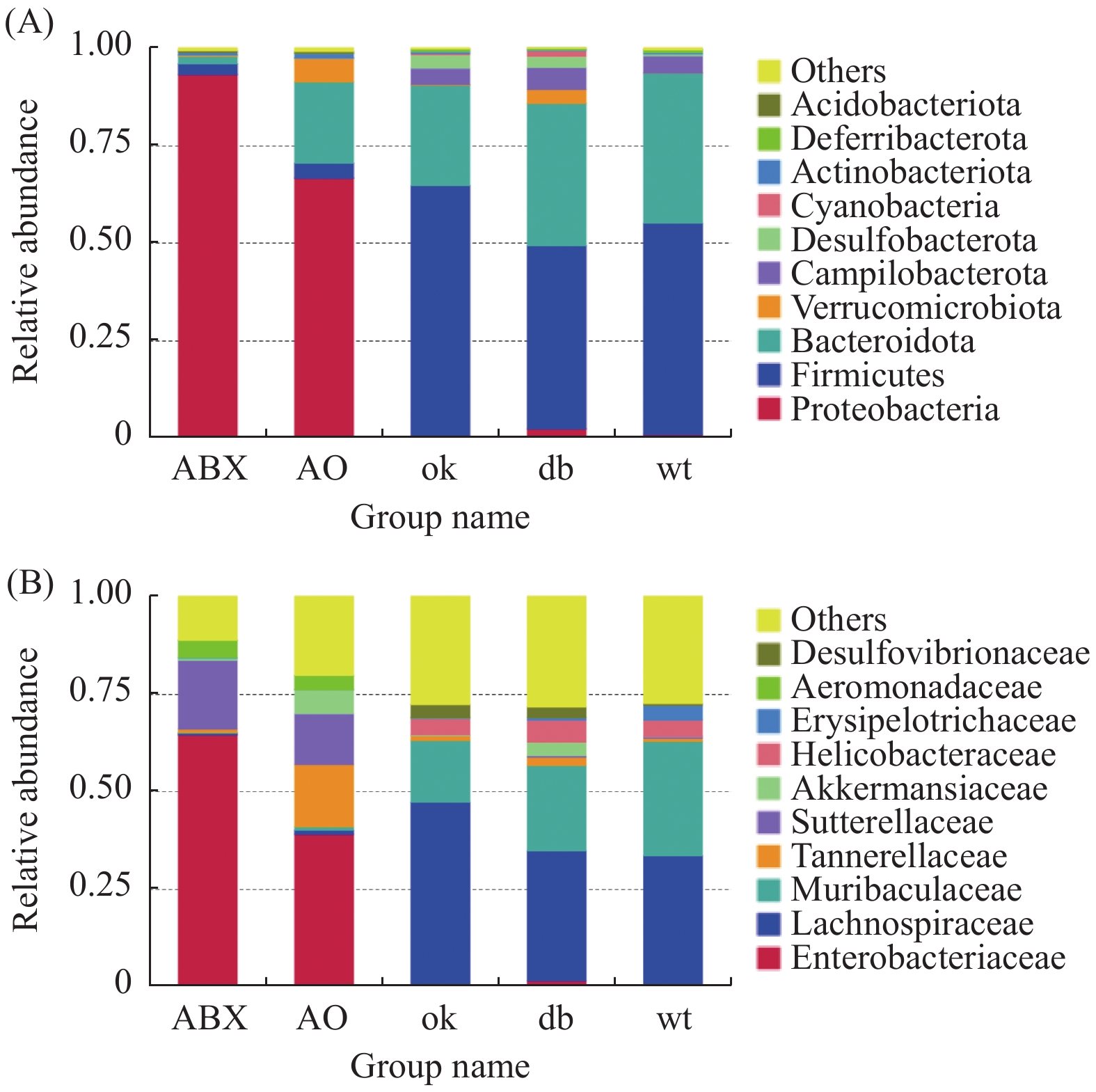
门水平上(图2A),db/db 和OK组厚壁菌门(Firmicutes)(0.471,0.644)和拟杆菌门(Bacteroidota)相对丰度最高(0.366,0.259)。ABX组变形菌门(Proteobacteria)的相对丰度高达0.934,AO组变形菌门相对丰度为0.667,其次为拟杆菌门(0.209)。进一步分析发现各组小鼠肠道菌门构成有明显差异,相对与db/db 组,OK组厚壁菌门(P < 0.05)、ABX( P < 0.01)和AO组( P < 0.01)变形菌门的丰度升高,OK组( P < 0.05)、AO组( P < 0.01)和ABX( P < 0.01)的拟杆菌门相对丰度降低。
科层面(图2B),db/db 小鼠和OK组毛螺菌科(Lachnospiraceae)(0.333、0.472)和Muribaculaceae科(0.219、0.159)群丰度最高。ABX组肠杆菌科(Enterobacteriaceae)和萨特菌科(Sutterellaceae)(0.650,0.178)丰度最高,AO组肠杆菌科、坦纳菌科(Tannerellaceae)和萨特菌科(0.390、0.162、0.130)丰度最高。5组小鼠肠道菌科构成差异明显,与db/db 组比较,OK组毛螺菌科(P < 0.05)、ABX和AO组肠杆菌科、萨特菌科及AO组坦纳菌科的丰度均升高( P < 0.01),而OK组Muribaculaceae丰度降低( P < 0.05)。
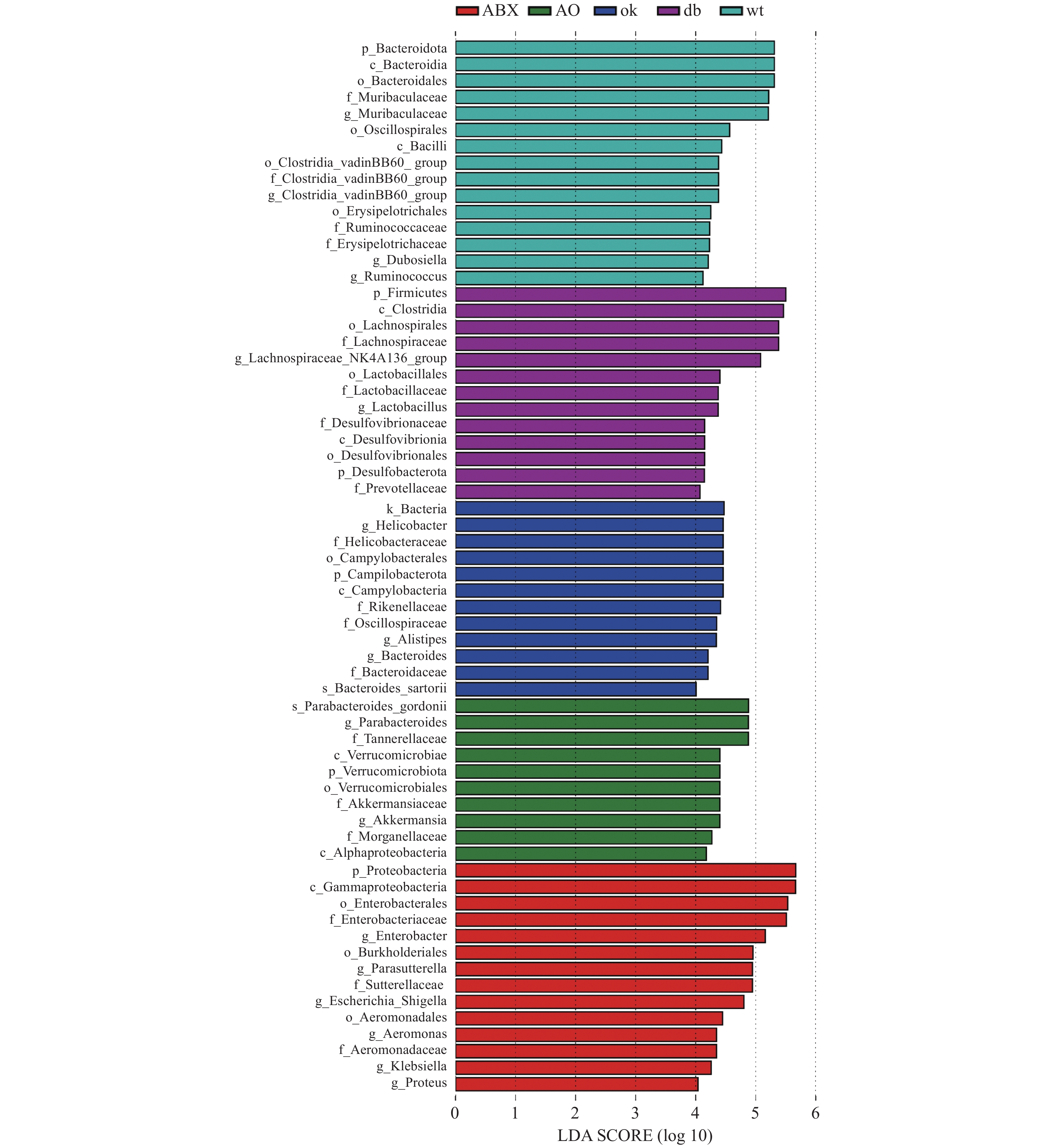
2.5 差异菌属筛
LEFse分析组间菌群差异(图3),野生型小鼠肠道菌群的优势物种为拟杆菌门Muribaculaceae属和厚壁菌门瓦丁梭菌BB60群(VadinBB60-group)、杜氏乳杆菌(Dubosiella)、瘤胃球菌属(Ruminococcus)。db/db小鼠的优势菌属为弯曲杆菌门螺旋杆菌属(Helicobacter)和拟杆菌门另枝菌属(Alistipes)和拟杆菌属(Bacteroides)。OK组的优势菌种为厚壁菌门毛螺菌科NK4A136组(Lachnospiraceae NK4A136 group)和乳杆菌属(Lactobacillus),脱硫杆菌门(Desulfobacterota)脱硫杆菌科(Desulfobacteraceae) )和拟杆菌门的普雷沃氏菌科(Prevotellaceae)等。ABX组的优势菌均为变形菌门,包括γ-变形菌纲肠杆菌科(Enterobacteriaceae)、气单胞菌科(Aeromonadaceae)和β-变形菌纲的萨特氏菌科(Sutterellaceae)等。AO组肠道菌群的优势物种包括古氏副拟杆菌(Parabacteroides gordonii),疣微菌门阿克曼菌属(Akkermansia)和变形菌门γ-变形菌纲肠杆菌目的摩根菌科(Morganellaceae)及α-变形菌纲(Alphaproteobacteria)等。 3. 讨论
糖尿病在中医学中属于“消渴病”范畴,由于先天不足、劳倦内伤、饮食失节、情志失调等导致。中医药治疗糖尿病已千余年,在“天人合一”的整体观念指导下,注重对患者全身功能的调节[5]。彝药恒古骨伤愈合剂由陈皮、红花、三七、杜仲、人参、黄芪、洋金花、钻地风、鳖甲等配方制成,功能主治为活血益气、补肝肾,符合中药治疗糖尿病的原则。课题前期发现恒古骨伤愈合剂改善db/db小鼠糖尿病性骨代谢异常和降低空腹血糖[3]。由于胰岛素在糖脂代谢中发挥重要作用,胰岛素抵抗指数(homeostasis model assessment-insulin resistance,HOMA-IR)是判断胰岛素抵抗的有效指标[6],本实验进一步发现恒古骨伤愈合剂改善db/db小鼠胰岛素抵抗。
肠道菌通过微生物-宿主共代谢系统影响药物代谢。通过抗生素处理杀灭微生物是研究肠道菌群变化的替代方法。抗生素可通过种类、剂量、给药途径及时间的不同,影响肠道菌群组成和结构[7]。抗生素诱导的肠道群的改变与代谢性疾病相关,如肥胖、糖尿病等[4]。选择广谱抑菌且肠道内吸收率相对缓慢的抗生素,其中包括万古霉素、氨苄西林、甲硝唑、新霉素的组合,已被证明对肠道菌群有杀伤作用[8]。本实验选择了以上混合抗生素,通过比对抗生素、恒古骨伤愈合剂单独和联合作用于db/db小鼠的效应。结果显示:经11周干预,抗生素、恒古骨伤愈合剂单独和联合作用,均降低2型糖尿病db/db小鼠空腹血糖和胰岛素抵抗,联合用药组效果更优。与本实验结果类似,出生至成年的长期抗生素暴露,通过调节肠道菌群在一定程度上抵抗高脂饮食负荷小鼠的糖代谢失调[9]。万古霉素降低NOD小鼠的糖尿病发病率[10]。广谱抗生素(给药12 d)降低db/db小鼠随机血糖,改变肠道微生物群组成[11]。本实验也表明广谱抗生素降低 db/db小鼠肠道菌群的丰富度与均匀性。广谱抗生素主要通过抑制拟杆菌门等常见的共生专性厌氧菌,并扩大正常情况下丰度较低变形杆菌门等兼性厌氧菌重塑肠道微生态[12-13]。相似地,本实验也发现ABX和AO组拟杆菌门相对丰度降低,变形菌门的丰度升高。而恒古骨伤愈合剂改善抗生素作用下的菌群的多样性。
LEFse分析显示野生型小鼠拟杆菌门Muribaculaceae属和厚壁菌门瓦丁梭菌BB60群(VadinBB60 -group)、杜氏乳杆菌(Dubosiella)、瘤胃球菌属(Ruminococcus)等益生菌富集。Muribaculaceae属在肠道的能量代谢、血糖血脂等发挥作用。Dubosiella的代谢产物为丁酸,可预防、改善肥胖。VadinBB60-group的代谢产物为短链脂肪酸,与丙酸盐比例呈正相关[14]。较高比例的丙酸盐会导致糖异生增加,减低肥胖[15]。提示野生型小鼠的标志菌在调节血糖中发挥作用。在中国人群中,拟杆菌属(Bacteroides)和另枝菌属(Alistipes)在2型糖尿病患者中比在糖代谢正常的人中更为丰富。另外,它们也与胆固醇水平升高相关[16]。糖尿病患者的幽门螺旋杆菌感染率明显更高[17]。以上临床结果与本实验中db/db小鼠螺旋杆菌属、Alistipes和Bacteroides富集相符。大豆不溶性膳食纤维通过增加乳杆菌属(Lactobacillus)和毛螺菌科NK4A136组(Lachnospiraceae _NK4A136_group)与肥胖相关的潜在有益菌的相对丰度,预防高脂肪饮食喂养小鼠肥胖[18]。黄芩和黄连处理增加产生短链脂肪酸的细菌Prevotellaceae UCG-001等的富集,改善2型糖尿病大鼠糖脂代谢[19]。提示本实验恒古骨伤愈合剂通过Lachnospiraceae NK4A136 group、Lactobacillus和Prevotellaceae等的富集,调节糖脂代谢。而广谱抗生素组中变形菌门γ-和β-变形菌纲的相关致病菌富集,提示抗生素虽然降低血糖,但可能导致重要的肠道微生物共生体从各群体中消失,使能够提供重要健康益处的细菌物种减少[20]。此外,本实验发现恒古骨伤愈合剂联合广谱抗生素干预组中,表现为有益菌Parabacteroides、Akkermansia和有害菌Morganellaceae、Alphaproteobacteria并存富集的状态。提示尽管ABS、OK和联合干预下db/db小鼠血糖及胰岛素抵抗均有所改善,但从肠道优势菌群富集的角度,各干预组涉及的具体调控机制可能有所不同。已有研究表明狄氏副拟杆菌胞外多糖粗提物具良好的免疫调节功能,由可减轻肥胖、缓解结肠炎[21]。阿克曼菌与肥胖、糖尿病、肝脏和心血管疾病等的发生发展相关,在调节机体代谢紊乱方面发挥着至关重要的作用[22-23]。恒古骨伤愈合剂改善抗生素导致肠道微生态紊乱,增加改善糖代谢的有益微生物的富集。推测恒古骨伤愈合剂改善血糖、胰岛素抵抗与这些有益微生物的富集有关,值得进一步探讨。
综上,广谱抗生素、恒古骨伤愈合剂单独和联合作用,均降低2型糖尿病db/db小鼠空腹血糖和胰岛素抵抗。且恒古骨伤愈合剂可改善抗生素对db/db肠道微生态损害。
-
表 1 GSE11969、TCGA的年龄、分期、性别分布情况
Table 1. GSE11969、TCGA age,stage,gender distribution
项目 合计(n = 580) GSE11969(n = 90) TCGA(n = 490) 年龄 平均值 64.55 (10.08) 61.02 (9.80) 65.21 (10.01) 中位值 65.00(59.00、72.00) 62.00(55.00、67.00) 66.00(59.00、72.00) 不详 10 (1.72%) 0 (0.00%) 10 (2.04%) 分期n(%) Ⅰ 315 (54.31) 52 (57.78) 263 (53.67) Ⅱ 128 (22.07) 13 (14.44) 115 (23.47) Ⅲ 104 (17.93) 25 (27.78) 79 (16.12) Ⅳ 25 (4.31) 0 (0.00) 25 (5.10) 不详 8 (1.38) 0 (0.00) 8 (1.63) 性别n(%) 女 305 (52.59) 43 (47.78) 262 (53.47) 男 275 (47.41) 47 (52.22) 228 (46.53) 表 2 GSE72094、TCGA的年龄、分期、性别分布情况
Table 2. GSE72094、TCGA age,stage,gender distribution overall
项目 合计(n = 888) GSE72094(n = 398) TCGA(n = 490) 年龄 平均值 67.09 (9.97) 69.36 (9.45) 65.21 (10.01) 中位值 68.00(60.25、74.00) 70.00(64.00、76.00) 66.00(59.00、72.00) 不详 10 (1.13%) 0 (0.00%) 10 (2.04%) 分期n(%) Ⅰ 517 (58.22) 254 (63.82) 263 (53.67) Ⅱ 182 (20.50) 67 (16.83) 115 (23.47) Ⅲ 136 (15.32) 57 (14.32) 79 (16.12) Ⅳ 40 (4.50) 15 (3.77) 25 (5.10) 不详 13 (1.46) 5 (1.26) 8 (1.63) 性别n(%) 女 484 (54.40) 222 (55.78) 262 (53.47) 男 404 (45.50) 176 (44.22) 228 (46.53) -
[1] Siegel,R L,K. D. Miller,A. Jemal. Cancer statistics 2020[J]. CA Cancer J Clin,2020,70(1):7-30. doi: 10.3322/caac.21590 [2] Sung H,Ferlay J,Siegel R L,et al. Global cancer stastistics 2020:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin,2021,71(3):209-249. doi: 10.3322/caac.21660 [3] Little A G,Gay E G,Gaspar L E,et al. National survey of non-small cell lung cancer in the United States:Epidemiology,pathology and patterns of care[J]. Lung Cancer,2007,57(3):253-260. doi: 10.1016/j.lungcan.2007.03.012 [4] Kleczko E K,Kwak J W,Schenk E L,et al. Targeting the complement pathway as a therapeutic strategy in lung cancer[J]. Front Immunol,2019,10(10):954. [5] Herbst R S,Soria J C,Kowanetz M,et al. Predictive correlates of response to the anti-PD-L1 antibody MPDL3280A in cancer patients[J]. Nature,2014,515(7528):563-567. doi: 10.1038/nature14011 [6] Shukuya T,Carbone D P. Predictive markers for the efficacy of anti-PD-1/PD-L1 antibodies in lung cancer[J]. J Thorac Oncol,2016,11(7):976-988. doi: 10.1016/j.jtho.2016.02.015 [7] Rizvi N A,Hellmann M D,Snyder A,et al. Cancer immunology. Mutational landscape determines sensitivity to PD-1 blockade in non-small cell lung cancer[J]. Science,2015,348(6230):124-128. doi: 10.1126/science.aaa1348 [8] Kanda A,Tamaki M,Nakamura E,et al. Characterization of recombinant human and rat pancreatic phospholipases A2 secreted from saccharomyces cerevisiae:Difference in proteolytic processing[J]. Biochim Biophys Acta,1992,1171(1):1-10. doi: 10.1016/0167-4781(92)90133-K [9] Hu B,Yang X B,Sang X T. Construction of a lipid metabolism-related and immune-associated prognostic signature for hepatocellular carcinoma[J]. Cancer Medicine,2020,9(20):7646-7662. doi: 10.1002/cam4.3353 [10] Matsuyama Y,Suzuki M,Arima C,et al. Proteasomal non-catalytic subunit PSMD2 as a potential therapeutic target in association with various clinicopathologic features in lung adenocarcinomas[J]. Mol Carcinog.,2011,50(4):301-309. doi: 10.1002/mc.20632 [11] Schabath M B,Welsh E A,Fulp W J,et al. Differential association of STK11 and TP53 with KRAS mutation-associated gene expression,proliferation and immune surveillance in lung adenocarcinoma[J]. Oncogene,2016,35(24):3209-3216. doi: 10.1038/onc.2015.375 [12] Chen B,Khodadoust M S,Liu CL,et al. Profiling tumor infiltrating immune cells with CIBERSORT[J]. Methods Mol Biol,2018,17(11):243-259. [13] Geeleher P,Cox N,Huang R S. pRRophetic:An R package for prediction of clinical chemotherapeutic response from tumor gene expression levels[J]. PloS One,2014,9(9):e107468. doi: 10.1371/journal.pone.0107468 [14] Selvanesan B C,Chandra D,Quispe-Tintaya W,et al. Listeria delivers tetanus toxoid protein to pancreatic tumors and induces cancer cell death in mice[J]. Sci Transl Med,2022,14(637):1600. doi: 10.1126/scitranslmed.abc1600 -






 下载:
下载:
 下载:
下载: